网页版Rstudio安装与使用
Rstudio server
RStudio Server enables you to run the RStudio IDE you know and love on a Linux server, accessed from your web browser, bringing the power and productivity of the RStudio IDE to a centralized server-based environment.
RStudio Server是网页版的RStudio,部署在服务器上后,能够在从Web浏览器访问的Linux服务器上运行RStudio,可随时随地完成R语言的工作,并且能很方便的完成R项目的部署调试。使将RStudio IDE的强大功能和工作效率带到基于服务器的集中式环境中。
之前读研的时候,师弟貌似就很想往课题组服务器中装一个Rstudio Server,我当时一直没太有实感,不过最近突发奇想想试试看。
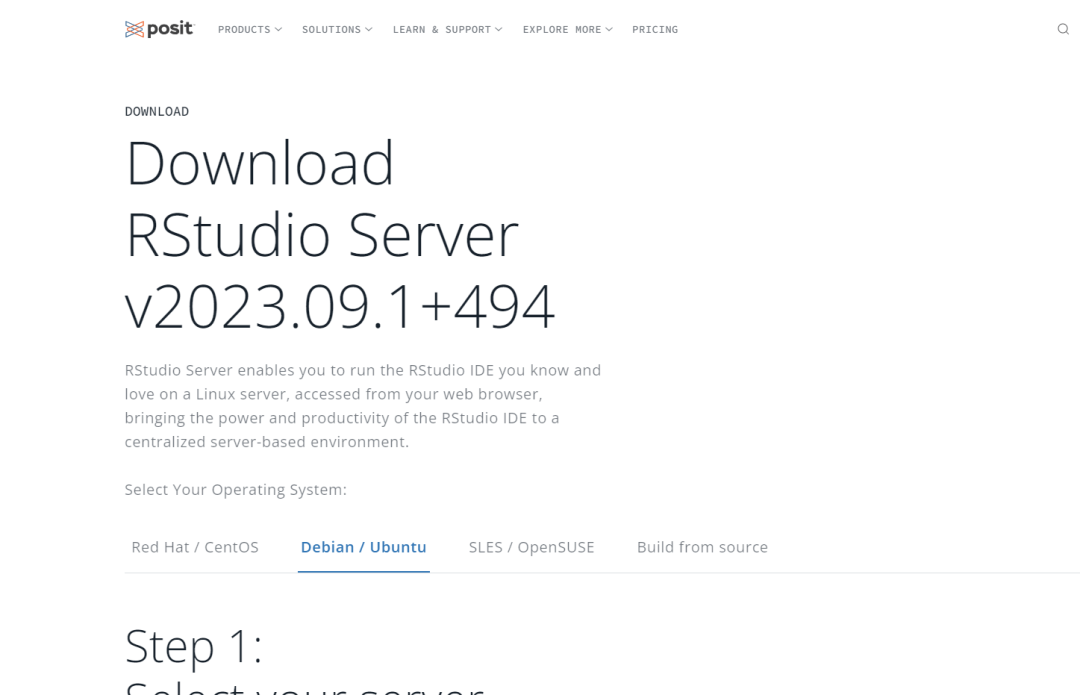
官网安装教程
Bing检索Rstudio server,选择国际版,就可以看到下载界面。
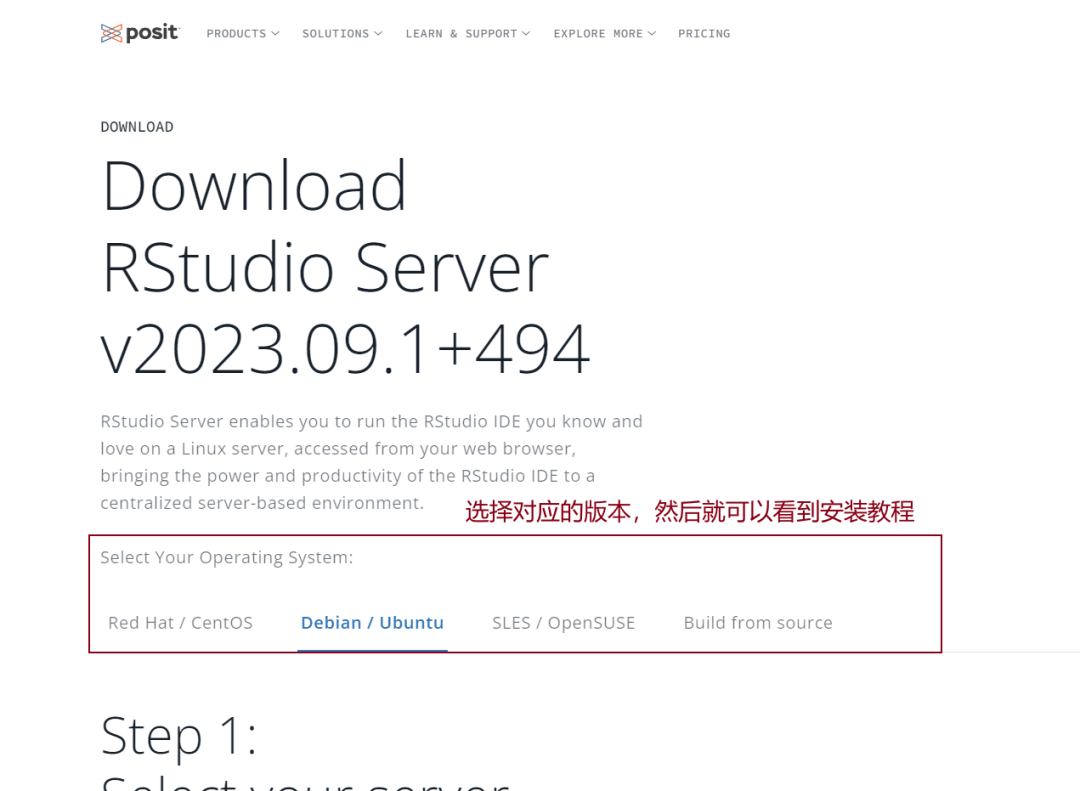
点击进入下载页面之后选择对应的安装版本,再根据教程去安装就可以了。
负责搭建的服务器的同事说不难,就是输入对应的命令,然后开相应的端口即可,但是需要管理员权限。(我没有实际操作过,我没有发言权。)
注:生信技能树的服务器已配置好Rstudio Server无需再自行配置哦!
安装步骤:
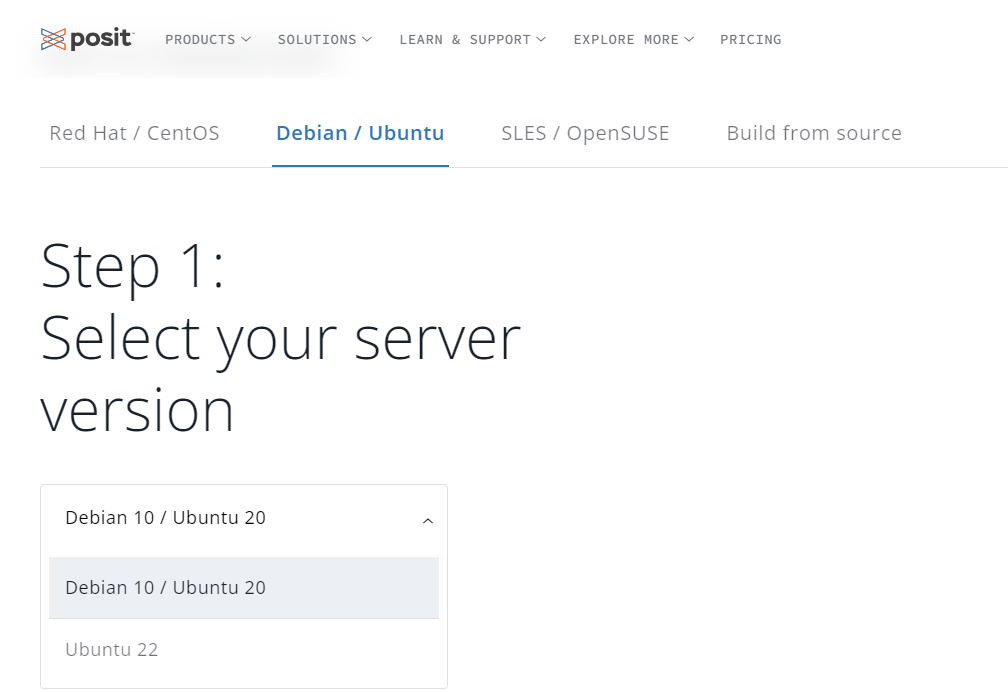
- 选择对应的版本:Debian 10 / Ubuntu 20
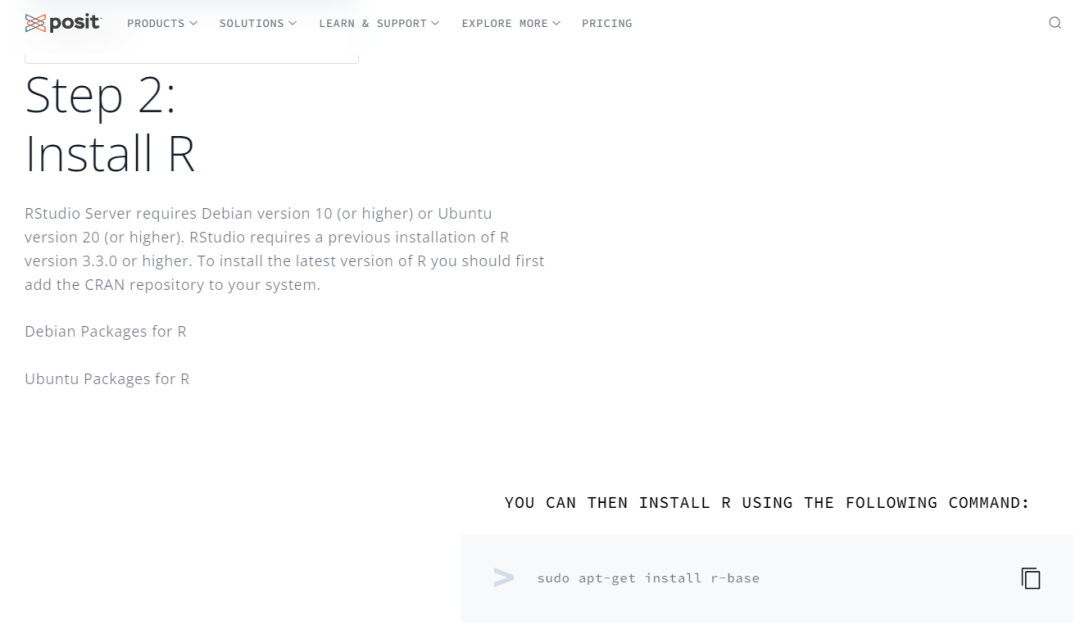
- 下载R-base
sudo apt-get install r-base
- 安装Rstudio Server
sudo apt-get install gdebi-core
wget https://download2.rstudio.org/server/focal/amd64/rstudio-server-2023.09.1-494-amd64.deb
sudo gdebi rstudio-server-2023.09.1-494-amd64.deb
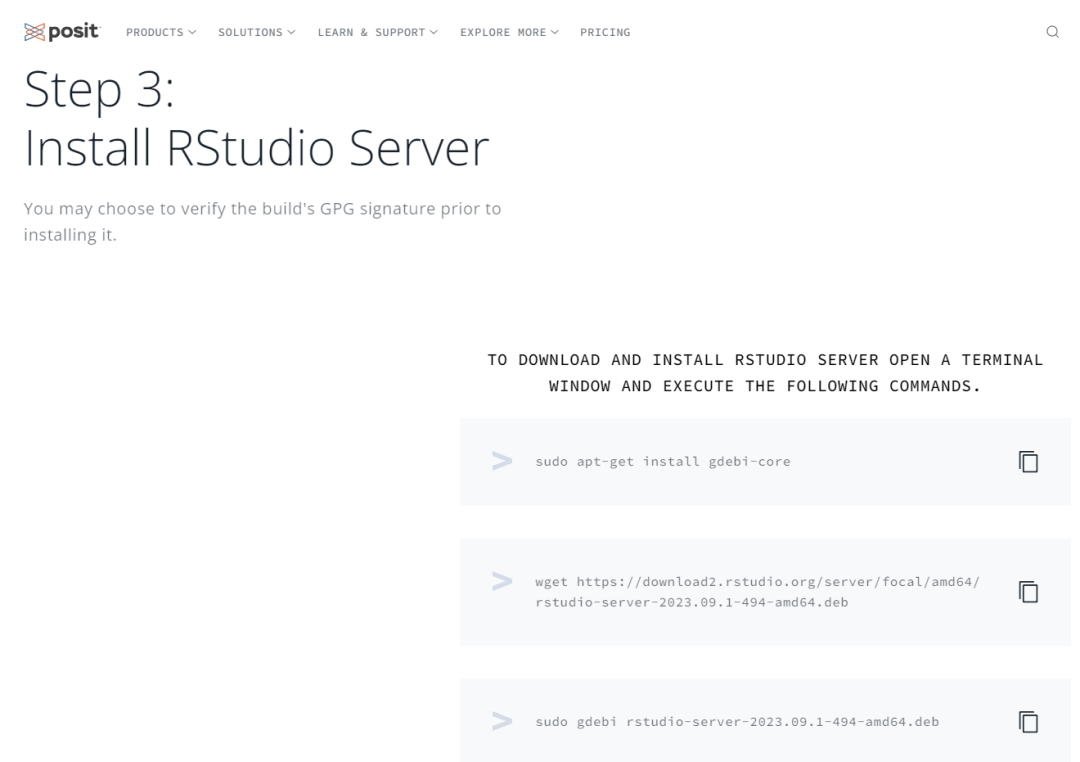
安装成功就可以,但是官网一般是默认不能指定版本,所以如果需要对应的R语言版本的话,使用wget下载下来然后安装编译即可。
以下命令取自配置服务器的劳模小张
- 开启对应的端口
sudo ufw allow 8787
- 启动Rstudio Server
## rstudio-server 开启
sudo rstudio-server start
sudo rstudio-server status
sudo rstudio-server stop
sudo rstudio-server restart
Rstudio Server使用
使用初衷:根本原因是更新了本地的R和Rstudio版本,详情请见R和Rstudio版本更新,然后我的R包没有同步更新,所以就无了。
所以就想偷懒,先试试看网页版的Rstudio,反正咱们自己的服务器里面配套安装了几百个常用的R包。
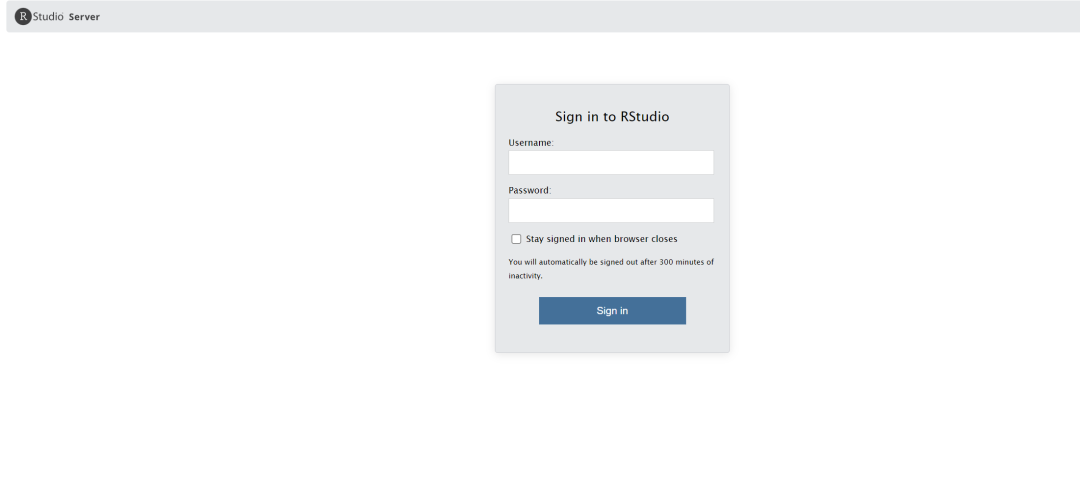
1. 登录Rstudio Server
点击对应的网页链接,然后输入账号和密码信息即可登录。
2.Rstudio界面
和本地的Rstudio是一样一样的,只不过它是基于服务器的计算资源和存储空间而已。
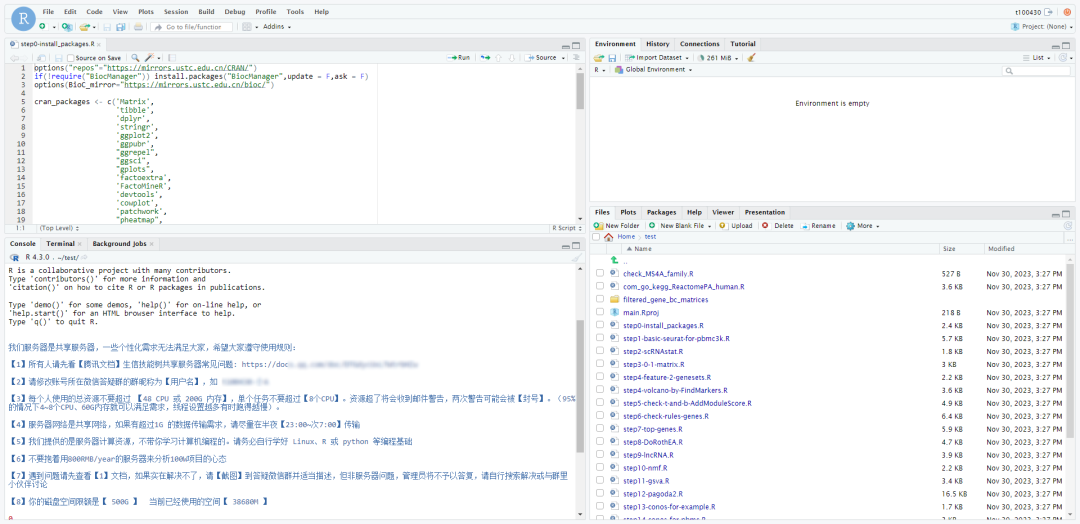
那上面两个区域——文本展示以及环境和数据展示界面就不给大家介绍啦,和本地的是一模一样的。主要介绍一下下面两个界面。
Consle命令运行界面:
会直接显示Terminal,方便我们链接到服务器上面使用linux命令去查看文件和处理一些文件格式之类的问题。
网页版Rstudio
当然Windows里面也可以连接本地Terminal,Shift+Alt+R新建 Code
本地版Rstudio
Files文件传输:
在Files区域,除了基本的查看文件、新建文件夹以及空白文件之外,多了一个Upload选项,可以从本地上传文件到服务器上,方便我们分析
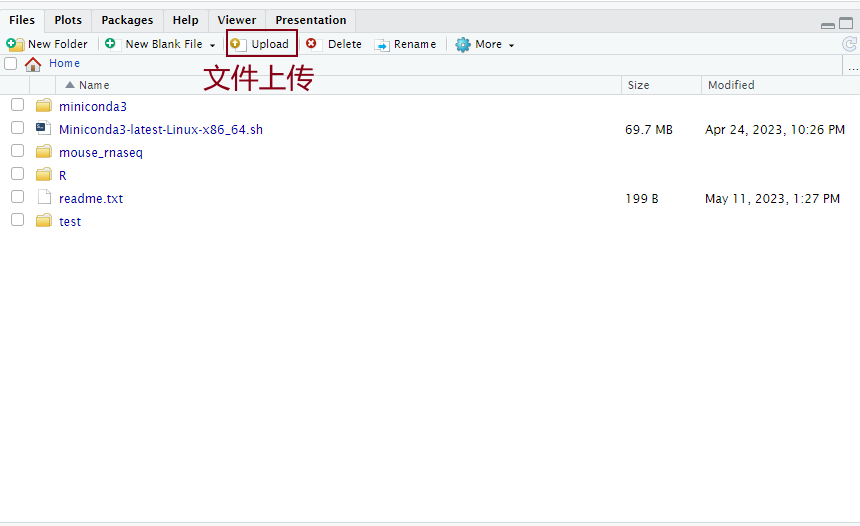
上传步骤:点击Upload——选择需要上传到的文件夹——选择相应的文件——点击OK上传。
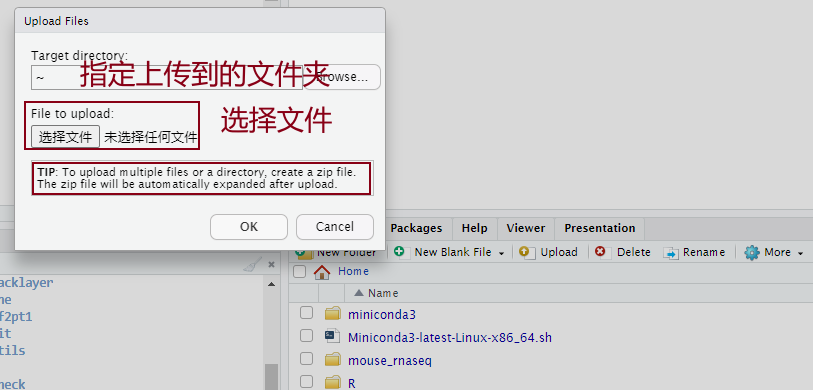
小Tips:如果需要上传多个文件或一个目录,需要创建一个zip文件。上传完成后,zip文件会自动展开。
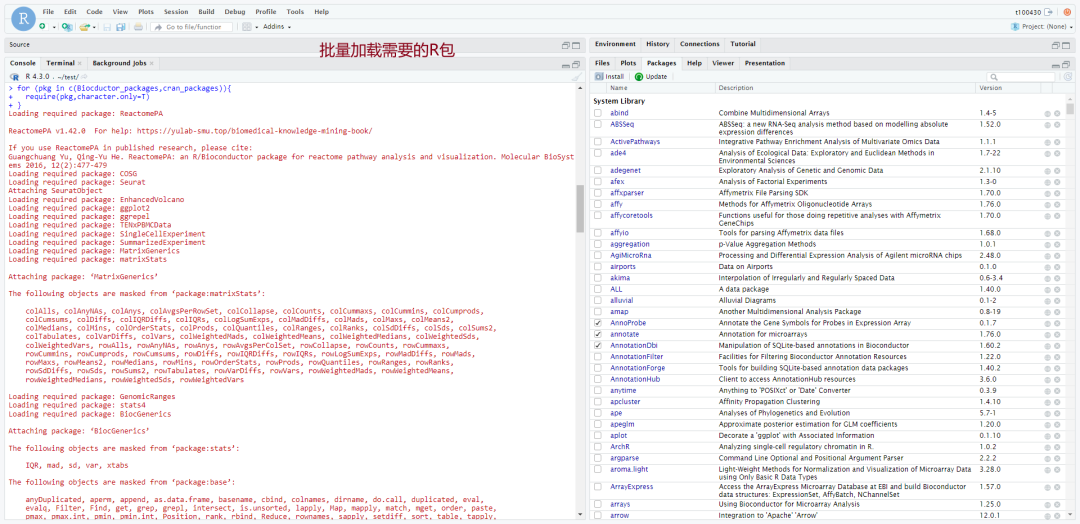
批量加载需要的R包
因为咱们的服务器是已经安装了很多的R包,所以我们可以指定一下R包的临时路径,然后方便加载需要的R包
#将服务器中的R包路径设置为临时的路径
.libPaths(c(.libPaths(),"/home/data/refdir/Rlib"))
#确定目前的R包路径
.libPaths()
[1] "/home/data/t100430/R/x86_64-pc-linux-gnu-library/4.3" "/usr/local/lib/R/library"
[3] "/home/data/refdir/Rlib"
批量加载R包:因为很多常用的包已经安装好了,所以我们直接加载一下服务器的R包即可
#单细胞分析需要的基础R包
cran_packages <- c('Matrix',
'tibble',
'dplyr',
'stringr',
'ggplot2',
'ggpubr',
"ggrepel",
"ggsci",
"gplots",
'factoextra',
'FactoMineR',
'devtools',
'cowplot',
'patchwork',
"pheatmap",
'basetheme',
'paletteer',
'AnnoProbe',
'ggthemes',
'VennDiagram',
'tinyarray')
Biocductor_packages <- c('ReactomePA',
'COSG',
"Seurat",
'EnhancedVolcano',
"Seurat",
"TENxPBMCData",
"GSEABase",
"GSVA",
"clusterProfiler",
"org.Hs.eg.db",
"UpSetR",
"clustree",
"conos",
"cowplot",
"dorothea",
"entropy",
"future",
"msigdbr",
"pagoda2",
"scRNAseq",
"scRNAstat",
"tidyverse",
"viper",
"progeny",
"preprocessCore",
"enrichplot")
#批量读取
for (pkg in c(Biocductor_packages,cran_packages)){
require(pkg,character.only=T)
}
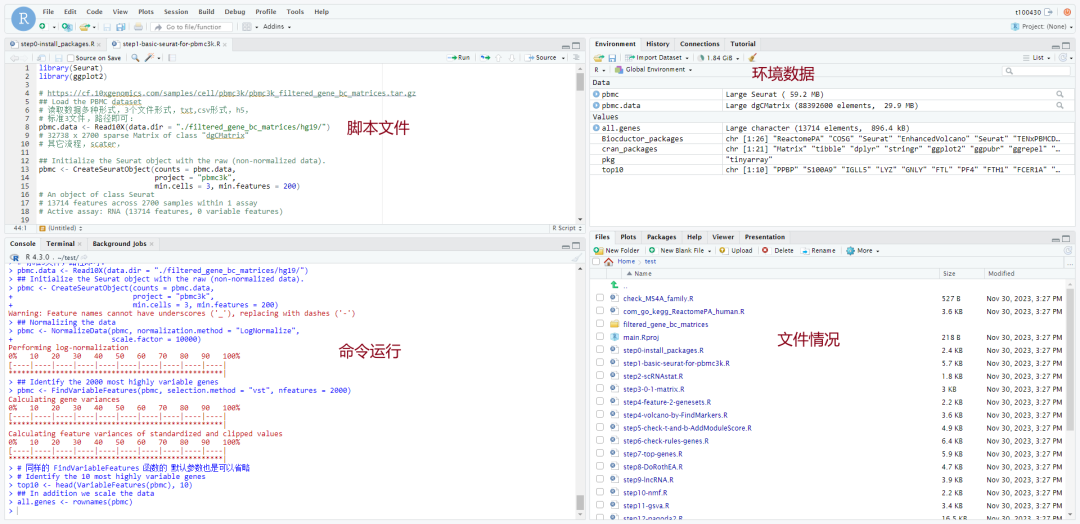
读取单细胞数据进行分析
基本的使用和本地版是没有什么去别的,就是运行对应的命令,读取需要的数据文件,然后进行分析即可
hhh感觉配置好的Rstudio真的很不错,那在我把本地的包搞定之前就先用网页版的Rstudio来学习单细胞啦!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-12-01,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

