单细胞分析工具 | ScSmOP:用于单细胞多组学数据分析的通用计算流程
单细胞分析工具 | ScSmOP:用于单细胞多组学数据分析的通用计算流程

单细胞多组学技术为阐明单个细胞的基因组、表观基因组和转录组异质性的特征提供了见解。然而,它们给数据处理带来了新的计算挑战。2023年10月,《Briefings in Bioinformatics》发表了一种用于条形码索引的单细胞-单分子多组学数据分析的通用流程——ScSmOP,用于多模态数据分析。

ScSmOP是什么?

ScSmOP是一个用于条形码索引单细胞单分子多组学数据分析的通用流程。ScSmOP的本质是利用C语言,根据基于连接的条形码数据和基于合成的条形码数据,建立基于间隔种子哈希表的条形码识别算法,然后进行数据映射和去卷积。
ScSmOP安装方便,一键完成,运行速度快,广泛适用于处理各种单细胞、单分子、多组学技术和不同物种产生的数据。此外,ScSmOP为用户提供了一个接口,以便随后处理他们定制的条形码并执行自动数据处理。
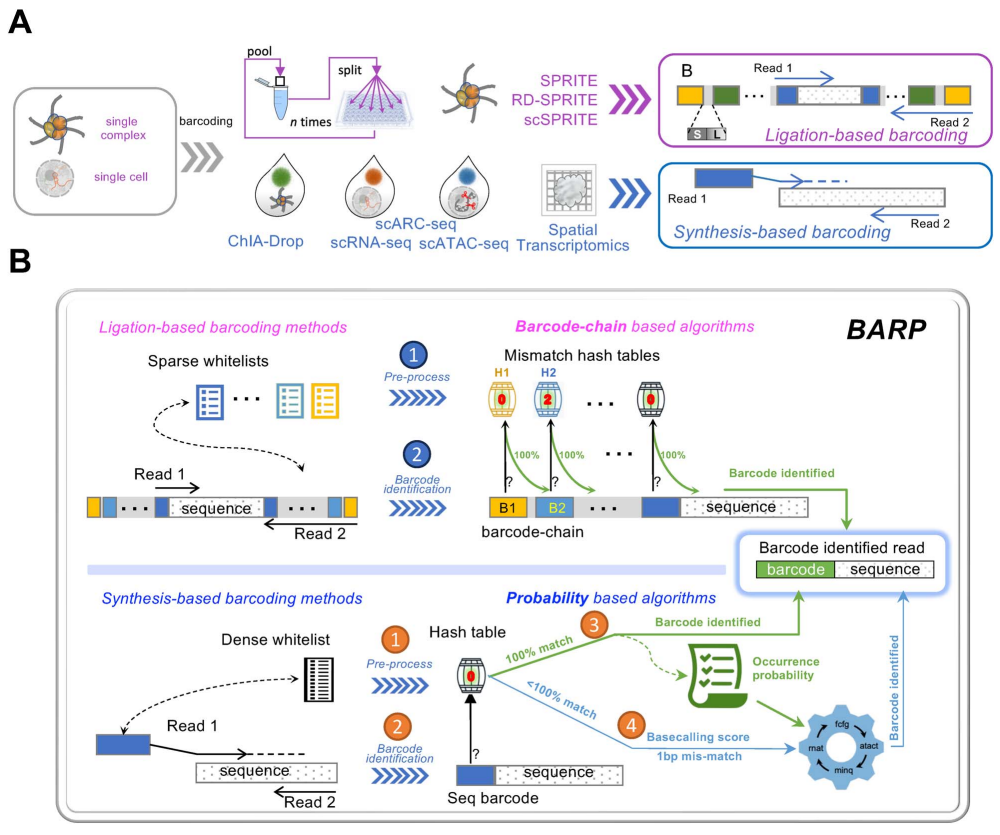
ScSmOP的条码策略和条码识别算法
开发团队开发了基于间隔种子哈希表的条形码识别算法,这些算法被封装在C语言编码的程序barcode Processing(BARP)中。为单细胞或单分子染色质复合物条形码设计的核苷酸通常长度固定,在测序文库结构中的位置也固定;因此,开发团队应用间隔种子算法将白名单条形码转换为n个不匹配的哈希表,并将其存储在计算机内存中,以便一次性进行条形码匹配。得益于这种策略和C语言的特点,BARP可以加快条形码识别的进程。通过使用BARP,开发团队建立了一个用于多模式数据分析流程——ScSmOP。
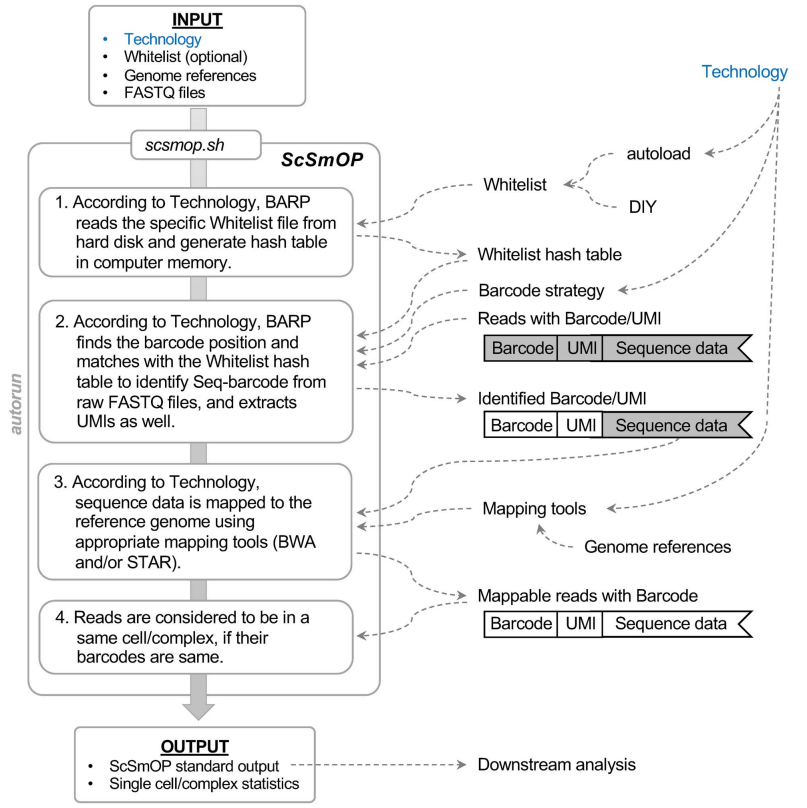
ScSmOP执行接口
ScSmOP的性能测试

开发团队在对单细胞组学数据(scRNA-seq、scATAC-seq、scARC-seq)、单分子染色质相互作用数据(ChIA-Drop、SPRITE、RD-SPRITE)、单细胞单分子染色质相互作用数据(scSPRITE)以及来自各种细胞类型和物种的空间转录组学数据的综合分析中,证明了:

ScSmOP与已发表流程之间数据处理的高重现性。此外,ScSmOP表现出更快的性能,是用于单细胞单分子多组学数据分析的多功能、高效、易用和稳健的管道。
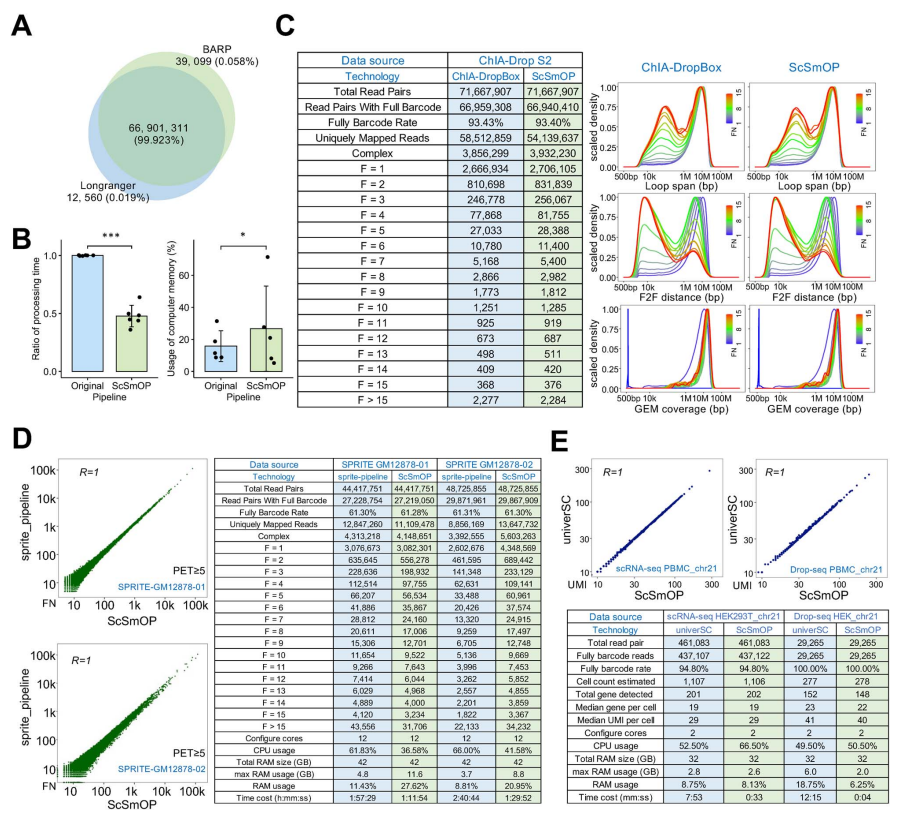
ScSmOP的性能
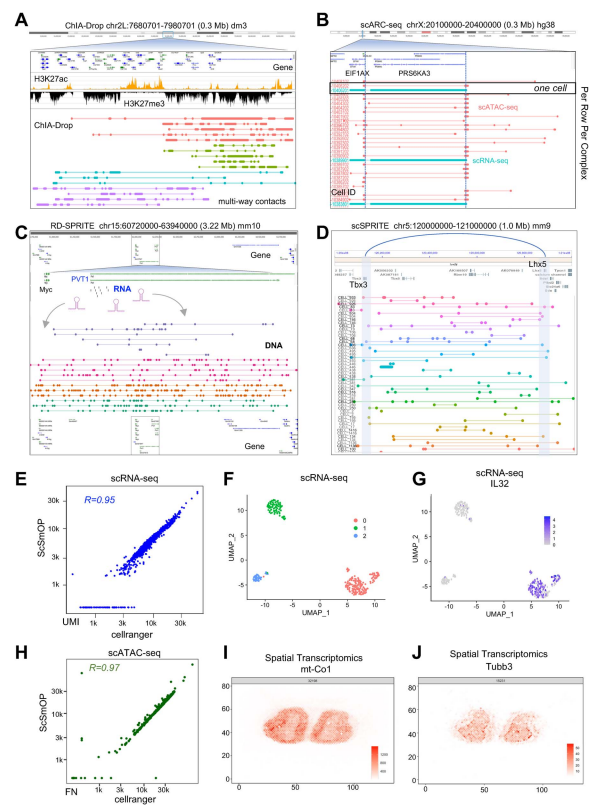
ScSmOP的可视化和统计结果
综上,ScSmOP是一种多功能、高效、易于使用且强大的流程,用于单细胞单分子多组学数据分析。
ScSmOP代码可在如下链接获取:
👉 https://github.com/ZhengmzLab/ScSmOP.
//
建议对技术细节感兴趣的小伙伴请参考文献原文~
对于文献整理过程中有翻译不当或错误也欢迎大家在评论区留言指出,互相交流学习!

参考文献
Kai Jing, Yewen Xu, Yang Yang, Pengfei Yin, Duo Ning, Guangyu Huang, Yuqing Deng, Gengzhan Chen, Guoliang Li, Simon Zhongyuan Tian, Meizhen Zheng, ScSmOP: a universal computational pipeline for single-cell single-molecule multiomics data analysis, Briefings in Bioinformatics, Volume 24, Issue 6, November 2023,bbad343.

