全网首发 | Visium HD空转数据开箱测试
2024年1月份,10X公司预告了一个新技术10X Visium HD,分辨率可达2 μm,可实现单细胞分辨率的全转录组空间分析,并且可实现连续的组织覆盖率。千呼万唤始出来,10X官网在前几天公开了2个10X Visium HD测试数据,分别是
- Mouse Small Intestine,https://www.10xgenomics.com/datasets/visium-hd-cytassist-gene-expression-libraries-of-mouse-intestine
- Human Colorectal Cancer (CRC),https://www.10xgenomics.com/datasets/visium-hd-cytassist-gene-expression-libraries-of-human-crc
我也在第一时间进行了开箱测试,然而万事开头难,在数据读入阶段就遇到一堆Bug,详见我的踩坑笔记:
下文记录了CRC数据开箱测试的标准分析结果。
Step1. 加载数据
这里读取我上期保存好的数据【踩坑实录 | 使用R语言读入Visium HD空转数据】:
library(Seurat)
library(ggplot2)
library(patchwork)
library(dplyr)
library(qs)
### 1.读入数据
CRC_data = qread("./Outdata/CRC_Visium_HD_8bin_image_solo.qs")
CRC_data
# An object of class Seurat
# 18085 features across 545913 samples within 1 assay
# Active assay: Spatial (18085 features, 0 variable features)
# 1 images present: CRC
p1 = SpatialDimPlot(CRC_data, alpha = 0)
ggsave(filename = "./Outplot/SpatialDimPlot.png", plot = p1)
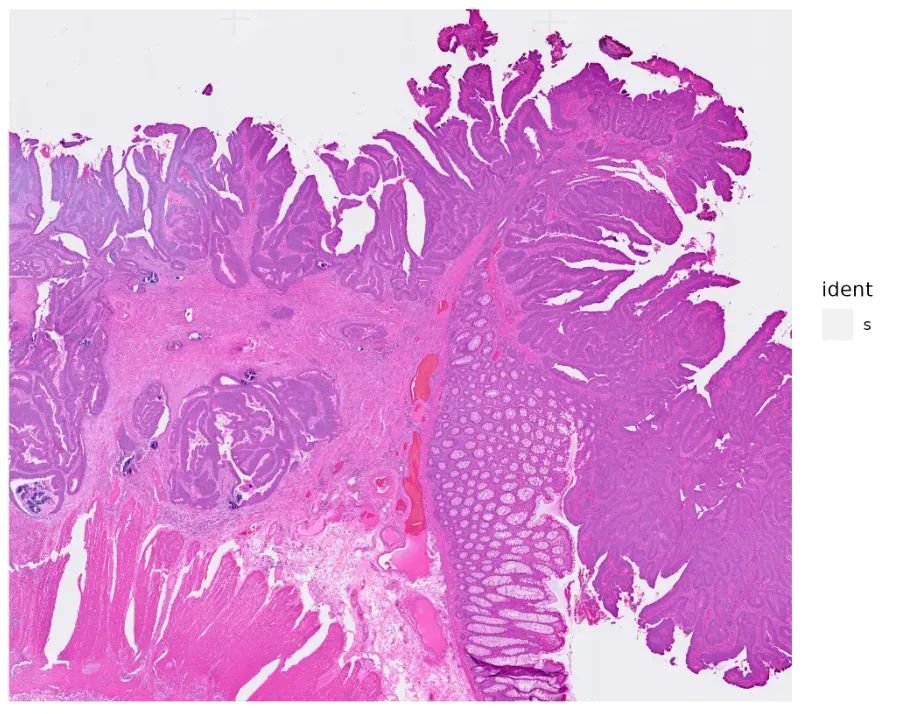
image-20240329220908253
Step2. 数据质控
### 2.质控
sc.QC.Spatial = function(merged_seurat){
### 1.1 Number of genes detected per UMI
# Add number of genes per UMI for each cell to metadata
merged_seurat$log10GenesPerUMI <- log10(merged_seurat$nFeature_Spatial) / log10(merged_seurat$nCount_Spatial)
### 1.2 QC index calculation
## 1.2.1 Mitochondrial ratio
mito_genes=rownames(merged_seurat)[grep("^MT-", rownames(merged_seurat))]
print("mito_genes:")
print(mito_genes)
merged_seurat <- PercentageFeatureSet(object = merged_seurat, pattern = "^MT-",col.name = "percent.mt")
## 1.2.2 Ribosomal ratio
ribo_genes=rownames(merged_seurat)[grep("^RP[SL]", rownames(merged_seurat),ignore.case = T)]
print("ribo_genes:")
print(ribo_genes)
merged_seurat <- PercentageFeatureSet(merged_seurat,"^RP[SL]",col.name = "percent.ribo")
print(summary(merged_seurat@meta.data$percent.ribo))
## 1.2.3 Erythrocyte ratio
hb_genes <- rownames(merged_seurat)[grep("^HB[^(P)]", rownames(merged_seurat),ignore.case = T)]
print("hb_genes")
print(hb_genes)
merged_seurat=PercentageFeatureSet(merged_seurat, "^HB[^(P)]", col.name = "percent.hb")
print(summary(merged_seurat@meta.data$percent.hb))
return(merged_seurat)
}
CRC_data = NormalizeData(CRC_data)
CRC_data = sc.QC.Spatial(CRC_data)
p.VlnPlot = VlnPlot(CRC_data,pt.size = 0,
features = c("nFeature_Spatial", "nCount_Spatial", "percent.mt"),
ncol = 3)
ggsave(filename = "./Outplot/Step2.QC.VlnPlot.png",width = 14,height = 5)
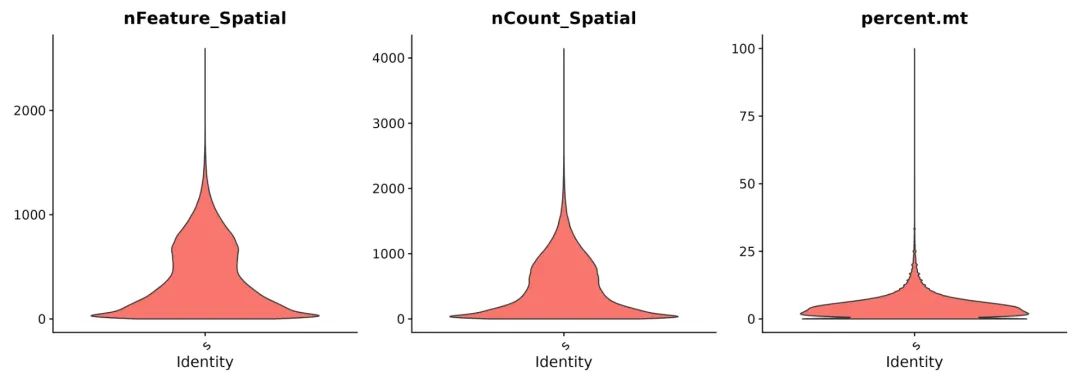
image-20240329211412281
可以看到Visium HD空转数据的测序深度似乎比10X单细胞低一些。另外,在过滤低质量细胞过程中,我发现按照10X单细胞的过滤标准对于Visium HD空转数据而言不太合适:
## 过滤
qc.test <- subset(CRC_data, subset = nFeature_Spatial >= 250 &
nCount_Spatial>= 500 & percent.mt < 20)
qc.test #过滤后 18085 features across 224858 samples
CRC_data #过滤前 18085 features across 545913 samples
然后我查阅了华大的过滤标准,华大对percent.mt的设置为5,在这里我设置为15,其他指标和华大的过滤标准一致:
## 华大QC流程:
CRC.qc <- subset(CRC_data, subset = nFeature_Spatial >= 20 &
nCount_Spatial>= 3 & percent.mt < 15)
CRC.qc #过滤后 18085 features across 497201 samples
这里我没有过滤低表达的基因。
Step3. 标准流程
这里我采用了Seurat单细胞的标准流程进行分析:
### 三步走:标准化、特征选择和归一化分析
CRC.qc <- CRC.qc %>% NormalizeData(verbose = F) %>%
FindVariableFeatures(selection.method = "vst", nfeatures = 2000, verbose = F) %>%
ScaleData(verbose = F) %>%
RunPCA(npcs = 50, verbose = F)
CRC.qc
这里没有使用SCTransform流程,因为近50w的细胞量,如果用SCTransform内存会爆炸的。有足够计算机资源的朋友可以试试:
#CRC.qc <- SCTransform(CRC.qc, assay = "Spatial", return.only.var.genes = FALSE, verbose = FALSE)
由于是单个样本,因此不需要进行整合去批次,直接进行降维聚类即可:
CRC.qc <- RunUMAP(CRC.qc, reduction = "pca", dims = 1:30) %>%
FindNeighbors(reduction = "pca", dims = 1:30) %>%
FindClusters(verbose = FALSE)
p2 <- DimPlot(CRC.qc, reduction = "umap", label = TRUE, raster= T)
ggsave(filename = "./Outplot/Step3.UMAP.png",
plot = p2, width = 6, height = 6)
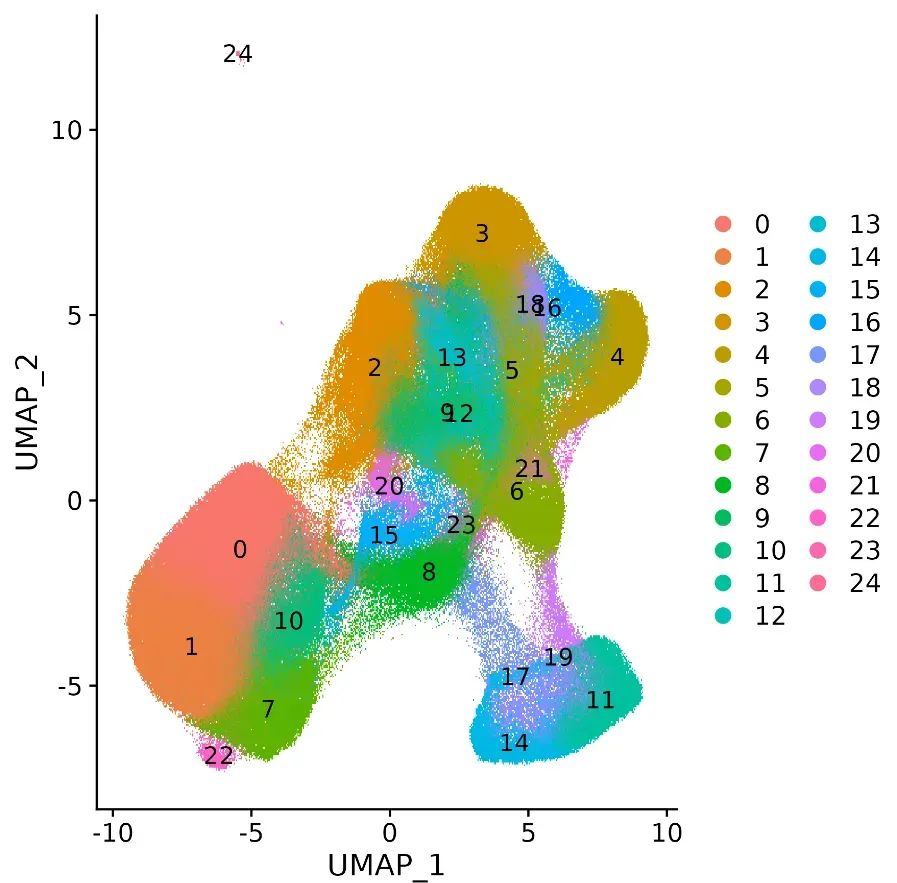
image-20240329221425781
p3 <- SpatialDimPlot(CRC.qc, label = F,
pt.size.factor = 0.15,
stroke = 0)
ggsave(filename = "./Outplot/Step3.SpatialDimPlot.png",
plot = p3, width = 6, height = 6)
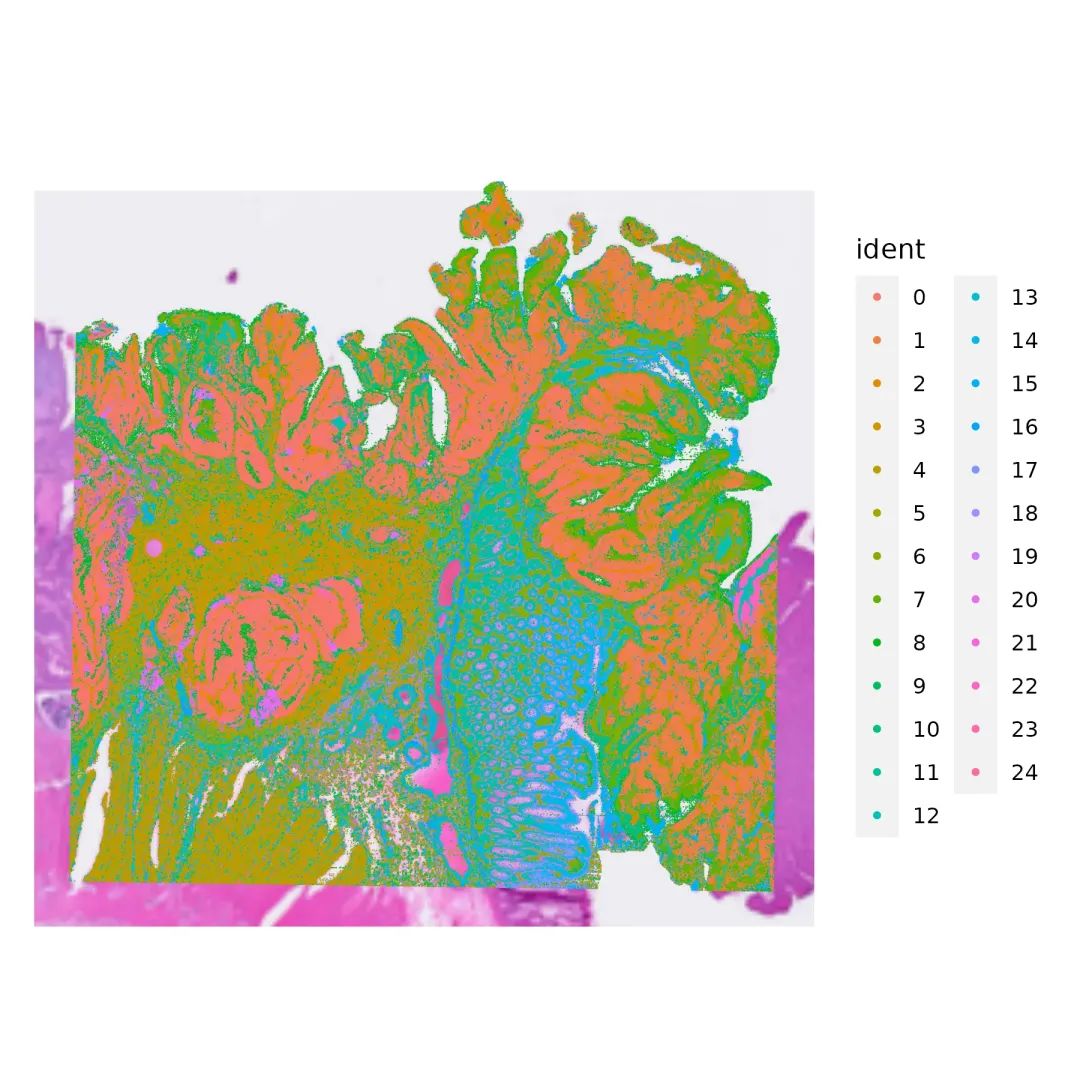
Step3.SpatialDimPlot
SpatialDimPlot(CRC.qc,
cells.highlight = CellsByIdentities(object = CRC.qc,
idents = c(12, 14, 18)),
pt.size.factor = 0.15,
facet.highlight = TRUE, ncol = 3)

image-20240329225203052
总体来说,聚类效果还可以,但是使用默认分辨率识别到的亚群有点多。用户可以自行更改分辨率。
对经典的marker进行可视化检查(由于我不是做CRC的,因此没有特别收集CRC上皮细胞类型的标志物):
DotPlot(object = CRC_data,
features = c("RGS1","PTPRC",'TYROBP', #Leukocytes (Leu)
'CD3D','CD3E',"CD3G", #T cells
"NKG7","GZMB","KLRD1", #NK cells
"IGHM","CD22","CD79A", #B cells
"JCHAIN","MZB1","PRDM1", #Plasma cells
'CD14',"HLA-DQB1","C1QA", #monocyte/Macrophages
"CLEC10A","CLEC4C", #DC cells
"KIT","GATA2",'TPSB2', #Mast cells
'EPCAM',#Epithelial
"REG1A","REG1B","CEACAM6","TGFBI", #Tumor cells
"MUC2","FCGBP", #goblet cells
"ACKR1",'CLDN5','CDH5', #Endo
"DCN","VIM","LUM","ACTA2","MYH11"#Fibroblast
),
group.by = "seurat_clusters")+coord_flip()
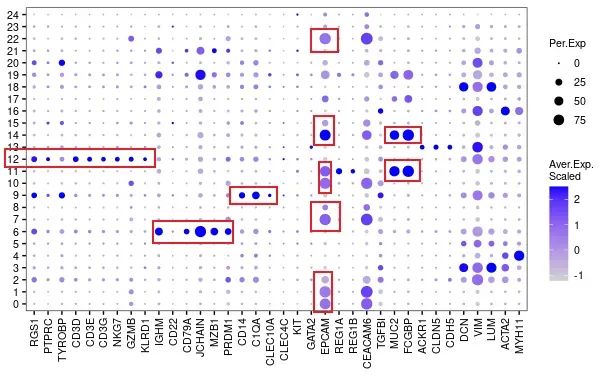
image-20240329223050600
这效果比10X Visium好太多了!
这里由于我对CRC不太熟悉,没有继续做亚群注释。
我挑选了几个经典的marker继续进行可视化:
- 例如成纤维细胞(DCN)
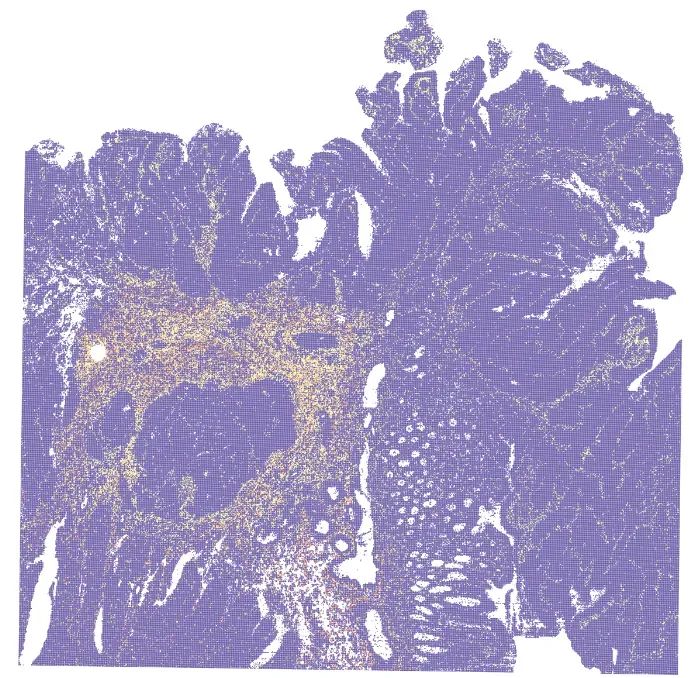
image-20240329224131223
- 上皮细胞(EPCAM)
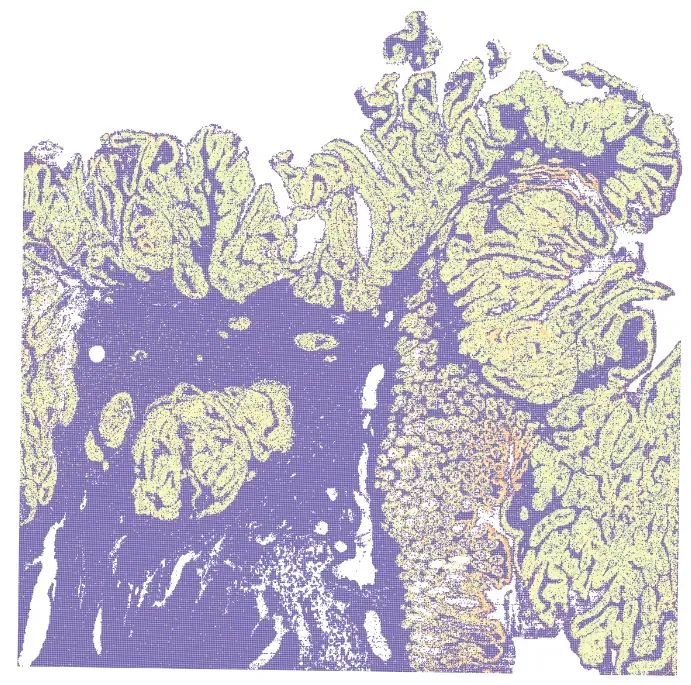
image-20240329223707998
- 杯状细胞(FCGBP)
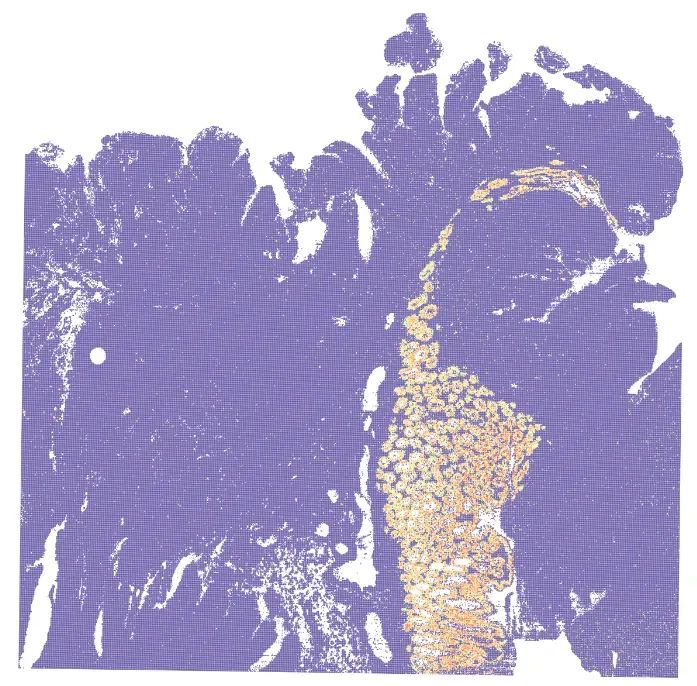
image-20240329223438050
- T细胞(CD3E)
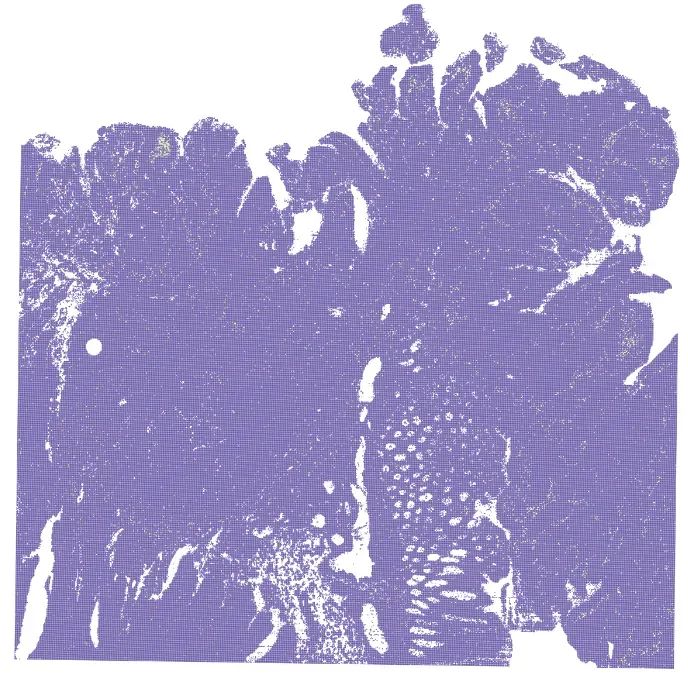
image-20240329223909593
- 单核细胞(CD14)
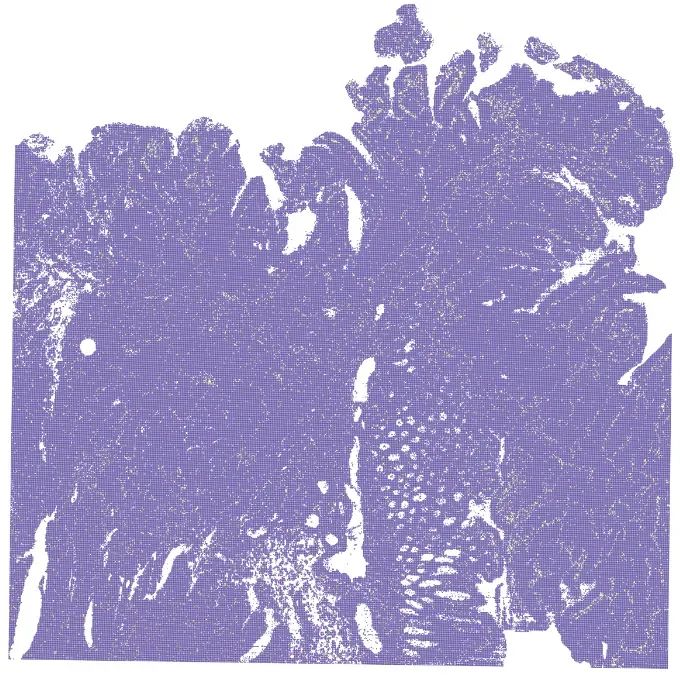
image-20240329224046924
总结
实际上,从标准分析流程开始,后续的分析和10X单细胞分析非常相似,但是会涉及一些空间层面的分析,例如细胞通讯及空间生态位分析,我们后续再进行讨论~
总体来说,Visium HD空转数据的精细度相较于10X Visium大大提高。但是仍存在一些问题和挑战,例如:
- 一张8微米的切片具有50w个单细胞,而上述单样本的分析内存最大峰值近60GB(并未涉及高级分析)。后续开展进阶/高级分析,包括差异表达分析、转录因子分析以及细胞通讯分析等,必然需要消耗极大的资源。
- 未来在进行多样本的Visium HD数据联合分析时,分析难度必然会陡增。
- 与10X Visium 相比,Visium HD空转数据可以做哪些不同角度且更深入地挖掘和分析?这点可能需要我们在阅读大量文献的基础上,结合专业背景进行设计和思考。
- 由于我暂时没有接触过华大等单细胞精度的空转数据,我比较好奇与其他大品牌的空转数据相比,Visium HD空转数据的表现如何?
当然,我只是一个空转数据的初学者,而且对CRC这个癌种也不熟悉,所以只是管中窥豹,理解不多。还请各位各抒己见,如有错误多多批评~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-03-30,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

