比较两种不同算法的表达量矩阵的差异分析结果
我们分享了一个案例,就是GSE30122这个数据集的作者给出来的表达量矩阵是被zscore的,所以我们可以下载它的cel文件自己制作表达量矩阵,详见:
然后这两个表达量矩阵其实都是可以做标准差异分析流程的,各自独立分析都有差异结果,这个时候我们就可以比较两种不同算法的表达量矩阵的差异分析结果。
第一次差异分析结果(基于zscore表达量矩阵)
虽然GSE30122这个数据集的作者给出来的表达量矩阵是被zscore的,但是也是可以走limma这样的差异分析流程的,就有上下调基因,可以绘制火山图和热图,如下所示:
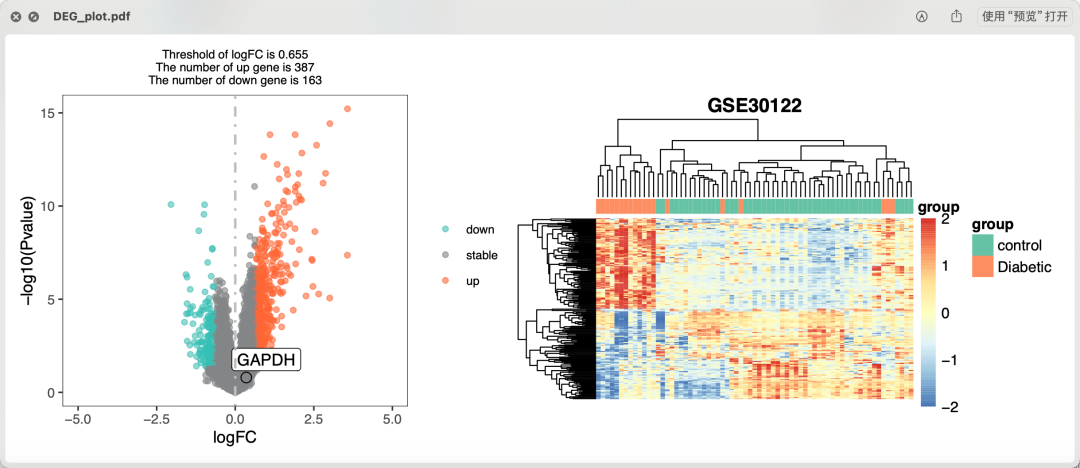
基于zscore表达量矩阵
可以看到, 火山图和热图,肉眼看起来并没有太大的问题哦。当然了,这个时候并不能说明差异分析的合理性,因为毕竟GSE30122这个数据集的作者给出来的表达量矩阵是被zscore的。
第二次差异分析(基于cel文件)
同样的也是可以走limma这样的差异分析流程的,就有上下调基因,可以绘制火山图和热图,如下所示:
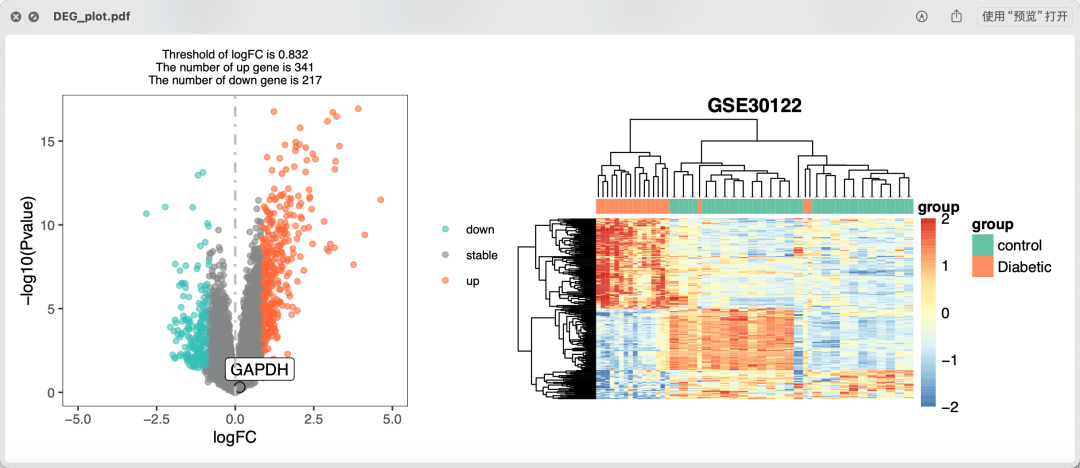
基于cel文件
两次差异分析的比较
这个时候需要载入上面的两个表达量矩阵的各自的差异分析矩阵,首先看看变化倍数的散点图,然后看各自的阈值筛选到的统计学显著的上下调差异基因的冲突性。如下所示:
rm(list = ls())
library(data.table)
cel_deg = fread(file = '../02-GSE30122-cel/GSE30122/DEG.csv',data.table = F)
zscore_deg = fread(file = '../01-GSE30122/GSE30122/DEG.csv',data.table = F)
colnames(zscore_deg)
rownames(cel_deg)=cel_deg$name
rownames(zscore_deg)=zscore_deg$name
ids=intersect(rownames(cel_deg),
rownames(zscore_deg))
df= data.frame(
cel_deg = cel_deg[ids,'logFC'],
zscore_deg = zscore_deg[ids,'logFC']
)
library(ggpubr)
ggscatter(df, x = "cel_deg", y = "zscore_deg",
color = "black", shape = 21, size = 3, # Points color, shape and size
add = "reg.line", # Add regressin line
add.params = list(color = "blue", fill = "lightgray"), # Customize reg. line
conf.int = TRUE, # Add confidence interval
cor.coef = TRUE, # Add correlation coefficient. see ?stat_cor
cor.coeff.args = list(method = "pearson", label.sep = "\n")
)
df= data.frame(
cel_deg = cel_deg[ids,'g'],
zscore_deg = zscore_deg[ids,'g']
)
table(df)
gplots::balloonplot(table(df))
总体上来说,两种不同算法的表达量矩阵的差异分析结果一致性还行;
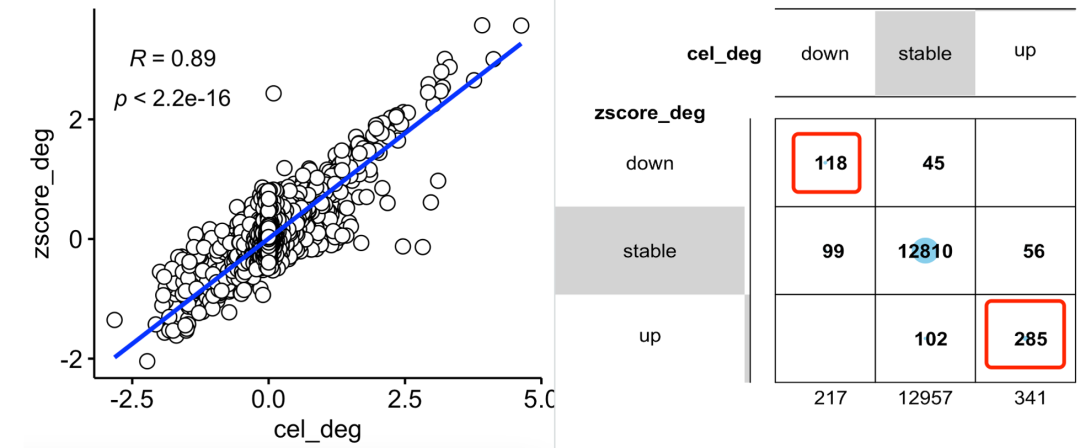
这个时候,可以重点看看两种不同算法的表达量矩阵的差异分析结果的冲突的那些基因,以及一致性的那些基因的功能情况。
symbols_list = split(ids,paste(df[,1],df[,2]))
library(clusterProfiler)
library(org.Hs.eg.db)
library(ReactomePA)
library(ggplot2)
library(stringr)
# 首先全部的symbol 需要转为 entrezID
gcSample = lapply(symbols_list, function(y){
y=as.character(na.omit(AnnotationDbi::select(org.Hs.eg.db,
keys = y,
columns = 'ENTREZID',
keytype = 'SYMBOL')[,2])
)
y
})
gcSample
pro='test'
# 第1个注释是 KEGG
xx <- compareCluster(gcSample, fun="enrichKEGG",
organism="hsa", pvalueCutoff=0.3)
dotplot(xx) + theme(axis.text.x=element_text(angle=45,hjust = 1)) +
scale_y_discrete(labels=function(x) str_wrap(x, width=50))
ggsave(paste0(pro,'_kegg.pdf'),width = 10,height = 8)
可以看到, 冲突的基因列表和一致性的基因列表,都是有生物学功能的
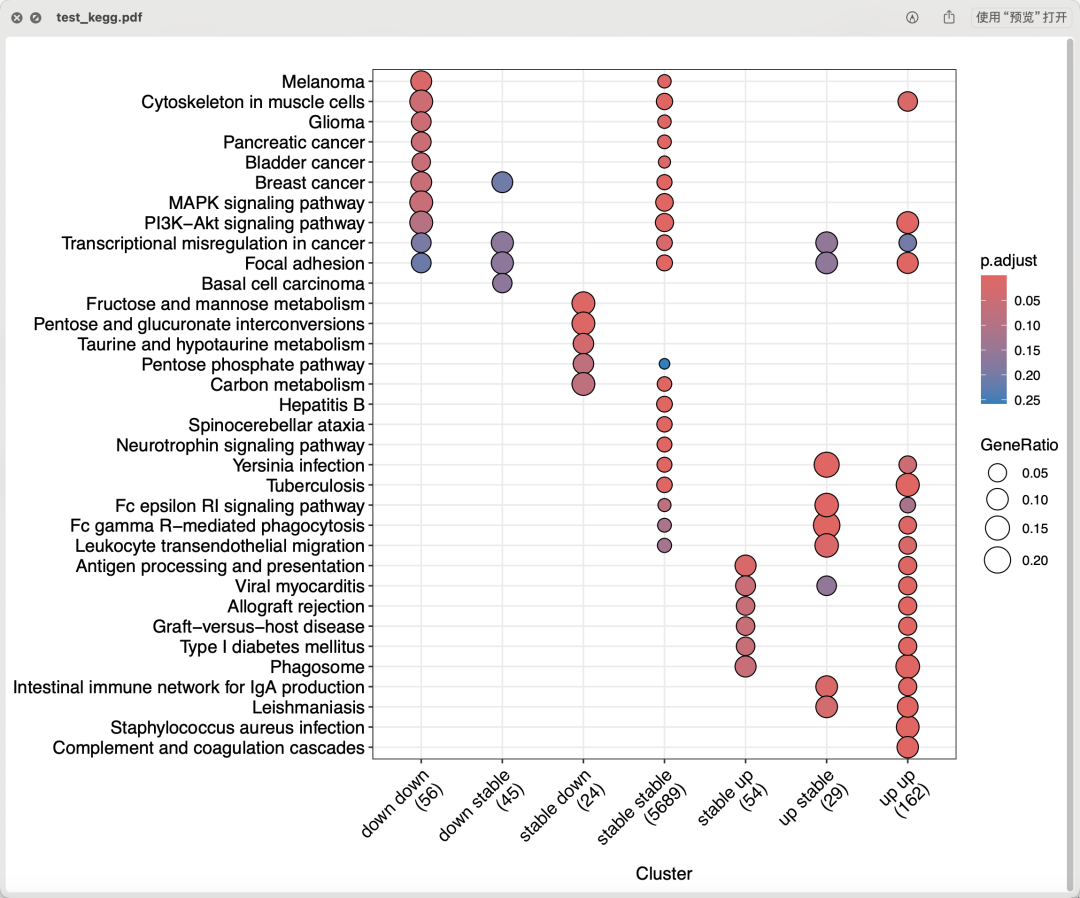
原则上,我们肯定是相信我们从cel文件开始自己制作好的affymetrix的表达量芯片矩阵的差异分析结果啦。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-05-29,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录
