单细胞数据的GSVA
单细胞数据的GSVA和芯片、bulk转录组的GSVA没有本质区别,就使用AverageExpression获取平均表达量得到新的表达矩阵再计算即可。
1.加载数据和R包
获得每种细胞的平均表达量
rm(list = ls())
library(Seurat)
library(GSVA)
library(clusterProfiler)
load("seu.obj.Rdata")
table(Idents(seu.obj))
##
## Naive CD4 T CD14+ Mono B CD8 T NK FCGR3A+ Mono
## 1675 1206 598 406 337 125
## Platelet DC
## 48 88
exp = AverageExpression(seu.obj)[[1]]
#exp = AggregateExpression(seu.obj)[[1]]
exp = as.matrix(exp)
exp = exp[rowSums(exp)>0,]
exp[1:4,1:4]
## Naive CD4 T CD14+ Mono B CD8 T
## TSPAN6 0.01890007 0.000000000 0.00000000 0.00446691
## DPM1 0.50764534 0.398461857 0.52602493 0.49951298
## SCYL3 0.10701976 0.049771894 0.10397003 0.12101561
## C1orf112 0.02653607 0.005093801 0.05426134 0.02747031
Seurat v5 提示建议用AggregateExpression做伪bulk转录组分析,那个是用来求和的,目前查到的文献和教程都是使用平均值,这里就木有改动.
2.做GSVA
gmt文件下载自GSEA-msigdb官网
h_df = read.gmt("h.all.v2023.2.Hs.symbols.gmt")[,c(2,1)]
h_list = unstack(h_df)
ES = gsva(exp, h_list)
ES[1:4,1:4]
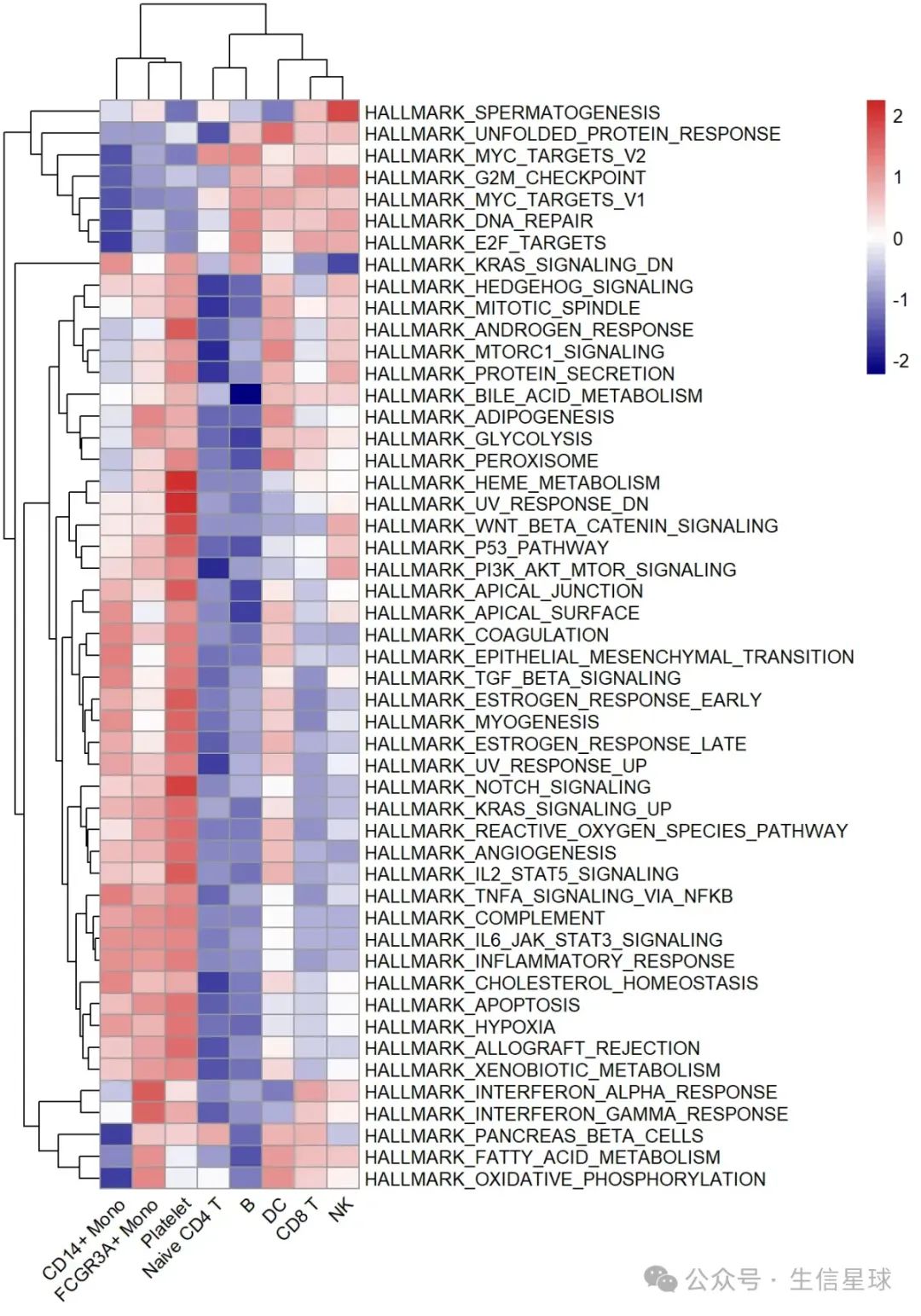
3.热图可视化
library(pheatmap)
pheatmap(ES, scale = "row",angle_col = "45",
color = colorRampPalette(c("navy", "white", "firebrick3"))(50))
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-06-25,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

