转录组数据过滤-4
生信技能树学习笔记
数据过滤条件
测序得到的原始序列含有接头序列或低质量序列,为了保证信息分析的准确性,
需要对原始数据进行质量控制,得到高质量序列(即Clean Reads),原始序
列质量控制的标准为:
(1) 去除含接头的reads;
(2) 过滤去除低质量值数据,确保数据质量;
(3) 去除含有N(无法确定碱基信息)的比例大于5%的reads;(可以根据实际情况)
数据过滤-trim_galore(第一个可选择软件)
trim_galore官网:http://www.bioinformatics.babraham.ac.uk/projects/trim_galore/
常用参数
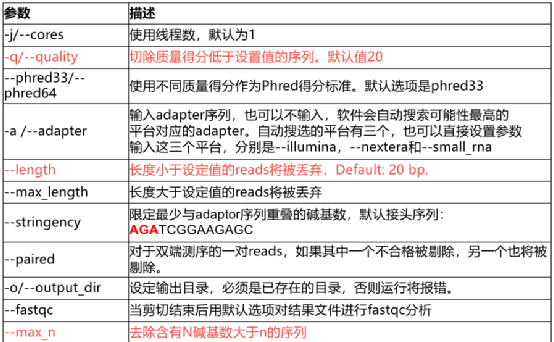
注意大小写
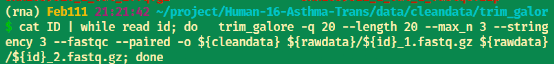
# 激活小环境conda activate rna# 新建文件夹trim_galorecd $HOME/project/Human-16-Asthma-Trans/data/cleandata/trim_galore # 先生成一个变量,为样本IDls $HOME/project/Human-16-Asthma-Trans/data/rawdata/*_1.fastq.gz | awk -F'/' '{print $NF}' | cut -d'_' -f1 >ID # 多个样本 vim trim_galore.sh,以下为sh的内容rawdata=$HOME/project/Human-16-Asthma-Trans/data/rawdatacleandata=$HOME/project/Human-16-Asthma-Trans/data/cleandata/trim_galorecat ID | while read iddo trim_galore -q 20 --length 20 --max_n 3 --stringency 3 --fastqc --paired -o ${cleandata} ${rawdata}/${id}_1.fastq.gz ${rawdata}/${id}_2.fastq.gzdone # 提交任务到后台 可以 用bash或者sh都行nohup bash trim_galore.sh >trim_galore.log & # 使用MultiQc整合FastQC结果multiqc *.zip |
|---|
## 补充技巧:使用掐头去尾获得样本IDls $rawdata/*_1.fastq.gz | while read iddoname=${id##*/}name=${name%_*} trim_galore -q 20 --length 20 --max_n 3 --stringency 3 --fastqc --paired -o ${cleandata} ${rawdata}/${name}_1.fastq.gz ${rawdata}/${name}_2.fastq.gzdone |
|---|
任务管理
1.任务投递:
前台运行:直接运行
后台运行:nohup,&
前台转后台/后台转前台:bg/fg
2.终止任务:
暂停:Ctrl+Z
终止:Ctrl+C,kill
3.任务查看:
top
ps fxww
jobs
示例
前台运行命令
暂停命令
Ctrl+Z
查看命令ID
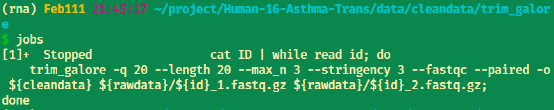
前台转后台
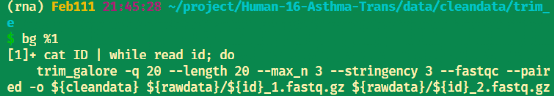
杀程序
后台:Kill -9 %1
前台:Ctrl+C
如何检查脚本内容:echo命令

使用echo将命令打印出来查看是否变量等有错误
数据过滤数据过滤-trim_galore运行结果

第二种数据过滤软件——fastp
https://github.com/OpenGene/fastp
特点:快
fastp常用参数

注意大小写
小技巧:\的妙用
表示手动换行,命令较长时,可以手动换行让命令可读性更高,更美观。
Note:\的后面不能有空格
cd $HOME/project/Human-16-Asthma-Trans/data/cleandata/fastp # 定义文件夹:vim fastp.shcleandata=$HOME/project/Human-16-Asthma-Trans/data/cleandata/fastp/rawdata=$HOME/project/Human-16-Asthma-Trans/data/rawdata/cat ../trim_galore/ID | while read iddofastp -l 20 -q 20 --compression=6 \ -i ${rawdata}/${id}_1.fastq.gz \ -I ${rawdata}/${id}_2.fastq.gz \ -o ${cleandata}/${id}_clean_1.fq.gz \ -O ${cleandata}/${id}_clean_2.fq.gz \ -R ${cleandata}/${id} \ -h ${cleandata}/${id}.fastp.html \ -j ${cleandata}/${id}.fastp.jsondone # 运行fastp脚本nohup bash fastp.sh >fastp.log & |
|---|

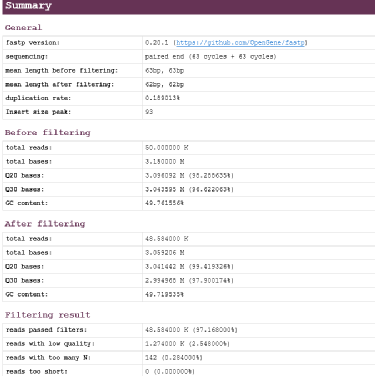
报告解读
总结
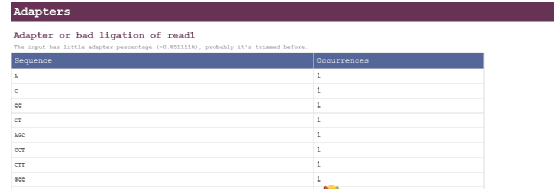
去掉的接头
过滤前后的碱基质量
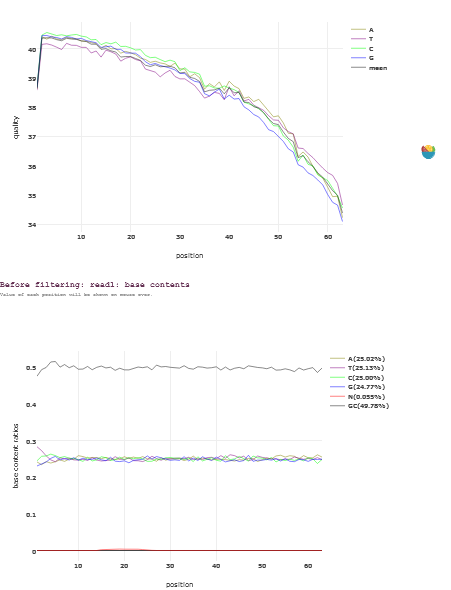
碱基组合排列:颜色越深出现越多。

