🤩 Mime1 | 终极机器学习算法大组合筛选你的最佳模型(一)(预后模型篇)
🤩 Mime1 | 终极机器学习算法大组合筛选你的最佳模型(一)(预后模型篇)
生信漫卷
发布于 2024-07-12 16:08:36
发布于 2024-07-12 16:08:36
写在前面
终于到了周六,这周手术做的有点多了。🫠
下周又是工作量满满的一周。🙃
今天讲一下机器学习的神包,Mime1,之所以写这个名字,是怕大家和Mime包混淆,影响安装。😂
用到的包
rm(list = ls())
# if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")
#
# depens<-c('GSEABase', 'GSVA', 'cancerclass', 'mixOmics', 'sparrow', 'sva' , 'ComplexHeatmap' )
#
# for(i in 1:length(depens)){
# depen<-depens[i]
# if (!requireNamespace(depen, quietly = TRUE)) BiocManager::install(depen,update = FALSE)
# }
#
# if (!requireNamespace("CoxBoost", quietly = TRUE))
# devtools::install_github("binderh/CoxBoost")
#
# if (!requireNamespace("fastAdaboost", quietly = TRUE))
# devtools::install_github("souravc83/fastAdaboost")
#
# if (!requireNamespace("Mime", quietly = TRUE))
# devtools::install_github("l-magnificence/Mime")
library(Mime1)
示例数据
load("./Example.cohort.Rdata")
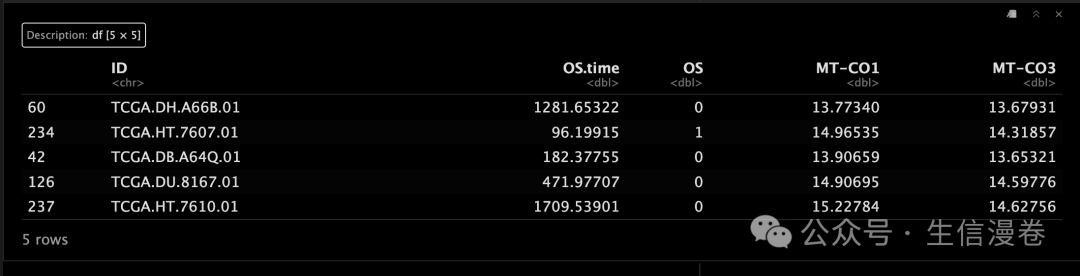
list_train_vali_Data[["Dataset1"]][1:5,1:5]
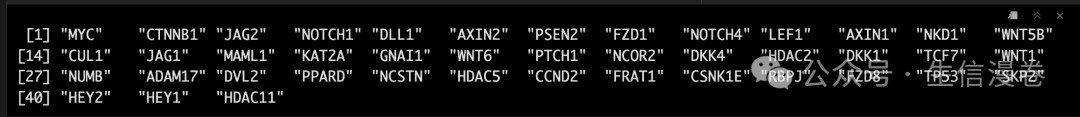
load("./genelist.Rdata")
genelist
创建预后模型并选择最优模型
来小试牛刀一下吧。😘
res <- Mime1::ML.Dev.Prog.Sig(train_data = list_train_vali_Data$Dataset1,
list_train_vali_Data = list_train_vali_Data,
unicox.filter.for.candi = T,
unicox_p_cutoff = 0.05,
candidate_genes = genelist,
mode = 'all', ## 'all', 'single', and 'double'
nodesize =5,
seed = 123
)
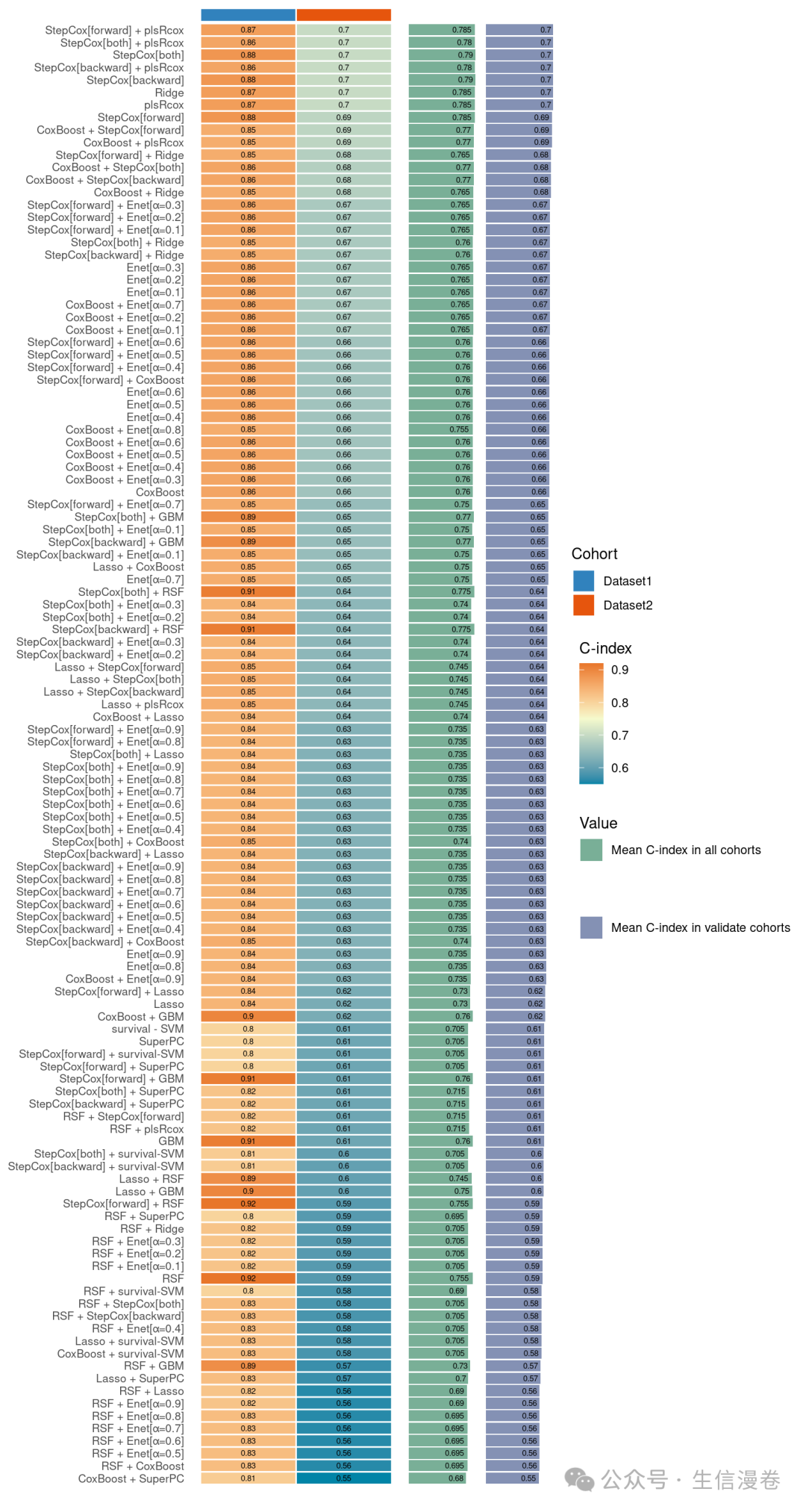
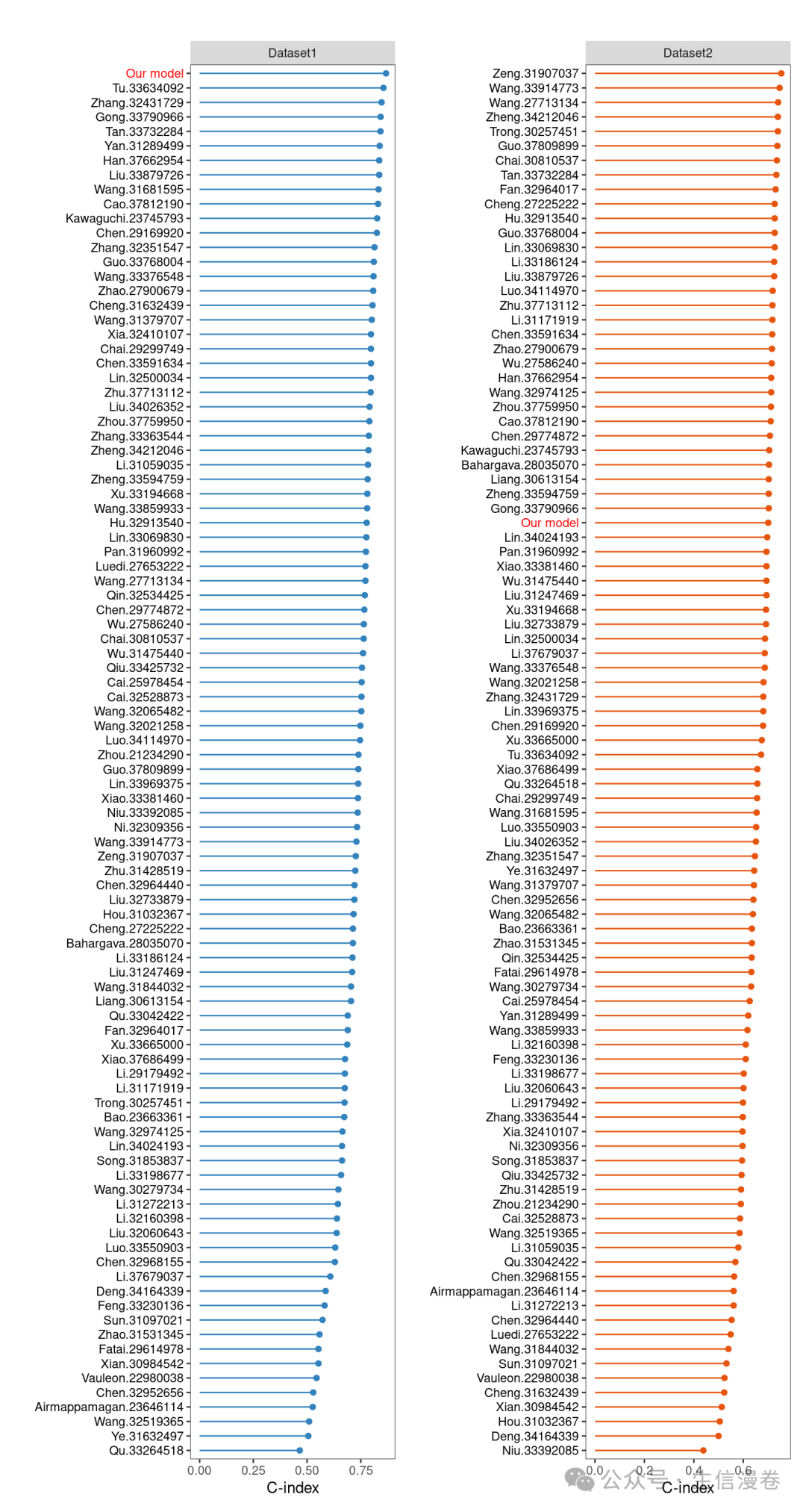
可视化C-index。😘
cindex_dis_all(res,
validate_set = names(list_train_vali_Data)[-1],
order = names(list_train_vali_Data),
width = 0.35
)
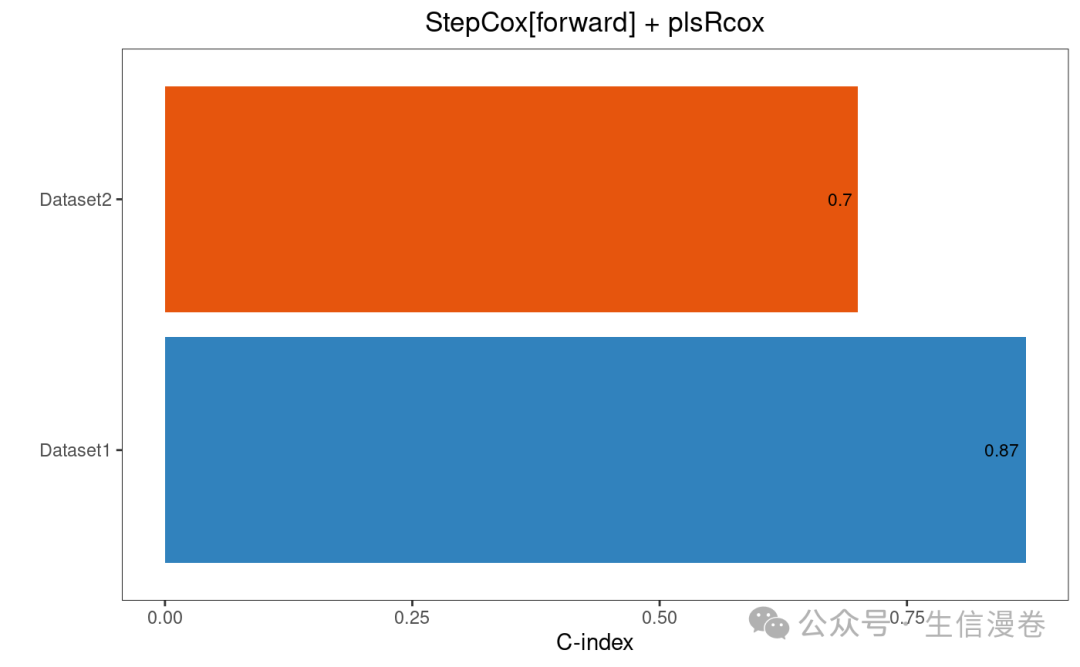
展示指定模型的C-index。🙊
cindex_dis_select(res,
model="StepCox[forward] + plsRcox",
order= names(list_train_vali_Data))
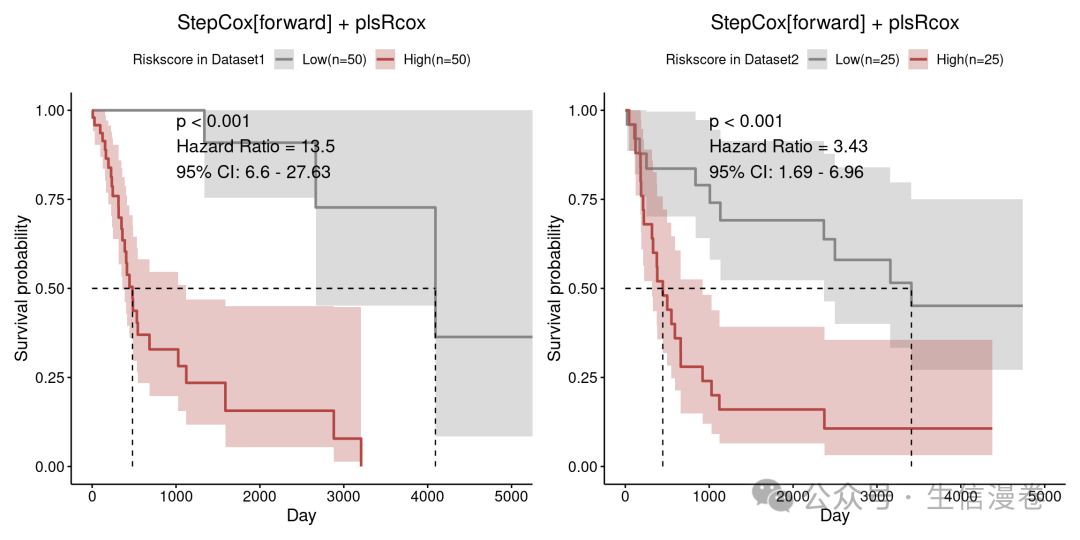
根据特定模型计算的风险评分,绘制不同数据集中患者的生存曲线。💪
survplot <- vector("list",2)
for (i in c(1:2)) {
print(survplot[[i]]<-rs_sur(res, model_name = "StepCox[forward] + plsRcox",
dataset = names(list_train_vali_Data)[i],
#color=c("blue","green"),
median.line = "hv",
cutoff = 0.5,
conf.int = T,
xlab="Day",pval.coord=c(1000,0.9))
)
}
aplot::plot_list(gglist=survplot,ncol=2)
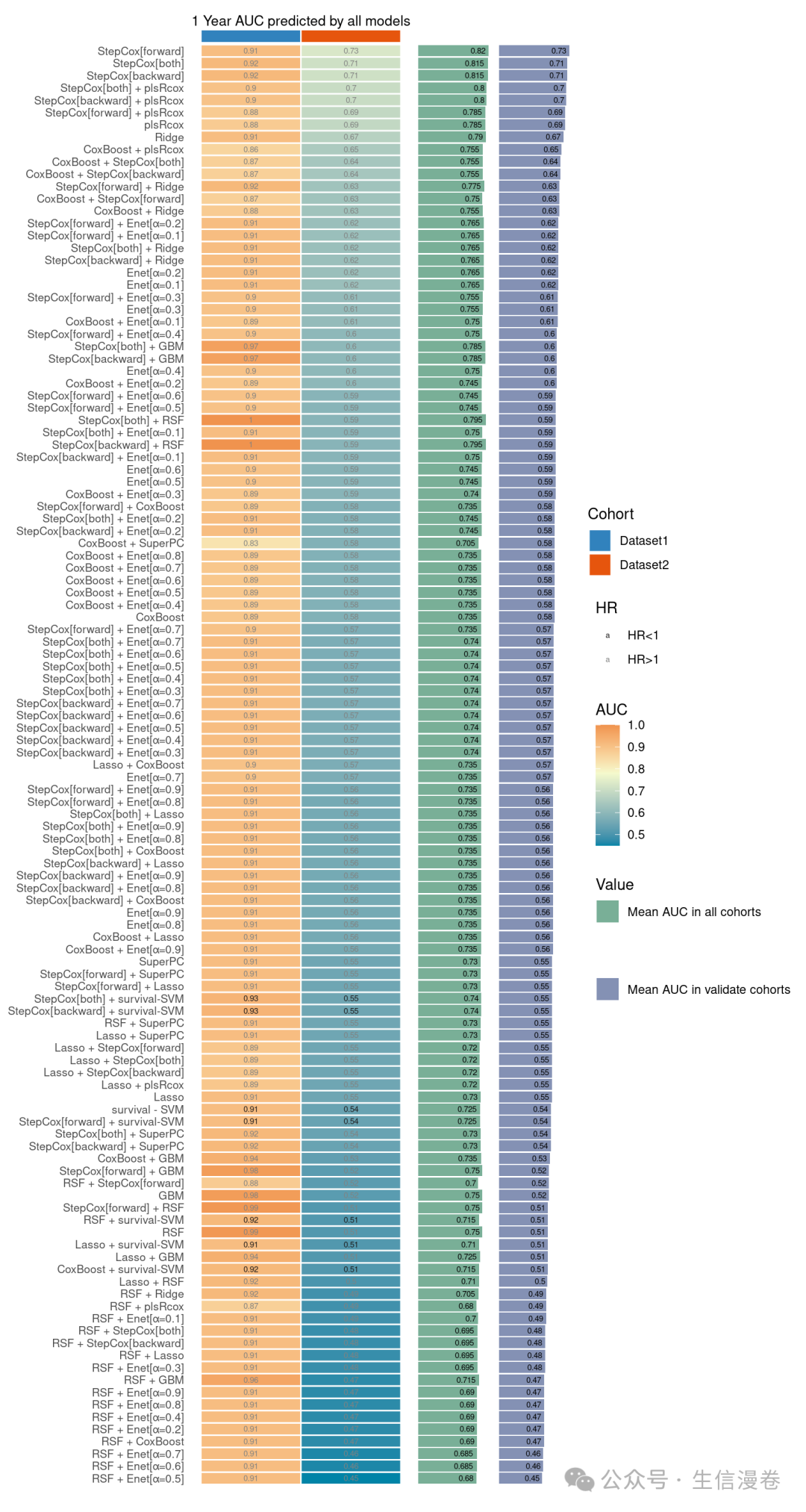
计算模型的AUC
all.auc.1y <- cal_AUC_ml_res(res.by.ML.Dev.Prog.Sig = res,train_data = list_train_vali_Data[["Dataset1"]],
inputmatrix.list = list_train_vali_Data,mode = 'all',AUC_time = 1,
auc_cal_method="KM")
all.auc.3y <- cal_AUC_ml_res(res.by.ML.Dev.Prog.Sig = res,train_data = list_train_vali_Data[["Dataset1"]],
inputmatrix.list = list_train_vali_Data,mode = 'all',AUC_time = 3,
auc_cal_method="KM")
all.auc.5y <- cal_AUC_ml_res(res.by.ML.Dev.Prog.Sig = res,train_data = list_train_vali_Data[["Dataset1"]],
inputmatrix.list = list_train_vali_Data,mode = 'all',AUC_time = 5,
auc_cal_method="KM")
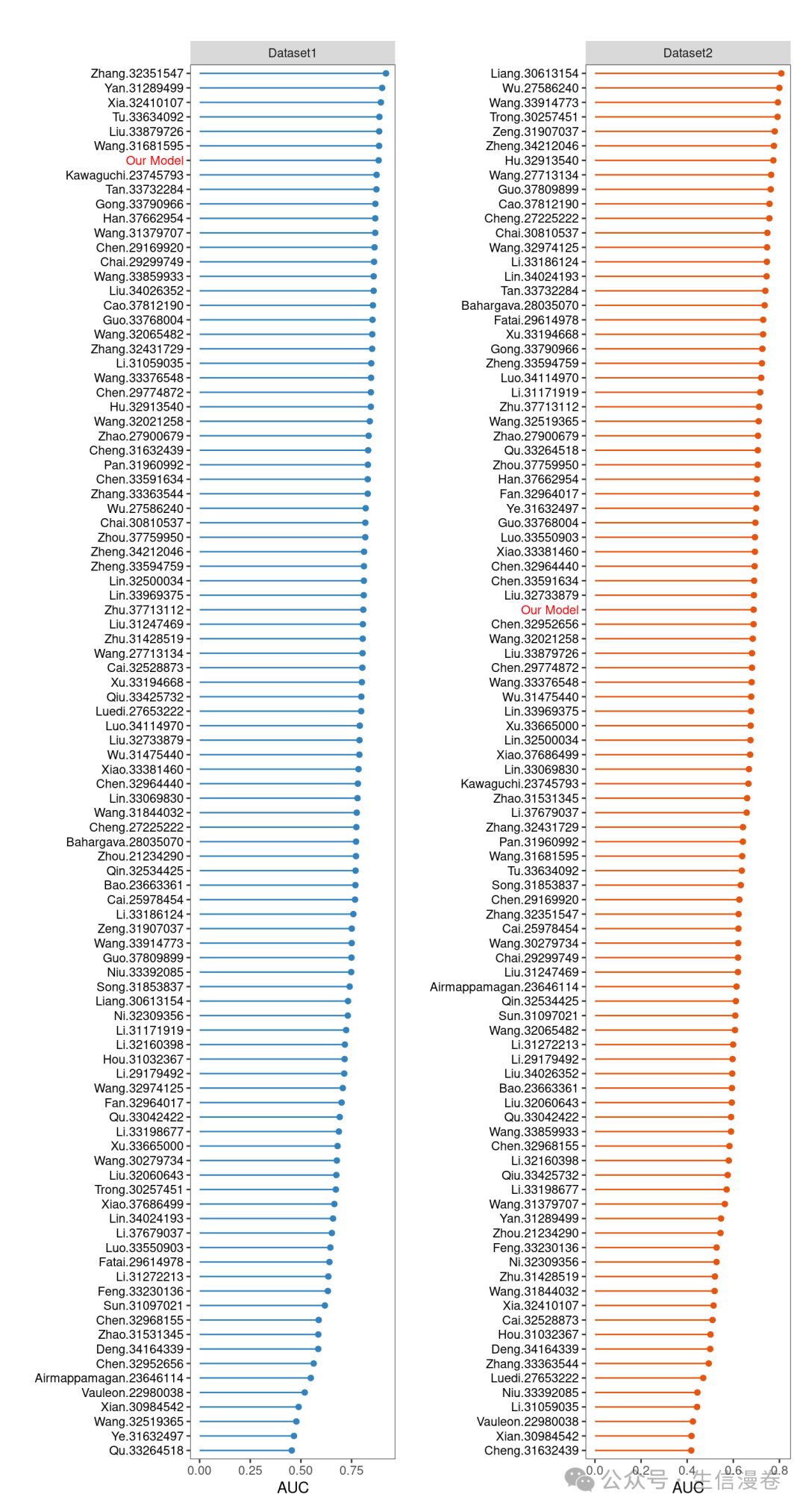
可视化AUC。😘
这里我们只展示一下1年的。😏
auc_dis_all(all.auc.1y,
dataset = names(list_train_vali_Data),
validate_set=names(list_train_vali_Data)[-1],
order= names(list_train_vali_Data),
width = 0.35,
year=1)
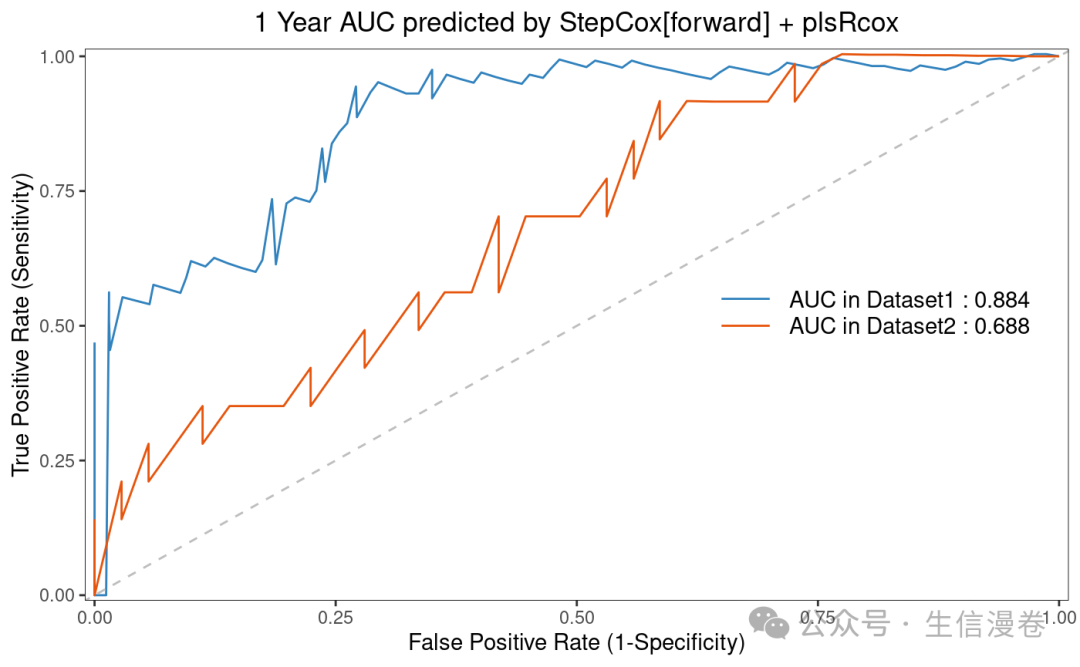
绘制不同数据集中特定模型的ROC曲线。😘
roc_vis(all.auc.1y,
model_name = "StepCox[forward] + plsRcox",
dataset = names(list_train_vali_Data),
order= names(list_train_vali_Data),
anno_position=c(0.65,0.55),
year=1)
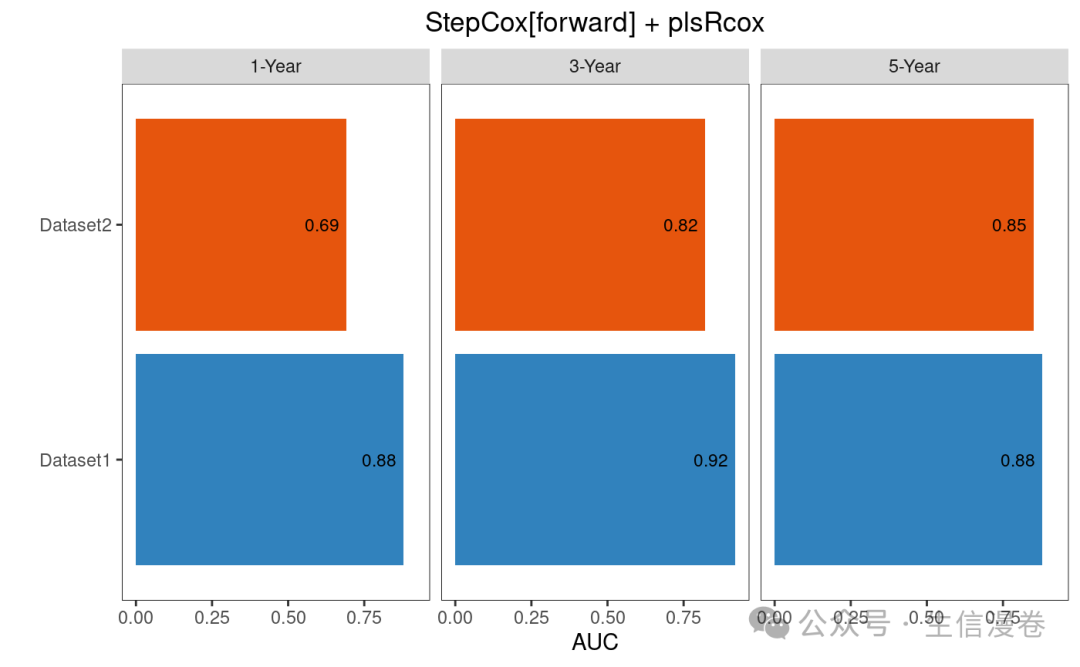
绘制不同数据集中特定模型的1、3、5年的AUC。😘
auc_dis_select(list(all.auc.1y,all.auc.3y,all.auc.5y),
model_name="StepCox[forward] + plsRcox",
dataset = names(list_train_vali_Data),
order= names(list_train_vali_Data),
year=c(1,3,5))
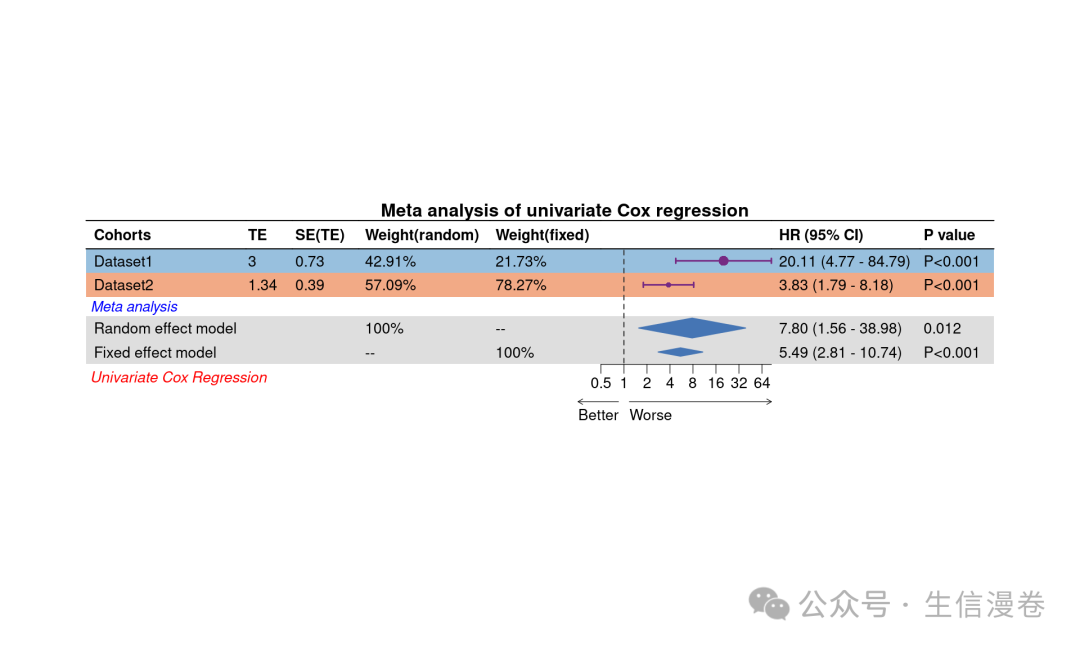
单变量cox回归特定模型的meta分析
unicox.rs.res <- cal_unicox_ml_res(res.by.ML.Dev.Prog.Sig = res,
optimal.model = "StepCox[forward] + plsRcox",
type ='categorical' # 'categorical' or 'continuous'
)
metamodel <- cal_unicox_meta_ml_res(input = unicox.rs.res)
meta_unicox_vis(metamodel,
dataset = names(list_train_vali_Data))
与已知模型相比较
7.1 计算risk score
rs.glioma.lgg.gbm <- cal_RS_pre.prog.sig(use_your_own_collected_sig = F,
type.sig = c('LGG','GBM','Glioma'),
list_input_data = list_train_vali_Data)
可视化比较HR。😘
HR_com(rs.glioma.lgg.gbm,
res,
model_name="StepCox[forward] + plsRcox",
dataset=names(list_train_vali_Data),
type = "categorical")

7.2 计算C-index
cc.glioma.lgg.gbm <- cal_cindex_pre.prog.sig(use_your_own_collected_sig = F,
type.sig = c('Glioma','LGG','GBM'),
list_input_data = list_train_vali_Data)
可视化比较C-index。🌟
cindex_comp(cc.glioma.lgg.gbm,
res,
model_name="StepCox[forward] + plsRcox",
dataset=names(list_train_vali_Data))
7.3 计算AUC
auc.glioma.lgg.gbm.1 <- cal_auc_pre.prog.sig(use_your_own_collected_sig = F,
type.sig = c('Glioma','LGG','GBM'),
list_input_data = list_train_vali_Data,AUC_time = 1,
auc_cal_method = 'KM')
可视化比较AUC。😘
auc_comp(auc.glioma.lgg.gbm.1,
all.auc.1y,
model_name="StepCox[forward] + plsRcox",
dataset=names(list_train_vali_Data))
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-07-06,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

