中南大学邓磊教授团队提出MolMVC模型,通过多视图对比学习增强药物相关任务的分子表征
中南大学邓磊教授团队提出MolMVC模型,通过多视图对比学习增强药物相关任务的分子表征

有效的分子表征在药物开发中至关重要。分子的复杂性质需要综合的多视图表征,包括1D, 2D和3D特征,以捕捉不同的视角。获得包含这些不同结构的表征对于全面理解药物相关环境中的分子至关重要。
2024年9月4日,中南大学邓磊教授团队在Bioinformatics上发表文章MolMVC: Enhancing molecular representations for drug-related tasks through multi-view contrastive learning。

作者提出了一种针对分子表征学习的多视图对比学习框架,称为MolMVC(Molecular Multi-View Contrastive learning)。MolMVC使用Transformer编码器捕获1D序列信息,并使用图Transformer对分子的复杂2D和3D结构细节进行编码。为了有效地融合多视图分子特征,作者还引入了自适应多视图对比损失(Adaptive Multi-View Contrastive loss, AMCLoss)。实验结果表明,MolMVC在一系列分子性质预测任务中超越了现有方法。
如图1所示,MolMVC框架包括预训练阶段和迁移学习阶段。在预训练阶段,作者首先通过1D、2D和3D分子编码器获取原始样本的表征。随后,作者引入了一种新的注意力引导增强样本生成方法,结合先验知识,为不同的分子数据模式生成阳性样本。然后作者获得这些扩增样品的分子表征。在此之后,作者引入了AMCLoss进行对比学习,同时对齐多视图分子表征。为了利用编码器在局部和全局级别的分层属性,作者对不同级别的三种分子数据模式的表征进行了对比学习。进入迁移学习阶段,下游任务的分子三维信息是稀缺的。得益于作者的预训练策略,作者专门使用1D和2D分子编码器,丰富了3D几何信息。为了获得具有多层信息的表征,作者对来自两个分子模态编码器的局部和全局表征使用了平均操作。随后,作者将这两种表征连接起来,并将它们用作下游任务的预测器的输入。
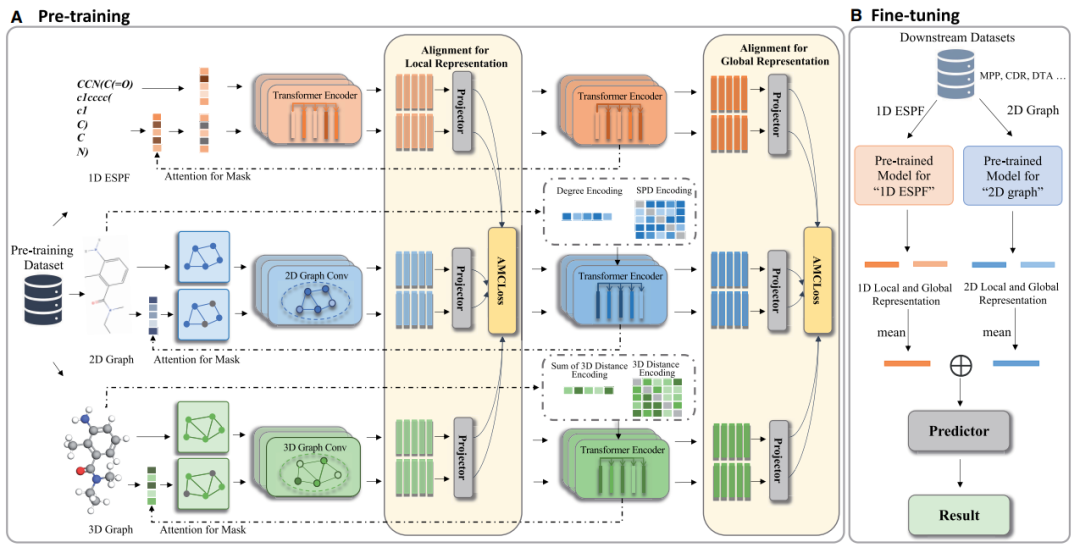
图1 MolMVC结构图
作者使用特定的编码器来嵌入潜在信息。对于1D的分子指纹,作者采用ESPF(Explainable Substructure Partition Fingerprint,可解释的子结构分割指纹),同时使用Transformer编码器,该编码器以其通过多头注意机制提取序列信息的有效性而闻名。Transformer最初是为自然语言处理任务设计的,但其也是从ESPF序列中挖掘上下文信息的合适选择。ESPF成功地识别了关键的官能团,利用嵌入层将该ESPF序列编码为高维空间内的嵌入。为了结合每个子结构的位置信息,通过一个独热编码和另一个嵌入层生成位置嵌入。这两个嵌入的组合产生了输入,包括ESPF嵌入和位置嵌入。
对于2D和3D图数据,作者使用了图Transformer,能够同时从邻近的原子中捕获局部信息和从整个分子中捕获全局信息,模型包括两个主要组件:图编码器和Transformer编码器。作者选择图同构网络(GIN) (Xu et al. 2018)用于二维拓扑图数据,SchNet用于三维几何图数据。GIN是图神经网络的一种变体,以其可量化的泛化能力而闻名。SchNet是一种几何表征学习方法,可以对原子的两两相互作用进行建模。在对GIN和SchNet进行处理后,通过原子表示池化得到全图表示,作为后续Transformer编码器的输入。
与一般的Transformer不同,对于2D图Transformer,作者引入原子的度(即与之有化学键相连的原子的个数)进行位置编码,引入自注意力层的最短路径距离(SPD),对原子对之间的拓扑关系进行编码。对于3D图Transformer,作者引入三维距离来编码分子中原子对之间的几何关系,其中三维距离指的是经过高斯基核函数处理的欧氏距离。
影响对比学习效果的一个关键因素是对原始样本生成的正样本的质量。为了确保作者的模型有效地从每个分子数据模态中捕获关键信息,作者提出了一种创新的掩膜策略,用于基于Transformer和图Transformer的注意力分数生成正样本。注意分数允许模型通过不同的加权分布为输入特征分配不同程度的重要性。通过利用注意分数的大小,作者可以在模型中建立输入特征的重要性排序。利用这个排名作为指导,作者选择屏蔽一定比例的1D ESPF片段,或者屏蔽注意力得分最高的2D和3D图形的原子。这是因为,对于1D分子数据,ESPF作为数据增强的目标,提供子结构信息,对于2D分子数据,在计算注意力分数时考虑SPD,而对于3D分子数据,在计算注意力分数时考虑了三维距离。因此,作者为每个模态获得具有不同掩模的增强样本,增强了预训练中信息的多样性。
对比学习的目标是同时对齐正样本和区分负样本。在分子对比学习的背景下,正样本对的选择可以大致分为两种策略:构建增强样本和利用不同的分子数据模式。然而,如何有效地将这两种策略结合起来,目前还存在研究空白。在这里,作者提出了自适应多视图对比损失(AMCLoss)。这种损失将分子数据的三种模式及其相应的增强分子作为正样本,利用它们的表示进行对比学习。考虑到由不同分子角度的样本组成的正样本对可以有多种形式,并且每种类型的对齐难度不同,训练的目标是在预训练中平衡不同正样本对之间的学习率。为了实现这一点,作者引入了动态权重平均(DWA)方案的概念,该方案最初是为多任务学习开发的。该方案根据损失的变化率调整每个任务的权重。
作者将MolMVC与一些具有代表性的方法进行了比较,如表1所示。在分子性质预测任务中,在BBBP等六个数据集上,将ROC曲线下的面积(AUC,越高越好)作为评测指标。MolMVC在以上数据集上的性能均超越了现有方法。
表1 与其他方法对比

作者设计了消融实验来验证模型设计的有效性。在这里,作者设计了MolMVC的五个变体。NoPretrain移除预训练方案。NoLocal只在全局范围内进行对比学习(不使用局部特征)。No3D在不使用3D分子数据的情况下预训练模型。RandomMask采用随机掩码的增强策略。SupLossPretrain使用有监督损失SupLoss代替AMCLoss。作者对BBBP、BACE和Sider数据集进行消融研究。结果如图2所示。NoPretrain变体性能的显著下降强调了预训练方案在没有标记数据的情况下学习潜在分子知识的有效性。涉及NoLocal的实验表明,MolMVC能够全面捕获来自局部和全局分子的信息。在预训练没有3D分子数据(No3D)的模型时,其性能一直低于MolMVC,这表明在预训练过程中引入3D数据可以有效地将分子几何知识注入到1D和2D编码器中。RandomMask的性能表明,掩膜策略产生更高质量的增强样本。在SupLossPretrain上的实验表明,AMCLoss可以通过平衡不同类型正对的学习率来达到更好的预训练效果。
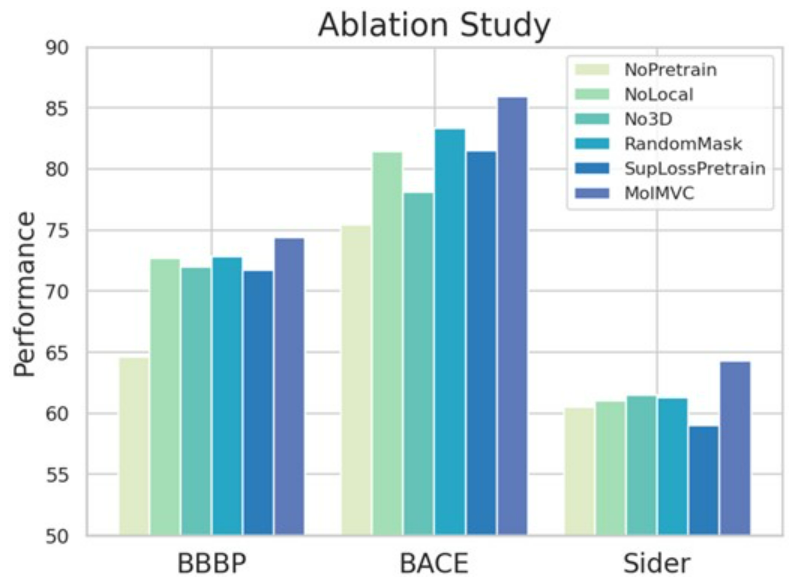
图2 消融实验
作者还进行了案例分析。作者使用t-SNE降维并可视化由MolMVC生成的BBBP数据集的表征。在图3A和B中,蓝色点和橙色点分别代表阳性和阴性样本。经过预训练,同一类的点聚集在一起,而不同类的点分散。较低的Davies Bouldin Index (DBI)表明来自预训练的MolMVC的表示获得更好的聚类性能。
作者还进行了分子检索实验。在BBBP数据集中随机选择两个分子作为查询分子,其余分子作为参考分子。计算查询分子与参考分子的MolMVC表示余弦相似度,排名前五的分子如图3C所示。值得注意的是,这些分子具有相似的结构。传统的分子指纹,即Morgan和ECFP指纹,被用来验证它们的相似性。结果表明这些分子具有很高的相似性。经过预训练,最初距离较远的分子变得更近,而最初距离较近的分子仍然很近。这些结果表明,MolMVC有助于模型学习分子知识和揭示分子之间的内在联系。
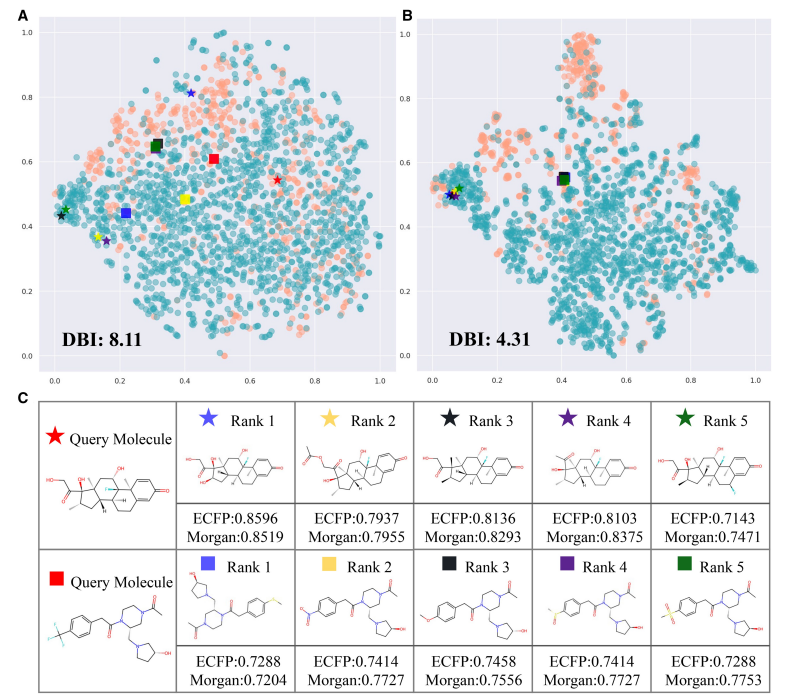
图3 案例分析
为了研究模型的注意力分数是否能够识别分子的关键部分,作者将分子的三种数据形式的注意力分数可视化。如图4所示,从预训练数据集中随机选择两个分子作为示例。每个部分或原子的颜色对应于一个色条梯度,最亮的在底部,最暗的在顶部,按比例变化。从可视化结果来看,有以下观察结果。首先,三种数据模式的注意力得分都集中在卤素原子上:图4A中的氯和图4B中的溴。卤素通常用于增强分子的脂溶性,帮助药物通过生物膜。其次,在2D和3D中,含氮组的注意力得分更高。含氮基团通常在生物体中起着至关重要的作用,参与氢键的形成,与蛋白质和核酸等生物分子相互作用,或作为药物分子的靶标。第三,二维注意力评分更侧重于不同子结构之间的连接。最后,三种模式的编码器表现出不同的注意模式,导致分子的不同部分被掩盖。在预训练阶段,这种多样性增加了正样本对中包含的信息。
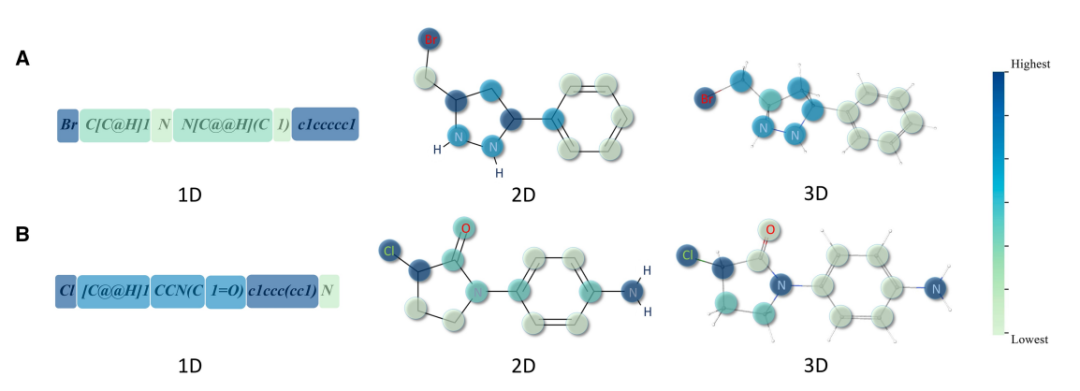
图4 注意力分数可视化
在本文中,作者引入了一种新的多视图对比学习框架MolMVC用于分子表征。MolMVC同时考虑分子的1D、2D和3D模式数据,利用提出的AMCLoss在对比学习中结合多视图信息。实验展示了MolMVC在多个基准测试中的先进性能,证明了其在药物相关任务中的有效性。
参考文献:
Huang et al. MolMVC: Enhancing molecular representations for drug-related tasks through multi-view contrastive learning. Bioinformatics. 2024
