能根据差异基因来反推样品的分组情况吗
通常情况下,我们拿到了表达量矩阵,就需要有样品信息,可以是癌症和癌旁,疗效好坏,处理前后等等都能用来把样品进行分组。然后就可以愉快的进行差异分析了,如果是转录组测序, 是需要count矩阵然后使用金标准算法(DESeq2,edgeR,limma-voom)计算。
比如2024年1月的文章:《End-to-end prognostication in colorectal cancer by deep learning: a retrospective, multicentre study》,就是针对tcga数据库里面的crc数据集的转录组测序后的count矩阵按照他们自己的预后模型把病人分成高低风险组,然后差异分析:

高低风险组,然后差异分析
大多数情况下,我们没办法直接复现人家的模型,而且很多模型甚至就是一个黑匣子。
既然是不能使用模型我们就没办法得到同样的分组信息,除非作者在附件给出来了样品信息表格,不过上面的文章并没有给出来。
但是呢, 文章给出来了差异基因列表:
By performing a bioinformatics analysis on the TCGA- CRC cohort, we identified 113 DEGs between the high-risk and low-risk groups
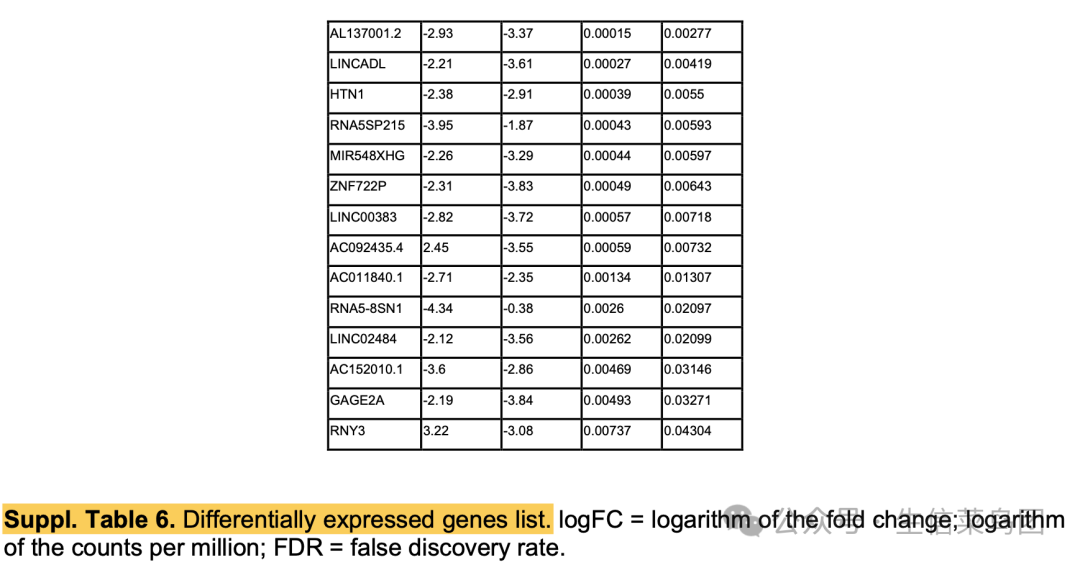
差异基因列表
我突然奇想,因为有了这113个基因的logFC和FDR,理论上是不是可以反推出来样品的分组情况呢?
大家试试看
这个tcga数据库里面的crc数据集的转录组测序后的count矩很容易下载,然后上面的文献里面的附件表格6里面的基因很容易提取出来,首先取这113个基因的表达量矩阵的子集绘制热图,样品层次聚类图,样品的pca图,就可以试试看反推样品的二分组情况。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-02-01,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录
