Nat. Commun. | 基于 Transformer 架构的序列水平虚拟筛选方法在药物研发中的突破性进展
Nat. Commun. | 基于 Transformer 架构的序列水平虚拟筛选方法在药物研发中的突破性进展
用户1151118
发布于 2026-01-08 13:04:10
发布于 2026-01-08 13:04:10
Sequence-based virtual screening using transformers
期刊: Nature Communications 链接: https://doi.org/10.1038/s41467-025-61833-8 代码: https://github.com/zshengyu14/LigandTransformer 简介: 该论文提出了一种基于 Transformer 架构的深度学习方法 Ligand-Transformer,其创新点在于通过序列水平的输入实现蛋白质 - 配体相互作用的预测,突破了传统方法对游离态分子构象的依赖。该方法以蛋白质的氨基酸序列和小分子的拓扑结构为输入,利用 AlphaFold2 和 GraphMVP 框架生成表征,通过跨模态注意力网络处理后,输出结合亲和力和构象空间的距离矩阵。实验中,在 PDBbind2020 数据集上与其他方法对比,表现出更优或相当的相关性;在 EGFR激酶抑制剂筛选中命中率达 58%,发现了低纳摩尔级效力的化合物;对 ABL 激酶抑制剂的构象选择性预测也与实验结果高度一致。该论文表明 Ligand-Transformer 能准确预测蛋白质与小分子的相互作用,为药物设计的初始步骤提供了高效工具。

在当代药物研发领域,蛋白质-配体相互作用的精准预测是推动新药发现的核心环节。传统高通量实验筛选不仅成本高昂、耗时漫长,且受限于假阳性/阴性率过高等问题,难以满足现代药物研发的效率需求。近期,发表于《Nature Communications》的研究论文《Sequence-based virtual screening using transformers》提出了一种基于Transformer架构的深度学习方法——Ligand-Transformer,通过序列水平的输入实现蛋白质-配体复合物构象空间与结合亲和力的精准预测,为药物设计领域带来了范式革新。本文将系统解析该方法的技术原理、性能验证及应用价值。
研究背景与技术突破
蛋白质-配体相互作用贯穿于几乎所有生物过程,其结合亲和力与构象动态是药物设计的关键指标。近年来,深度学习技术在蛋白质结构预测领域的突破性进展(如AlphaFold系列)为拓展至蛋白质-配体相互作用预测奠定了基础,但现有方法仍存在显著局限:基于结构的药物设计依赖游离态分子构象,难以捕捉复合物形成过程中的动态变化;基于配体的方法则受限于已知配体的结构特征迁移性不足。
Ligand-Transformer的核心创新在于构建了序列-拓扑驱动的预测框架:以蛋白质氨基酸序列和小分子拓扑结构为输入,直接输出复合物的构象空间分布(以距离矩阵表征)及结合亲和力,无需依赖游离态分子的三维结构。这种设计突破了传统方法对预处理结构数据的依赖,实现了从"静态结构依赖"向"动态序列解析"的转变,为未知靶点的虚拟筛选提供了全新可能。
模型架构与技术细节
Ligand-Transformer的架构设计融合了多模态信息处理的前沿思路,其核心由三个模块构成:
- • 表征生成层:采用AlphaFold2的Transformer框架提取蛋白质序列的中间表征(包括MSA特征、残基对特征及结构模块输出),同时利用GraphMVP框架处理小分子的2D分子图,通过自监督学习将3D几何信息嵌入2D图编码器,形成兼具拓扑特征与空间先验的配体表征。
- • 跨模态交互层:通过交叉注意力网络实现蛋白质与配体表征的信息交换,重点捕捉残基-原子间的距离约束与相互作用模式,解决了不同模态数据(序列vs分子图)的语义鸿沟问题。
- • 预测输出层:设计双预测头结构——亲和力预测头输出pKd/pIC50等量化指标,距离预测头生成残基-原子间的距离分布矩阵,共同构成复合物相互作用的完整描述。
这种架构设计的优势在于:通过复用AlphaFold2与GraphMVP的预训练知识,大幅降低了模型对标注数据量的依赖;中间表征的有效利用避免了直接预测三维结构带来的计算负担,使序列水平的快速筛选成为可能。
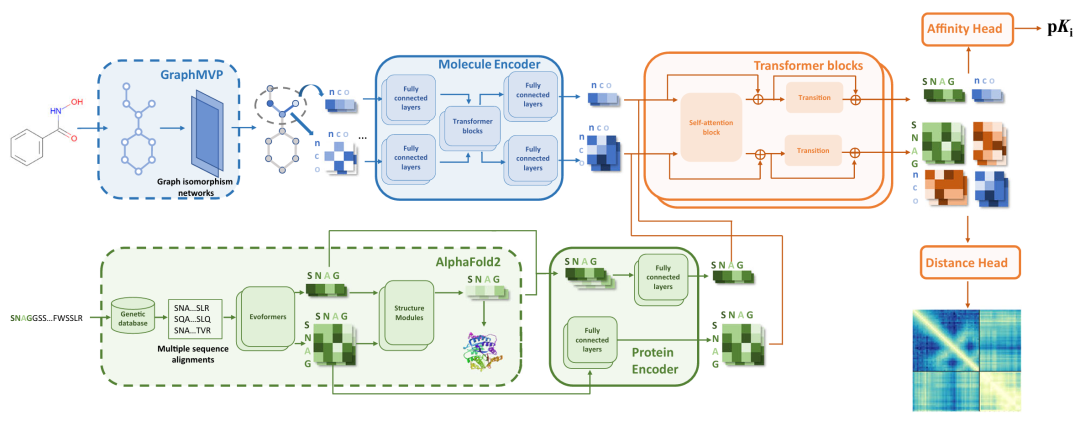
该图展示了 Ligand-Transformer 的模型结构,其将蛋白质 - 配体复合物表示为异质图,整合了蛋白质的残基和配体的原子集及成对特征。该图由 AlphaFold2 生成的蛋白质输入和 GraphMVP 生成的配体输入构建初始完整图,再经 12 层类 Transformer 网络优化,通过自注意力和成对偏置更新节点和边的表征。输出经亲和力头用于结合亲和力预测,经距离头用于距离分布预测。虚线框内组件参数固定,实线框内为可训练参数。
该图展示了 Ligand-Transformer 的模型结构,其将蛋白质 - 配体复合物表示为异质图,整合了蛋白质的残基和配体的原子集及成对特征。该图由 AlphaFold2 生成的蛋白质输入和 GraphMVP 生成的配体输入构建初始完整图,再经 12 层类 Transformer 网络优化,通过自注意力和成对偏置更新节点和边的表征。输出经亲和力头用于结合亲和力预测,经距离头用于距离分布预测。虚线框内组件参数固定,实线框内为可训练参数。
性能验证与基准对比
研究团队在多个基准数据集上对Ligand-Transformer进行了系统验证,其性能表现显著优于现有方法:
- • 亲和力预测精度:在PDBbind2020数据集(13,420个蛋白质-配体复合物)上,该模型与实验测量值的相关性(Pearson's R)显著高于HAC-Net、TankBind等主流方法,尤其在低数据量场景下表现出更强的泛化能力。
- • 构象预测准确性:对残基-残基距离的预测误差95%以上低于0.5Å,残基-配体原子距离误差95%在2Å以内,表明模型能够精准捕捉复合物的关键空间约束。
- • 计算效率:与传统分子对接方法(如Vina-GPU)和共折叠方法(Boltz-1)相比,Ligand-Transformer的筛选速度提升两个数量级以上,对10,000个分子的虚拟筛选可在常规计算资源下高效完成。
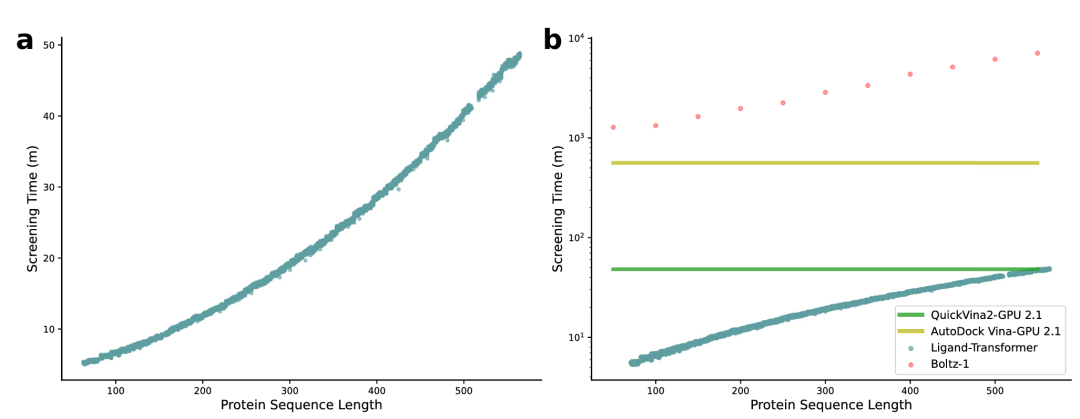
该图展示了 Ligand-Transformer(a)与其他主流对接方法(b)筛选 10,000 个分子的估计时间,结果显示 Ligand-Transformer 比 Boltz-1 快两个数量级,体现了其在计算效率上的显著优势,更适用于大规模虚拟筛选。
该图展示了 Ligand-Transformer(a)与其他主流对接方法(b)筛选 10,000 个分子的估计时间,结果显示 Ligand-Transformer 比 Boltz-1 快两个数量级,体现了其在计算效率上的显著优势,更适用于大规模虚拟筛选。
这些结果证实,Ligand-Transformer在预测精度与计算效率之间实现了优异平衡,为大规模虚拟筛选提供了实用工具。
实际应用与案例验证
EGFR耐药突变体抑制剂的发现
表皮生长因子受体(EGFR)的L858R/T790M/C797S(LTC)三重突变是肺癌治疗中的重大挑战,对现有抑制剂普遍耐药。研究团队利用Ligand-Transformer开展针对性筛选:
- 1. 模型微调:在包含290种已知抑制剂的EGFRLTC-290数据集上进行十折交叉验证,微调后模型预测IC50值的Pearson's R从0.57提升至0.88,表明模型对该特定靶点的适配性显著增强。
- 2. 虚拟筛选:对TargetMol库中的9,090种化合物进行筛选,基于11个模型(1个基础模型+10个微调模型)的一致性预测,最终选定12个候选化合物。
- 3. 实验验证:6个化合物表现出抑制活性,其中C1(萘啶衍生物)和C10的IC50值分别达到5.5nM和1.2nM,且具有全新化学骨架,为后续优化提供了优质起点。
尤为重要的是,模型通过预测E762与G857残基距离,成功区分了抑制剂的结合模式(正交位vs变构位),揭示了αC-螺旋构象变化与抑制活性的关联机制,为靶点理性设计提供了分子层面的解释。
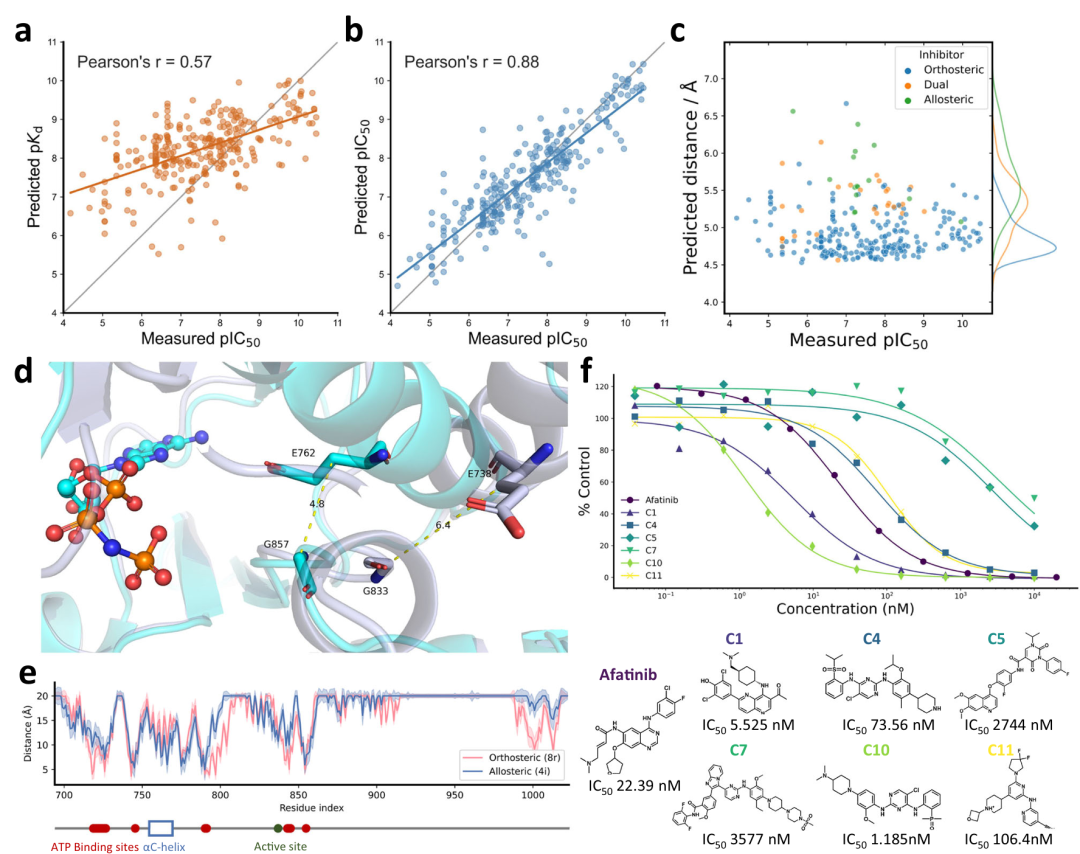
该图包含多个子图,a 和 b 分别展示了 Ligand-Transformer 在 EGFR-290 数据集上未使用迁移学习和使用迁移学习后,预测的结合参数与实验 plC₅₀值的相关性,后者 Pearson 相关系数提升至 0.88;c 展示了不同配体结合状态下 EGFR 的 E762 和 G857 残基间预测距离分布及结合模式;d 对比了 EGFR 激酶结构域活性与非活性状态的 X 射线结构;e 为配体与残基的预测距离线图;f 显示了所选化合物的体外激酶抑制实验结果及 IC50 值。
该图包含多个子图,a 和 b 分别展示了 Ligand-Transformer 在 EGFR-290 数据集上未使用迁移学习和使用迁移学习后,预测的结合参数与实验 plC₅₀值的相关性,后者 Pearson 相关系数提升至 0.88;c 展示了不同配体结合状态下 EGFR 的 E762 和 G857 残基间预测距离分布及结合模式;d 对比了 EGFR 激酶结构域活性与非活性状态的 X 射线结构;e 为配体与残基的预测距离线图;f 显示了所选化合物的体外激酶抑制实验结果及 IC50 值。
ABL激酶构象选择性预测
ABL激酶的构象动态(活性态A、非活性态I1/I2)与其功能调控密切相关,是设计高选择性抑制剂的关键。Ligand-Transformer通过预测残基距离矩阵,实现了构象种群迁移的精准预测:
- • 对12种已知选择性抑制剂的测试显示,11种化合物的预测主要结合构象与NMR实验结果一致。
- • 通过分析V308-F401(αC-螺旋与DFG基序)等关键残基对的距离分布,模型成功捕捉了不同抑制剂诱导的构象偏好性,如I2态抑制剂会显著缩短P-loop与DFG基序的距离,与晶体结构特征高度吻合。
这些结果表明,Ligand-Transformer不仅能预测结合强度,还能解析配体诱导的构象调控机制,为理解药物作用模式提供了全新视角。
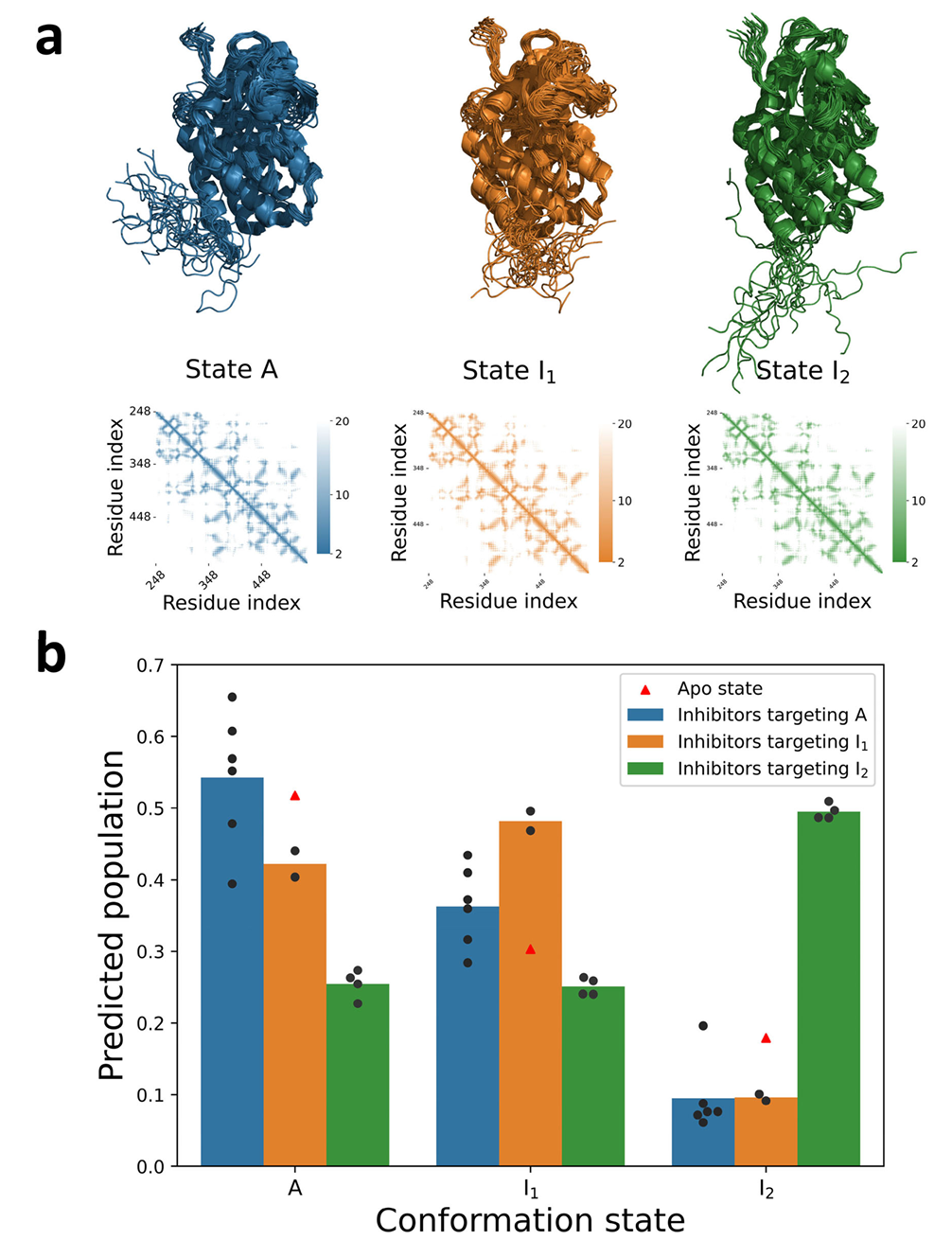
a 展示了 ABL 激酶 A、I₁、I₂三种状态(对应 PDB ID 6XR6、6XR7、6XRG)的平均距离图和结构集合;b 为不同抑制剂的构象选择性预测,12 种分子按文献分为三组,分组柱状图显示各组在对应状态下的预测种群显著更高,红色三角形代表 AlphaFold2 预测的 apo 状态构象种群,预测种群与实验结果高度一致。
a 展示了 ABL 激酶 A、I₁、I₂三种状态(对应 PDB ID 6XR6、6XR7、6XRG)的平均距离图和结构集合;b 为不同抑制剂的构象选择性预测,12 种分子按文献分为三组,分组柱状图显示各组在对应状态下的预测种群显著更高,红色三角形代表 AlphaFold2 预测的 apo 状态构象种群,预测种群与实验结果高度一致。
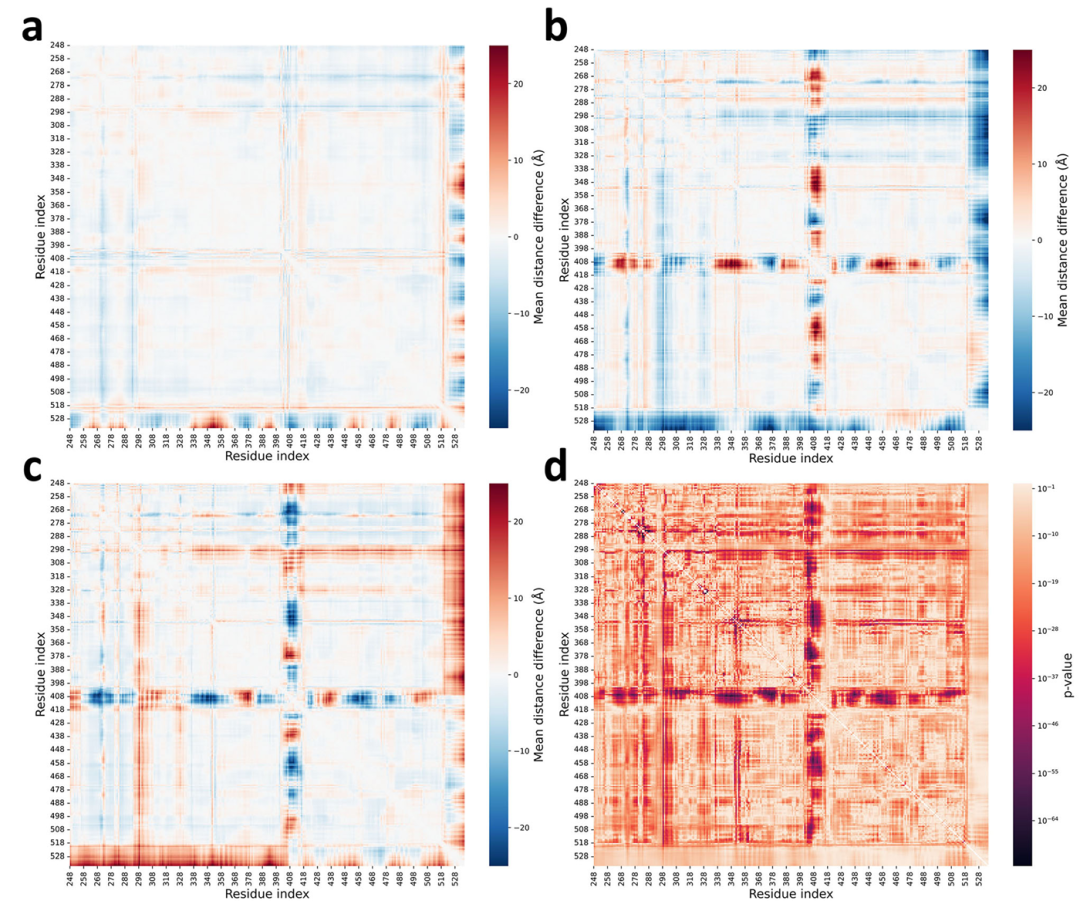
该图展示了 ABL 三种状态间的差异距离矩阵,a 为 I₁状态与 A 状态的差异,b 为 I₂状态与 A 状态的差异,c 为 I₁状态与 I₂状态的差异,同时包含单因素方差分析的 F 统计值,颜色强度表示残基对距离分布在不同状态间差异的显著性,为理解构象差异提供了量化依据。
该图展示了 ABL 三种状态间的差异距离矩阵,a 为 I₁状态与 A 状态的差异,b 为 I₂状态与 A 状态的差异,c 为 I₁状态与 I₂状态的差异,同时包含单因素方差分析的 F 统计值,颜色强度表示残基对距离分布在不同状态间差异的显著性,为理解构象差异提供了量化依据。
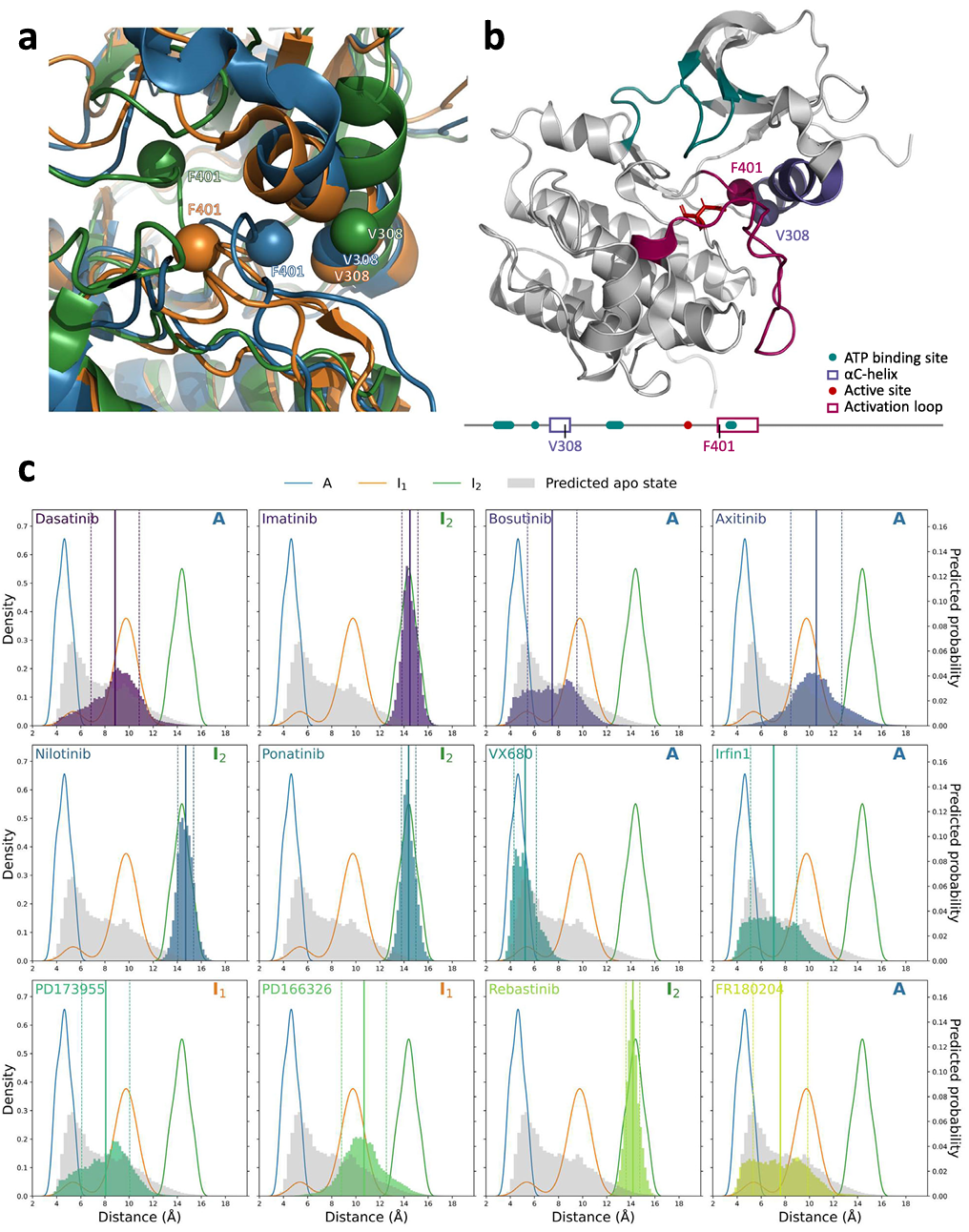
ABL 的 V308 和 F401 残基间距离分析。a 为 ABL 三种状态中 V308 和 F401 残基的空间距离叠加图;b 展示了 A 状态中这两个残基的空间位置及相关功能区域标注;c 为 12 种抑制剂结合时两残基的距离分布,包含三种状态的实测结构距离核密度估计、AlphaFold2 预测的 apo 状态距离分布,以及 Ligand-Transformer 的预测结果,体现了不同抑制剂对构象的诱导偏好。
ABL 的 V308 和 F401 残基间距离分析。a 为 ABL 三种状态中 V308 和 F401 残基的空间距离叠加图;b 展示了 A 状态中这两个残基的空间位置及相关功能区域标注;c 为 12 种抑制剂结合时两残基的距离分布,包含三种状态的实测结构距离核密度估计、AlphaFold2 预测的 apo 状态距离分布,以及 Ligand-Transformer 的预测结果,体现了不同抑制剂对构象的诱导偏好。
讨论与展望
Ligand-Transformer的突破性意义体现在三个维度:
- 1. 方法论创新:首次实现从序列-拓扑输入直接预测复合物构象空间,摆脱了对预处理结构的依赖,为缺乏结构信息的靶点提供了筛选可能。
- 2. 应用价值:在耐药突变体EGFR和构象动态丰富的ABL激酶上的成功应用,证实了其在难处理靶点中的实用价值,58%的实验验证命中率远超传统虚拟筛选方法。
- 3. 效率优势:两个数量级的速度提升使其能够应对大规模化合物库筛选,结合其开源特性(https://github.com/zshengyu14/LigandTransformer),有望成为学术界与工业界的标准化工具。
未来发展方向包括:拓展至蛋白-蛋白相互作用预测、整合自由能计算以提升亲和力预测精度、开发针对变构调节剂的专项优化模块等。可以预见,Ligand-Transformer将推动药物研发从"经验筛选"向"理性设计"的转型,为解决未满足的临床需求提供强大技术支撑。
(●'◡'●) 需要进一步讨论的同学欢迎留言交流!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-03,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

