Baishenglai 平台:AI 驱动的药物发现全流程解决方案
Baishenglai 平台:AI 驱动的药物发现全流程解决方案
用户1151118
发布于 2026-01-08 13:04:24
发布于 2026-01-08 13:04:24
Empowering Drug Discovery through Intelligent, Multitask Learning and OOD Generalization
期刊: arxiv 链接: https://www.arxiv.org/abs/2508.01195 简介: 本文提出了一个名为 Baishenglai(BSL)的深度学习增强型开放式虚拟药物发现平台,其创新点在于整合七大核心任务于统一模块化框架,并强调对分布外(OOD)分子结构的泛化能力评估。该平台结合生成模型、图神经网络等先进技术,针对分子生成与优化、药物-靶点相互作用预测等任务开发了多种创新方法,如 RMCD 分子生成方法、CLG-DTA 亲和力预测模型等。实验中,BSL 在多个基准数据集(如 QM7、KIBA、USPTO-50k 等)上均达到当前最优性能,且在 OOD 数据测试中表现优异,例如在血脑屏障通透性预测中对未见过的药物分类准确率达 100%;同时,其成功发现了 3 种对 GluN1/GluN3A NMDA 受体有明确生物活性的化合物。该论文表明,BSL 为虚拟药物发现提供了全面、可扩展且有效的解决方案,能加速生物医学研究和药物开发进程。

药物发现作为保障人类健康、应对重大疾病挑战的关键环节,其传统研发模式正面临严峻挑战——动辄 10-15 年的研发周期、数十亿美元的投入,以及居高不下的临床试验失败率,这些问题严重制约着新药研发的效率与进程。近年来,人工智能凭借高效的数据处理与表征能力,在生物信息学和药理学关键任务中展现出显著优势,然而现有计算平台大多仅覆盖部分核心任务,导致工作流程碎片化、效率低下,且在算法创新和分布外(OOD)数据泛化能力上存在不足,极大地阻碍了药物发现的进程。在此背景下,武汉大学团队开发的 Baishenglai(BSL)平台应运而生,作为一个深度学习增强的开放式虚拟药物发现平台,它为解决上述难题提供了全新的思路与方案。
现有药物发现平台的局限性剖析
随着计算技术的进步,众多与药物相关的虚拟筛选平台应运而生,这些平台大致可分为两类。一类是基于模拟的平台,如 Schrödinger,它提供了分子建模、结合亲和力预测和基于结构的药物设计等综合工具;OpenVS 则能在本地模拟药物-蛋白质相互作用,为特定研究环境的部署提供了灵活性。另一类是基于深度学习的平台,如 iDrug,它整合了多种预测方法,以满足更广泛的筛选需求,在药物发现中具有更强的通用性和适应性。这些平台通过快速识别有前景的候选药物、降低实验成本、提高虚拟筛选和预测工作流程的效率与准确性,显著加速了早期药物开发。
但在实际应用中,这些平台仍存在诸多局限性。以 DrugFlow 为例,它对 OOD 数据的支持有限,筛选任务仅局限于预设的 4317 个分子库和特定的靶口袋,难以适应开放式药物设计场景。而且,许多平台依赖固定的数据集进行模型训练,忽视了模型对 OOD 数据的泛化能力,当遇到结构新颖或临床未见的化合物时,模型性能会大幅下降,降低预测的准确性和可靠性。更严重的是,当前平台在任务完整性方面存在明显缺陷,尽管部分平台能完成基本的属性预测、分子生成和基于靶点的药物筛选任务,但缺乏药物-细胞响应预测、药物逆合成和药物-药物相互作用预测等任务的支持算法和数据,难以提供系统性解决方案。此外,多个平台间的功能碎片化和数据结构标准不一致,常常需要研究者手动转换数据格式和整合结果,显著增加了技术壁垒和协作成本。部分平台在自定义数据上传、使用收费和代码闭源等方面也存在限制,进一步提高了使用门槛。这些局限性对现代药物发现产生了切实影响,减缓了从靶点识别到先导化合物选择的进程,导致资源利用效率低下,阻碍了人工智能辅助设计的实际部署。因此,迫切需要一个具有强大集成性、泛化性和任务可扩展性的端到端药物设计平台,以满足现实生物医学挑战的需求。
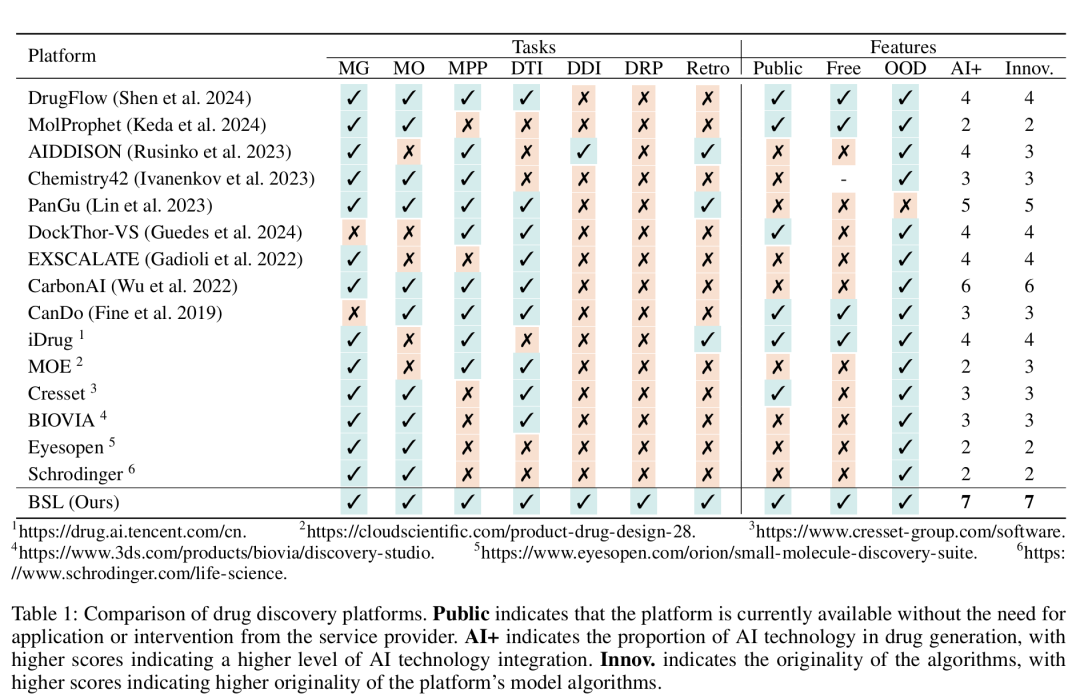
该表从任务覆盖和平台特性两个维度,将 BSL 与 DrugFlow、MolProphet、Schrödinger 等现有平台进行对比。任务覆盖方面,BSL 支持 7 项核心任务,而其他平台均存在任务缺失;平台特性方面,BSL 在公开性(无需申请即可使用)、AI 技术集成度、算法创新性等指标上均得分为 7(最高),且支持自定义数据上传,优于多数闭源、收费或功能受限的平台,突出了 BSL 的全面性和开放性。
该表从任务覆盖和平台特性两个维度,将 BSL 与 DrugFlow、MolProphet、Schrödinger 等现有平台进行对比。任务覆盖方面,BSL 支持 7 项核心任务,而其他平台均存在任务缺失;平台特性方面,BSL 在公开性(无需申请即可使用)、AI 技术集成度、算法创新性等指标上均得分为 7(最高),且支持自定义数据上传,优于多数闭源、收费或功能受限的平台,突出了 BSL 的全面性和开放性。
BSL 平台的核心架构与技术创新
BSL 平台作为一个全面的药物发现虚拟筛选平台,其核心设计围绕七大核心任务展开,包括分子条件生成与优化、药物-靶点亲和力预测、药物-细胞响应预测、药物-药物相互作用预测、属性预测和合成路径预测,并配备了 12 种不同的深度学习增强方法。该平台广泛融合了零样本学习、领域适应、扩散模型、图神经网络和对比学习等先进技术,奠定了坚实的技术基础。
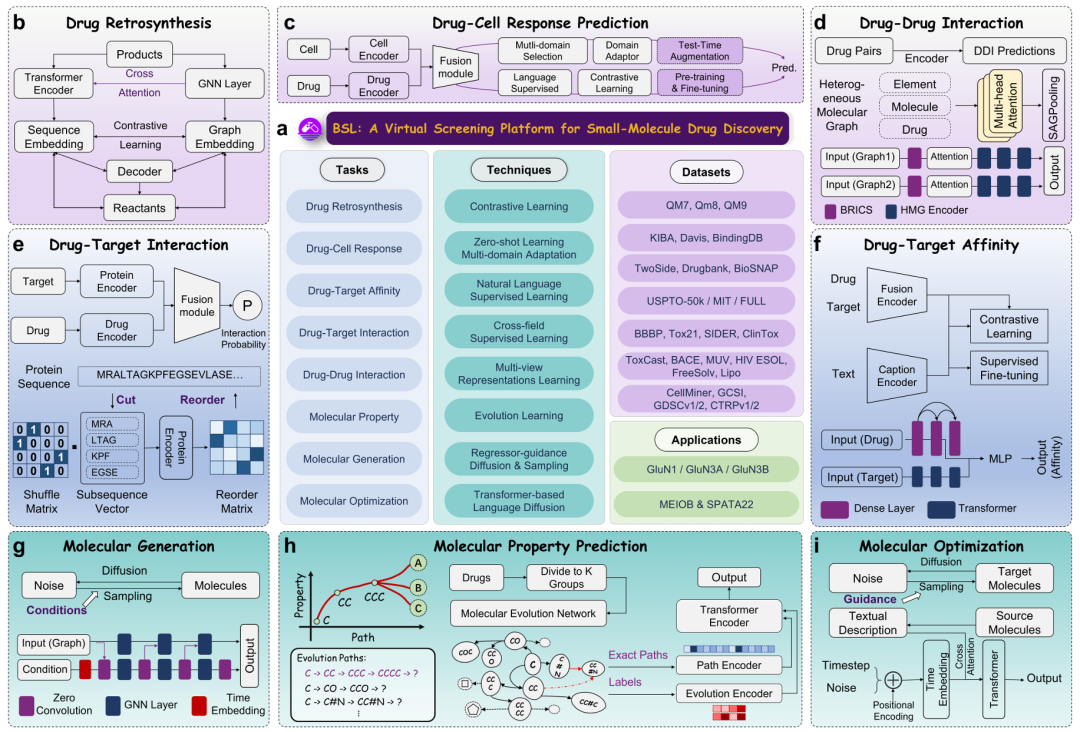
该图展示了 BSL 平台的整体架构,包括其支持的七大核心任务(分子生成与优化、药物逆合成、药物 - 细胞响应预测、药物 - 药物相互作用预测等)、底层技术(如扩散模型、图神经网络、对比学习等)、基准数据集(如 QM7、KIBA、USPTO-50k 等)以及下游应用。图中 b-i 分别呈现了每个特定任务的技术工作流程,例如分子生成采用扩散模型结合梯度控制,药物 - 靶点亲和力预测使用跨领域信息融合策略,直观体现了平台的模块化设计和多任务整合能力。
该图展示了 BSL 平台的整体架构,包括其支持的七大核心任务(分子生成与优化、药物逆合成、药物 - 细胞响应预测、药物 - 药物相互作用预测等)、底层技术(如扩散模型、图神经网络、对比学习等)、基准数据集(如 QM7、KIBA、USPTO-50k 等)以及下游应用。图中 b-i 分别呈现了每个特定任务的技术工作流程,例如分子生成采用扩散模型结合梯度控制,药物 - 靶点亲和力预测使用跨领域信息融合策略,直观体现了平台的模块化设计和多任务整合能力。
在分子生成任务中,传统基于条件的分子生成方法在确保采样空间有效性方面存在局限,导致生成大量无效分子。为解决这一问题,BSL 平台引入了 RMCD 方法,它将扩散模型的分数估计与基于靶细胞系和 IC50 分数的回归控制器模型的梯度相结合;同时,还引入了名为 DDIDiff 的新型协作药物设计方法,该方法利用已知的药物 - 药物相互作用(DDI)信息作为条件输入,引导扩散过程,从而生成具有潜在协同效应的新候选分子。
分子优化任务对于可控设计和微调治疗化合物至关重要。BSL 平台整合了专注于条件和多属性分子优化的新方法,如 FMOP,这是一种基于表型药物发现的片段掩码分子优化方法,它利用无回归扩散模型对分子的掩码区域进行条件优化,无需训练即可有效生成具有相似骨架的新分子;TransDLM 则使用 IUPAC 名称作为语义表示,并将属性要求编码为文本,减少了扩散过程中的误差传播。
分子属性预测(MPP)旨在估计决定药物潜力的关键化学和生物学属性,但现有方法往往缺乏有效利用属性间相关性或整合外部药理学知识的机制。为此,BSL 平台融入了两种新方法,MEvoN 是一种分子进化网络,通过将分子的结构信息与其进化背景相结合来创建统一的分子表示;KCHML 则通过将分子图编码为分子、元素和药理学三个视图的异质结构,整合外部知识以获得更丰富的表示,从而推进分子属性预测。
药物 - 靶点相互作用(DTI)和药物-靶点亲和力(DTA)预测任务通常分别被表述为分类和回归问题,然而现有方法普遍局限于分布内预测,且在遇到未见过的药物或靶点时,往往未能考虑 OOD 泛化。BSL 平台整合了两种先进方法分别处理这两项任务,有效解决了 OOD 问题。Siam-DTI 采用双通道网络结构进行跨领域监督学习,并采用跨领域信息融合策略捕捉蛋白质的局部和全局信息;CLG-DTA 引入了常识性数值知识图谱(CNKG),利用线性结构准确直观地表示数值及其相互关系。
药物响应预测(DRP)任务旨在预测不同化合物在生物环境中的表现,以便优先选择药物候选者。但许多现有方法在提取复杂分子结构方面存在局限,且在处理 OOD 样本时能力有限,无法解决泛化挑战。为应对 OOD 问题,BSL 平台整合了三种创新方法,TransEDRP 利用双 transformer 架构,整合边缘嵌入以有效捕捉药物分子的药理化学特性和细胞系的基因组序列;MSDA 引入了专为临床前药物筛选设计的零样本学习范式,通过设计多源域选择器从训练数据集中选择与目标域相似的多个药物域;CLDR 采用带有自然语言监督的对比学习模型,将回归标签转换为文本,并整合常识性数值知识图谱。
药物-药物相互作用预测旨在识别两种药物之间的潜在相互作用,传统的分子表示方法往往无法捕捉多粒度知识,且难以处理复杂药物相互作用中存在的分子异质性。BSL 平台融入的 KCHML 方法,首先为每种药物创建综合的个体表示,结合其结构、化学和药理学信息,然后使用神经网络对这些药物对之间的复杂相互作用进行建模,有效利用预训练过程中获得的知识准确识别潜在的 DDI。
逆合成预测以产物分子为输入,输出该产物可能的反应物。现有方法在推理过程中往往未能严格遵循分子序列规则,可能导致结构在化学上不准确。为深入整合多模态信息并提高预测的准确性和有效性,BSL 平台融入了 CFC-Retro 这一无模板逆合成预测模型,它通过带有跨模态注意力的双分支编码器整合序列和图表示,跨模态细粒度对比学习策略在不同模态和反应前后状态下对齐未改变子结构的原子级表示,无需手工对齐或数据约束。
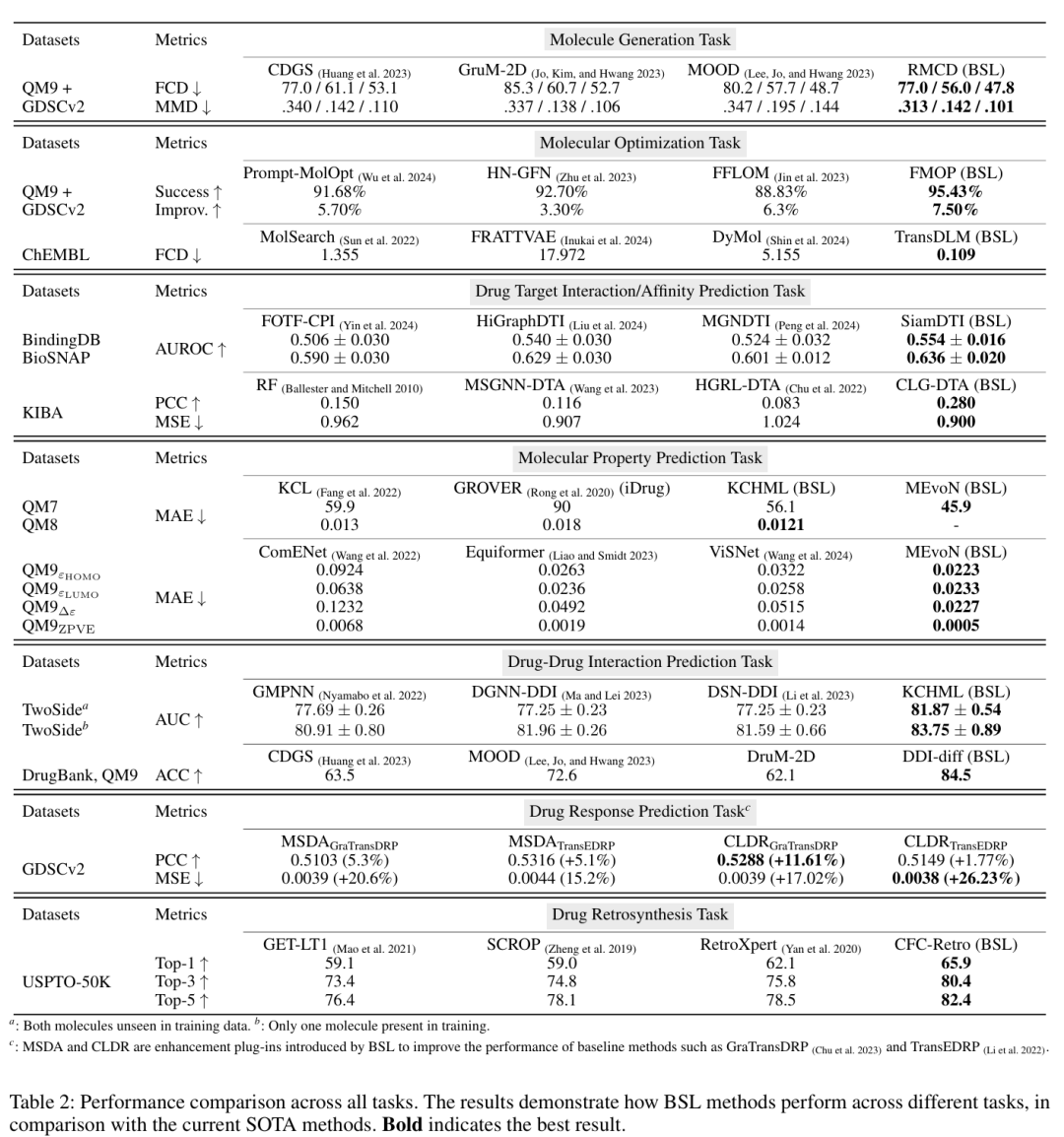
该表展示了 BSL 方法与当前最优模型在七大任务上的性能数据。例如,分子生成任务中 BSL 的 FCD 值更低(0.109),分子优化任务的成功率更高(95.43%);药物 - 靶点亲和力预测中,CLG-DTA 的 PCC 达 0.280,显著优于传统模型;药物 - 药物相互作用预测中,KCHML 的 AUC 达 81.87±0.54。所有任务中 BSL 方法的关键指标均为最优(加粗显示),验证了其在各领域的领先性能。
该表展示了 BSL 方法与当前最优模型在七大任务上的性能数据。例如,分子生成任务中 BSL 的 FCD 值更低(0.109),分子优化任务的成功率更高(95.43%);药物 - 靶点亲和力预测中,CLG-DTA 的 PCC 达 0.280,显著优于传统模型;药物 - 药物相互作用预测中,KCHML 的 AUC 达 81.87±0.54。所有任务中 BSL 方法的关键指标均为最优(加粗显示),验证了其在各领域的领先性能。
BSL 平台的性能评估与实际应用
为评估 BSL 平台的性能,研究团队进行了全面的基准测试研究。在平台层面,将 BSL 与现有可用平台进行了比较;在任务层面,使用标准化数据集和广泛采用的评估指标,在七个代表性任务上与最先进的模型进行了基准测试。
对于分子生成(MG)任务,采用了 Fréchet ChemNet 距离(FCD)和最大均值差异(MMD)进行评估;对于分子优化(MO)任务,使用成功率来衡量至少能生成一种改进分子的细胞系比例;对于分类和回归任务,使用皮尔逊相关系数(PCC)和平均绝对误差(MAE)等标准指标,以确保跨任务评估的一致性和公平性。所有指标均遵循原始论文中描述的标准定义和计算协议,详细信息见补充材料。
从评估结果来看,BSL 在多个任务上始终优于专门的基线模型,展现出较高的预测准确性和快速的推理能力。这些基线代表了过去三年为单个任务开发的最先进模型。据研究团队所知,BSL 是迄今为止公开可用的端到端平台中任务覆盖最广、预测准确性最高的平台,它的发布有望在药物研究中提供更高效、更易获取的人工智能驱动解决方案。
在实际应用中,针对与中风和阿尔茨海默病等神经系统疾病相关的 GluN1/GluN3A 亚型 NMDA 受体,由于缺乏其结构信息,传统方法难以奏效。研究团队利用 BSL 平台进行基于序列的虚拟筛选,绕过了对解析晶体结构的需求。该工作流程的输入包括 GluN1/GluN3A 受体的氨基酸序列和大规模小分子化合物库,这些数据通过 BSL 平台进行处理,平台利用 CLG-DTA 预测化合物-靶点结合亲和力并相应地优先选择候选化合物,然后对排名靠前的分子进行体外电生理测定验证,最终成功识别出三种在体外电生理实验中对 GluN1/GluN3A 受体表现出明确生物活性的新型化合物。这些发现凸显了 BSL 在结构信息有限或不可得的挑战性环境中促进药物发现的有效性。
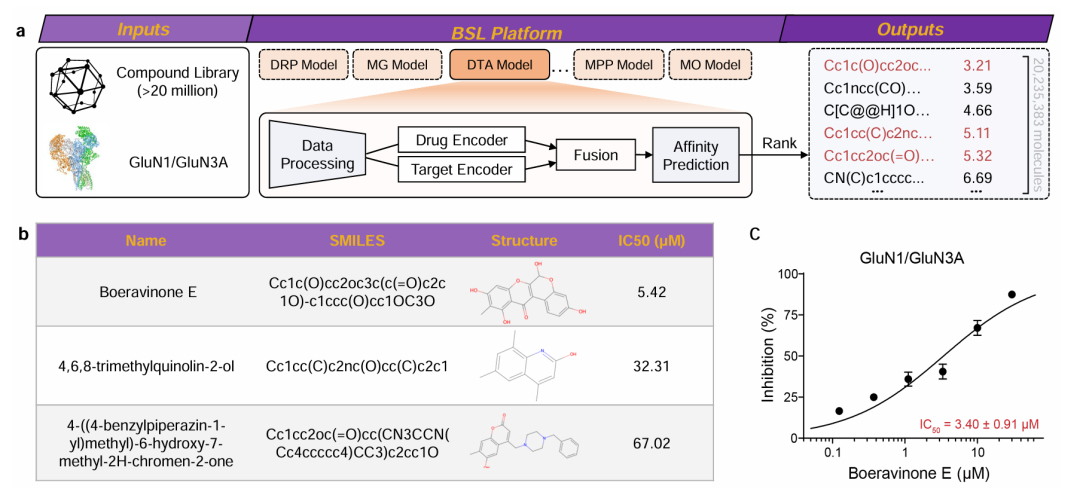
该图分为两部分:a 展示了 BSL 平台利用 AI 驱动的 CLG-DTA 模型进行 GluN1/GluN3A 受体药物筛选的工作流程,包括输入化合物库与受体序列、通过药物编码器和靶点编码器处理数据、融合预测亲和力并排序候选化合物;b 列出了筛选出的 3 种具有生物活性的化合物,包括其名称、SMILES 结构和 IC50 值(如 Boeravinone E 的 IC50 为 5.42μM),验证了平台在缺乏晶体结构时的实际筛选能力。
该图分为两部分:a 展示了 BSL 平台利用 AI 驱动的 CLG-DTA 模型进行 GluN1/GluN3A 受体药物筛选的工作流程,包括输入化合物库与受体序列、通过药物编码器和靶点编码器处理数据、融合预测亲和力并排序候选化合物;b 列出了筛选出的 3 种具有生物活性的化合物,包括其名称、SMILES 结构和 IC50 值(如 Boeravinone E 的 IC50 为 5.42μM),验证了平台在缺乏晶体结构时的实际筛选能力。
此外,为评估 MPP 模型的 OOD 性能,研究团队使用 BSL 和 iDrug 平台对四种未出现在公共数据集上的药物进行了血脑屏障通透性(BBBP)预测。结果显示,BSL 正确预测了所有药物的分类,而 iDrug 平台的准确率为 75%,且 BSL 的预测概率得分通常更接近真实标签,表现出更高的置信度,验证了 BSL 在分子属性预测任务中的高准确性和可靠性。
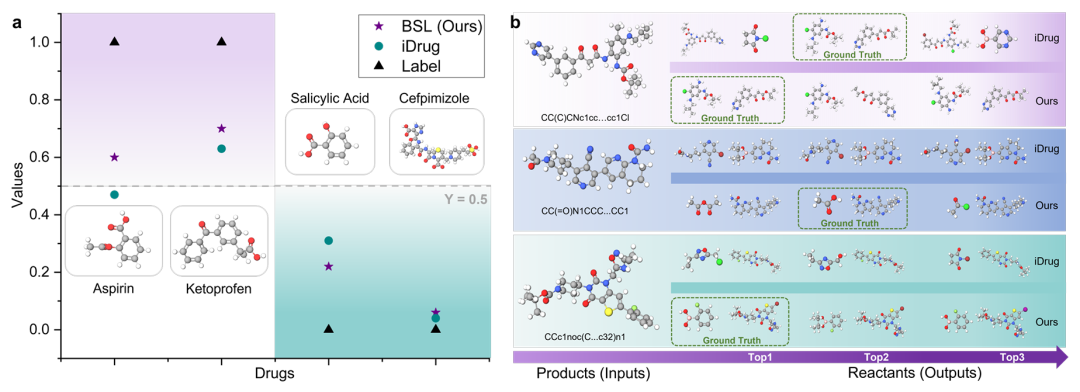
该图包含两个子图:a 对比了 BSL 和 iDrug 在 4 种未见于训练集的药物的血脑屏障通透性(BBBP)预测结果,BSL 正确分类所有药物,预测概率更接近真实标签,而 iDrug 准确率为 75%,体现 BSL 在分子属性预测任务中的 OOD 泛化优势;b 展示了两者在逆合成预测中的表现,BSL 能将 3 种复杂化合物的真实反应物排在前 2 位(其中 2 次为第 1 位),而 iDrug 仅成功 1 次,凸显 BSL 在复杂分子处理中的稳健性。
该图包含两个子图:a 对比了 BSL 和 iDrug 在 4 种未见于训练集的药物的血脑屏障通透性(BBBP)预测结果,BSL 正确分类所有药物,预测概率更接近真实标签,而 iDrug 准确率为 75%,体现 BSL 在分子属性预测任务中的 OOD 泛化优势;b 展示了两者在逆合成预测中的表现,BSL 能将 3 种复杂化合物的真实反应物排在前 2 位(其中 2 次为第 1 位),而 iDrug 仅成功 1 次,凸显 BSL 在复杂分子处理中的稳健性。
在逆合成模型的实际性能评估中,研究团队对三种结构多样的目标化合物进行了案例研究。结果表明,该模型准确识别了所有三个案例的真实反应物,并将它们排在前 2 位预测中,其中两次排在第 1 位,而 iDrug 平台仅在一个案例中成功,在另外两个案例中失败,这些结果凸显了该模型在应用于复杂化合物时的卓越稳健性。
总结与展望
BSL 平台作为一款专注于药物发现的全面虚拟筛选工具,其目标是成为药物研究的重要资源,帮助科学家更快、更准确地识别和开发药物。该平台的核心功能涵盖了药物开发的各个阶段,包括药物设计、属性预测和虚拟筛选。凭借高精度的预测模型、出色的零样本泛化能力以及对多模态输入的支持,BSL 平台有效应对了药物开发中的各种挑战。总体而言,BSL 为研究人员提供了强大、易用的工具,极大地提高了药物发现的效率,推动了药物开发的进程。
未来,随着技术的不断进步和研究的深入,BSL 平台有望在以下方面得到进一步发展。在算法方面,持续优化现有的深度学习模型,提高其在复杂场景下的预测精度和泛化能力;在功能方面,不断拓展新的任务类型,以覆盖药物研发的更多环节;在数据方面,整合更多高质量、多维度的生物医学数据,为模型训练和预测提供更坚实的基础。相信 BSL 平台将在加速药物发现、推动生物医药领域发展方面发挥越来越重要的作用。
如需了解更多关于 BSL 平台的详细信息,可访问其官方网站:https://www.baishenglai.net(小编在写本文的时候似乎还访问不了)。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-06,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

