Science | BioEmu:AI突破分子动力学极限 × Baker团队又一新作:RosettaFold-3
Science | BioEmu:AI突破分子动力学极限 × Baker团队又一新作:RosettaFold-3
用户1151118
发布于 2026-01-08 13:07:58
发布于 2026-01-08 13:07:58
今天想推荐的文章太多了,直接开启速递模式!😄
一句话总结
- 1. 提出生成式深度学习系统BioEmu,实现蛋白质平衡构象集合的高效模拟,在精度和速度上显著超越传统MD模拟,为基因组尺度生物分子功能预测和药物发现提供新路径。
- 2. 构建通用生物分子数据框架AtomWorks及其基础上的RosettaFold-3模型,显著提升跨任务性能并缩小与闭源AlphaFold3的差距,加速开源生物分子建模研发
- 3. 开发AdaptiveFlow平台,通过主动学习与并行化计算实现690亿分子级超大规模虚拟筛选,发现并结构验证纳摩尔级FSP1抑制剂
- 4. 基于3766个实验验证结合剂的元分析提出界面指标ipSAE_min及组合特征筛选策略,实现从头设计结合剂的靶标无关优先级排序
- 5. 系统基准测试25种分子嵌入模型,发现多数神经模型不优于ECFP指纹,唯有CLAMP模型表现显著更佳
- 6. 提出深度学习增强的结构基础虚拟筛选平台HelixVS,在提升筛选精度的同时将速度提高近15倍、成本降至每千分子1元,并验证多款新活性化合物
- 7. 开发Applm框架,利用超大蛋白质语言模型及泛化导向评估体系,在真实场景中过敏原预测性能显著优于现有方法
- 8. 提供贝叶斯优化在生物过程工程中的实用指南,结合案例和进阶方法降低非统计背景研究者的应用门槛
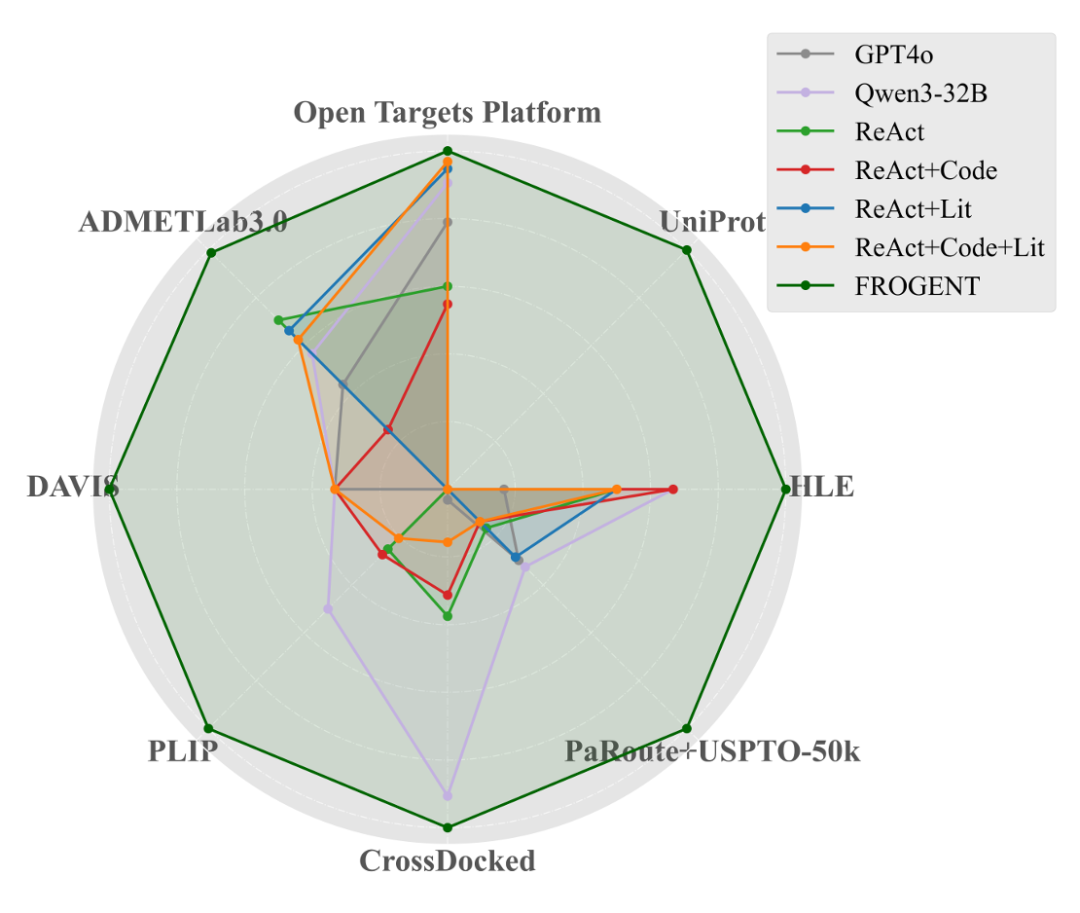
- 9. 实现首个小分子药物设计全流程自动化智能体Frogent,整合多层工具与模型显著提升药物研发效率
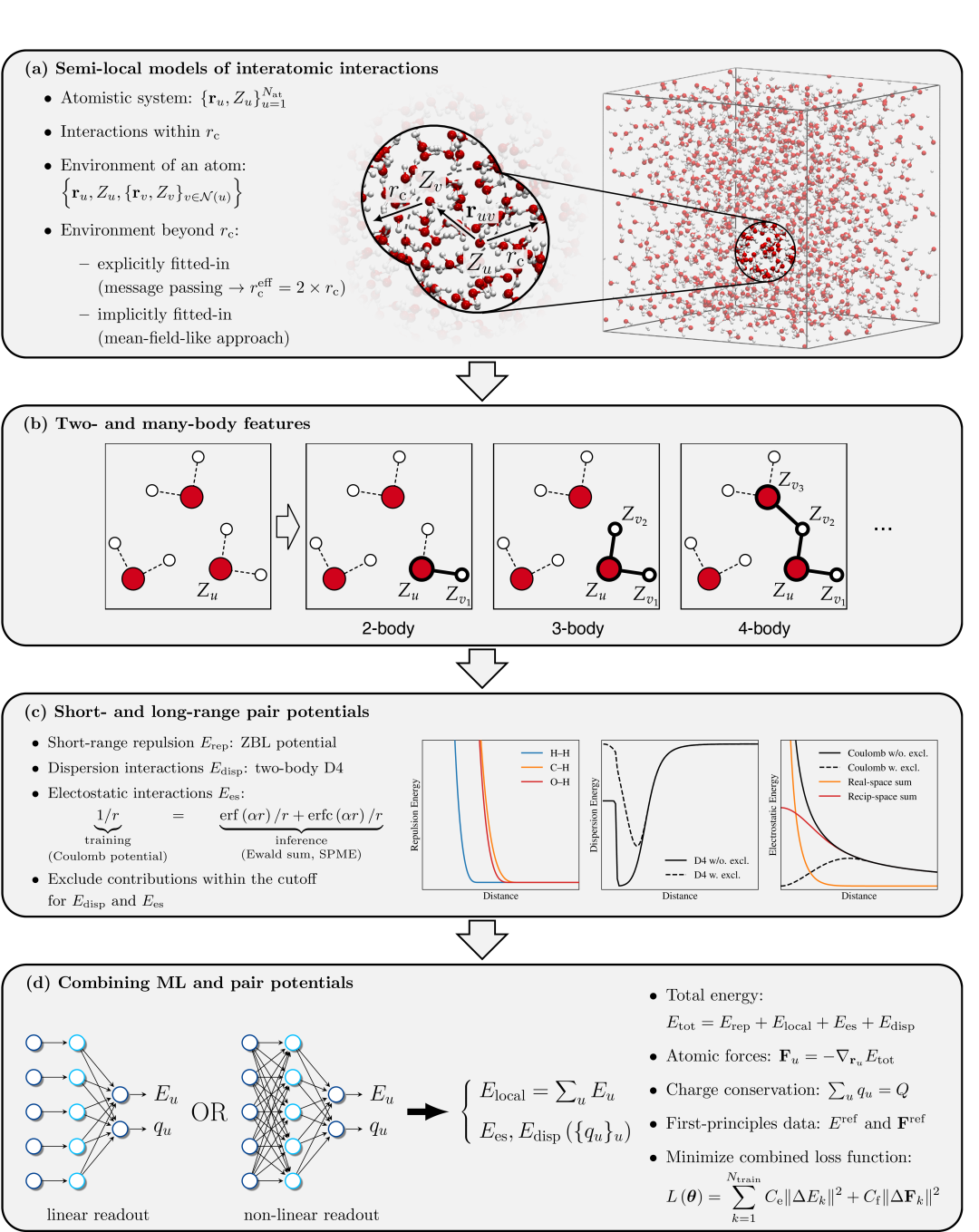
- 10. 评估显式长程作用的通用机器学习势在生物分子模拟中的表现,揭示数据构成与评估方法对性能的限制
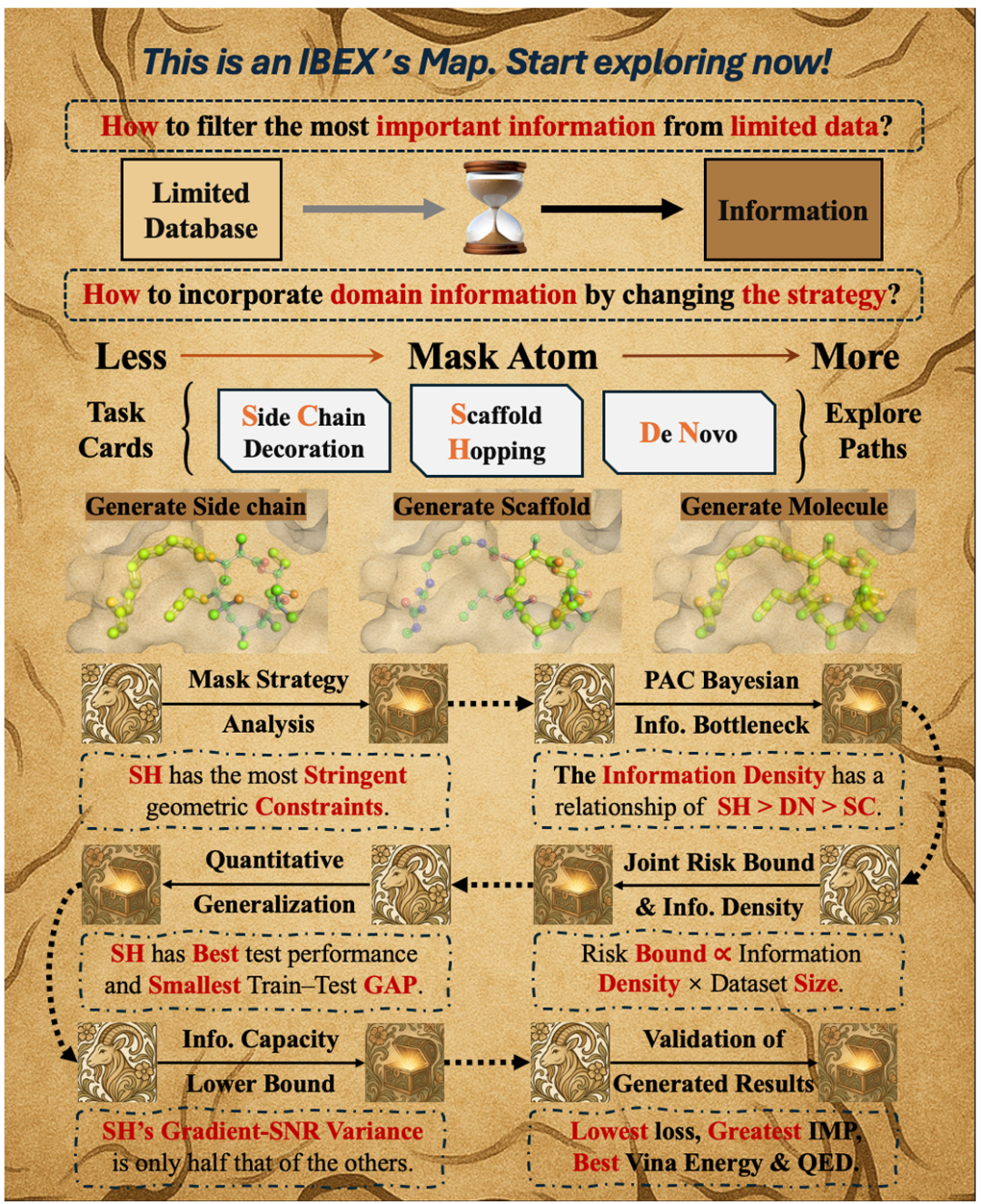
- 11. 提出基于信息瓶颈理论的粗到精分子生成框架IBEX,在数据稀缺场景下显著提升分子生成的对接成功率、能量和多样性
1. Scalable Emulation of Protein Equilibrium Ensembles with Generative Deep Learning
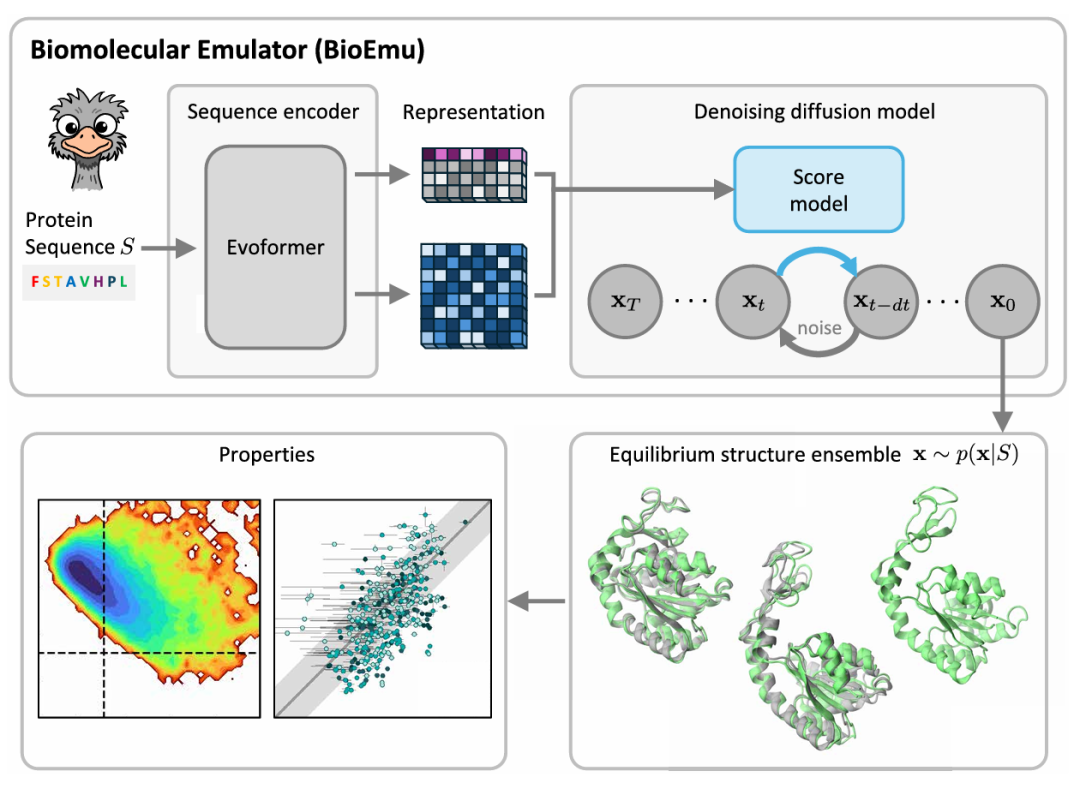
期刊: Science 链接: https://doi.org/10.1126/science.adv9817 代码: BioEmu 模型及推理代码: https://github.com/microsoft/bioemu 基准测试代码:https://github.com/microsoft/bioemu-benchmarks 模型测试(ColabFold): https://github.com/sokrypton/ColabFold 简介: 该论文提出生物分子模拟器 BioEmu,一种生成式深度学习系统,可高效模拟蛋白质平衡构象集合,解决现有生物物理实验和分子动力学(MD)模拟吞吐量低的问题。其创新点在于结合 AlphaFold 的序列编码与扩散模型,经三阶段训练(AlphaFold 数据库预训练、超 200 毫秒 MD 模拟数据训练、50 万 + 实验蛋白质稳定性数据微调)实现高效采样。实验中,BioEmu 在预测已知构象变化(成功率 55%-90%)、模拟 MD 平衡分布(自由能误差 <1 kcal/mol,速度提升 4-5 个数量级)、预测蛋白质稳定性(误差 < 1 kcal/mol,相关系数> 0.6)等任务表现优异。该研究证明 MD 和实验数据的前期成本可通过深度学习摊销,为基因组尺度预测生物分子功能提供可行路径,且 BioEmu 可辅助 MD 工作流、解读实验机制、助力药物发现等。
2. Accelerating Biomolecular Modeling with AtomWorks and RF3
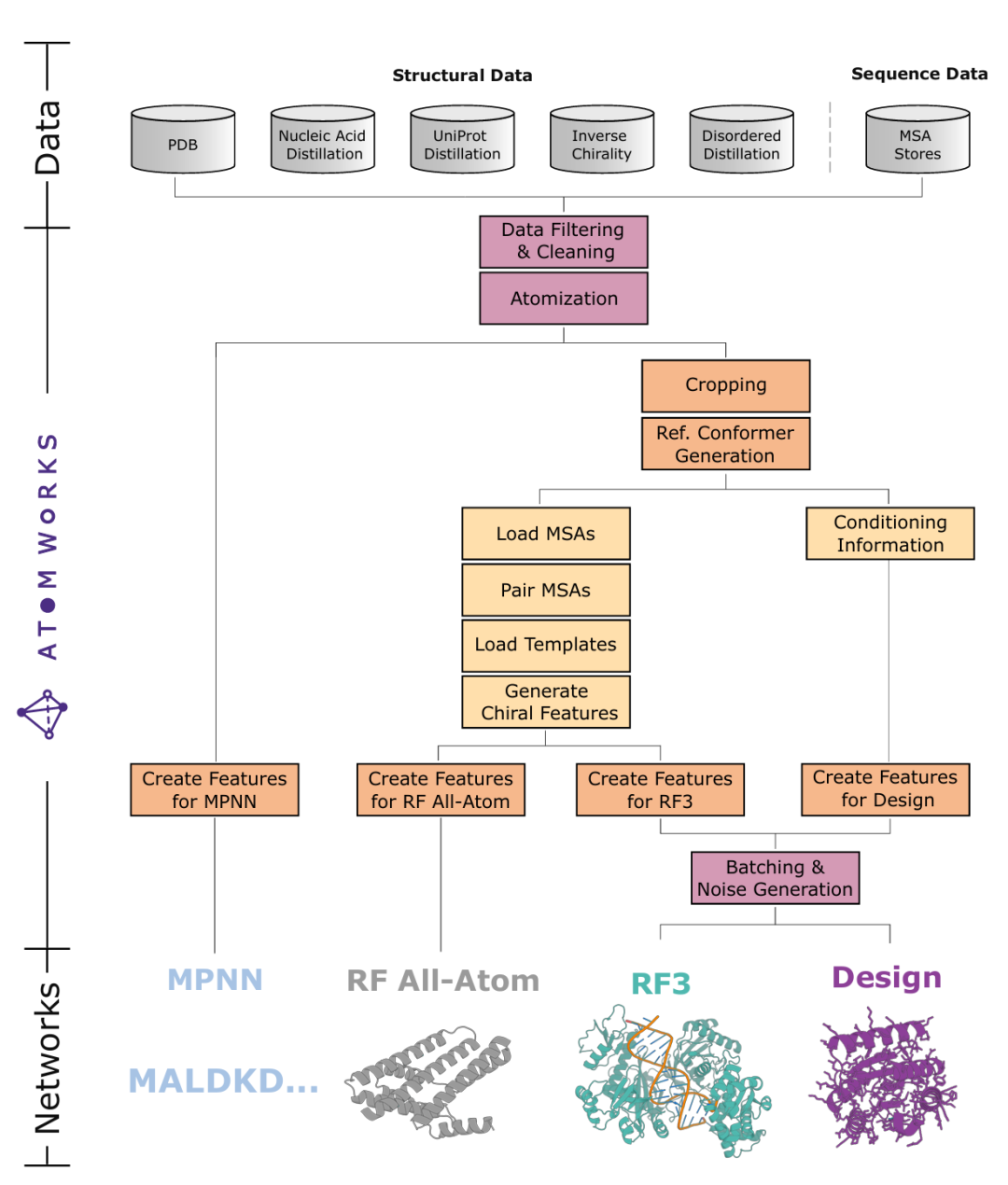
期刊: bioRxiv 链接: https://doi.org/10.1101/2025.08.14.670328 代码: AtomWorks框架:https://github.com/RosettaCommons/atomworks RF3的代码及模型权重: https://github.com/RosettaCommons/modelforge 简介: 该论文的要点是提出AtomWorks通用数据框架以简化生物分子基础模型开发,创新点在于其模块化设计支持多任务(结构预测、生成式蛋白质设计等)且提升数据质量,同时训练的RosettaFold-3(RF3) 通过优化手性处理缩小了开源模型与闭源AlphaFold3(AF3)的性能差距。方法上,AtomWorks以原子级结构表示为核心,标准化数据处理流程,支持多模型共享组件;RF3基于AtomWorks,整合新型蒸馏数据集(如RNA蒸馏集、核酸复合物蒸馏集),通过手性特征嵌入和原子级条件控制提升预测精度。实验中,RF3对配体手性中心预测正确率达88%(优于AF3的84%),混合L/D肽预测 backbone RMSD均值1.74 Å,且在蛋白质-蛋白质、蛋白质-配体等相互作用预测中性能介于AF3与开源Boltz之间;AtomWorks使超80%代码可跨模型共享,大幅提升研发效率。该论文发布的开源框架和模型为生物分子建模领域提供高效工具,加速下一代开源生物分子机器学习模型的开发与应用。
3. Ai-Enhanced Adaptive Virtual Screening Platform Enabling Exploration of 69 Billion Molecules Discovers Structurally Validated FSP1 Inhibitors
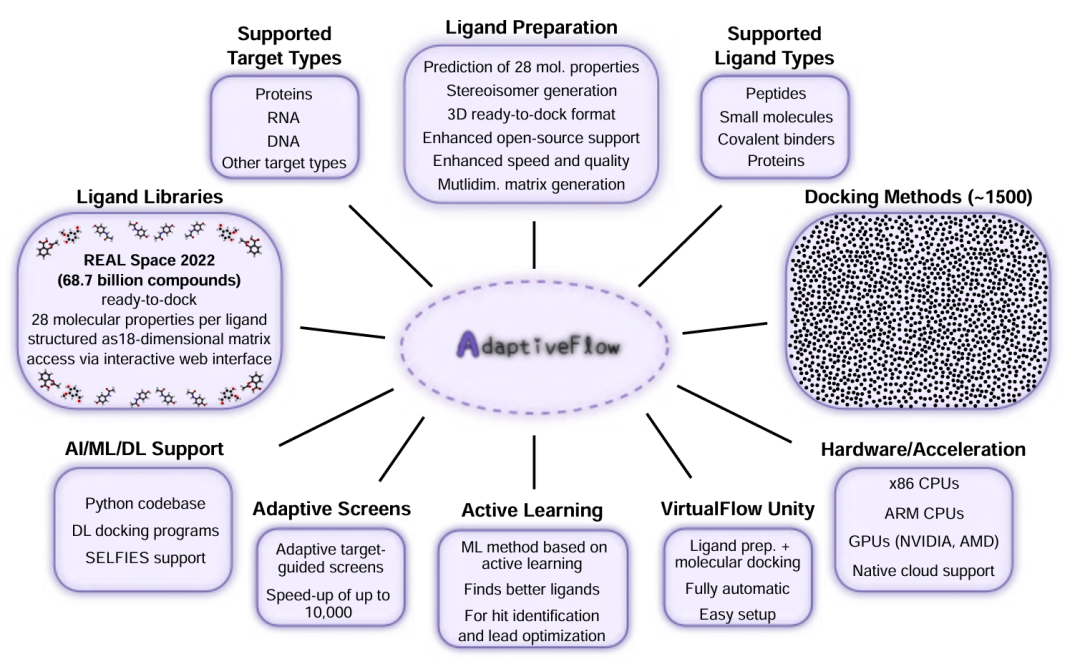
期刊: bioRxiv 链接: https://doi.org/10.1101/2023.04.25.537981 代码: AFLP模块:https://github.com/LigandUniverse/AFLP AFVS模块:https://github.com/LigandUniverse/VFVS Unity模块:https://github.com/LigandUniverse/AFU Unity Parallelized模块: https://github.com/LigandUniverse/AFUparr 简介: 该论文提出开源平台AdaptiveFlow,其要点是通过18维分子属性网格和可选主动学习组件实现超大规模虚拟筛选(ULVS)的高效化,创新点在于将计算成本降低约1000倍,且支持超1500种对接方法,在AWS云环境中实现近线性扩展至560万个虚拟CPU。方法上,AdaptiveFlow包含三个核心模块(AFLP用于配体制备、AFVS用于虚拟筛选、AFU整合工作流),基于Enamine REAL Space(690亿个可对接分子库),通过Adaptive Target-Guided Virtual Screens(ATG-VSs)先筛选代表性分子确定优质化学空间,再进行重点筛选。实验中,针对FSP1和PARP1两个疾病相关靶点,利用该平台筛选出纳摩尔级抑制剂,其中FSP1抑制剂经晶体结构验证结合模式,PARP1抑制剂(如iParp1)IC₅₀低至8.8 nM,与临床药物奥拉帕利相当;基准测试显示ATG-VS在筛选10万个分子时,对接分数与传统筛选100万个分子相当。该论文证明AdaptiveFlow能以空前规模和速度发现并优化药物候选分子,为早期药物研发提供高效工具。
4. Predicting Experimental Success in De Novo Binder Design: A Meta-Analysis of 3,766 Experimentally Characterised Binders
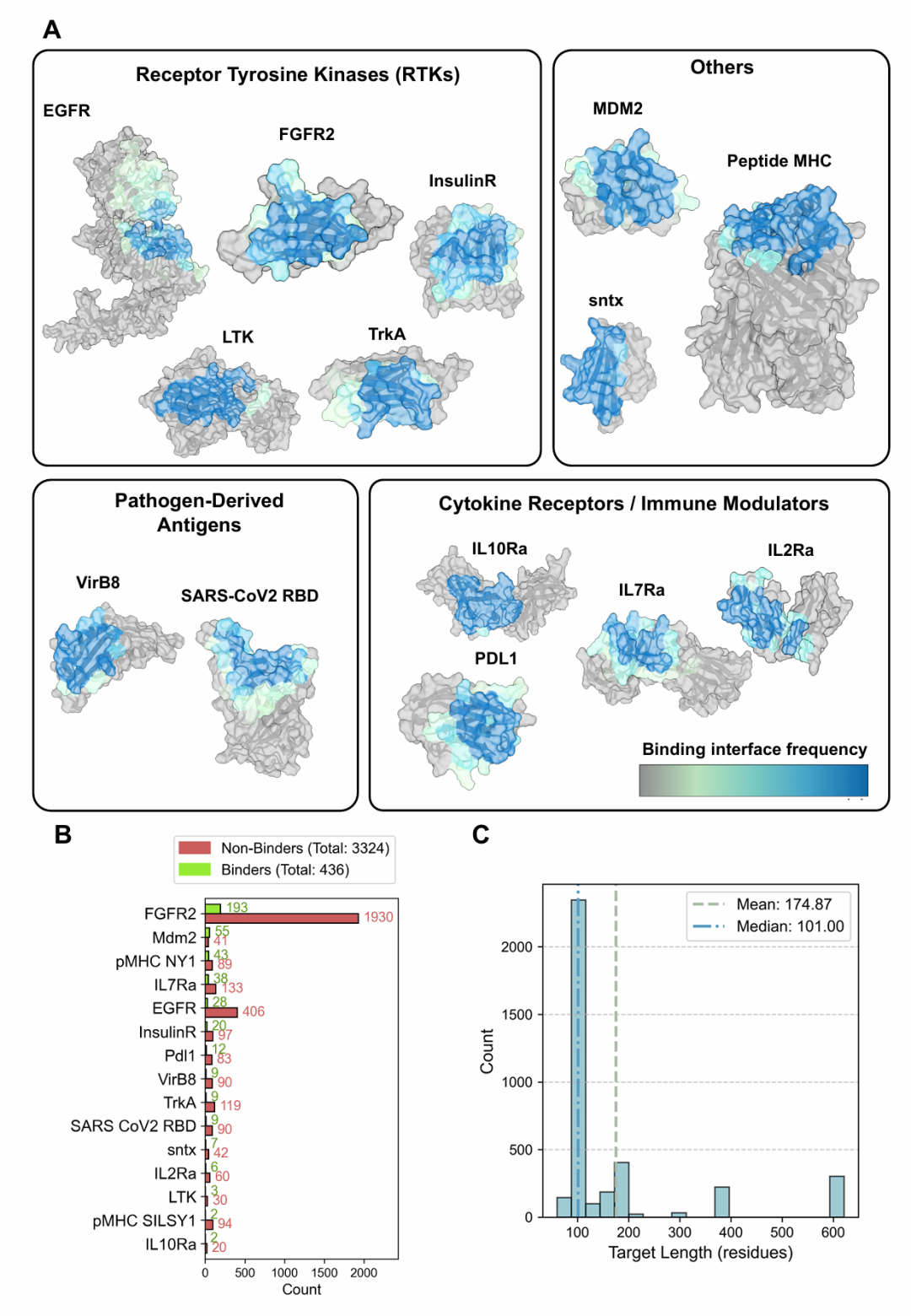
期刊: bioRxiv 链接: https://doi.org/10.1101/2025.08.14.670059 代码: (发表后公布) 简介: 该论文的要点是通过大规模元分析确定能预测从头设计蛋白质结合剂实验成功的关键指标,创新点在于发现AF3衍生的界面聚焦指标ipSAE_min预测性能优于常用的ipAE和ipTM,且结合物理化学界面描述符可进一步提升性能。方法上,研究构建了包含3766个经实验验证的结合剂(覆盖15个不同靶点)的数据集,用AF2(初始猜测和ColabFold版本)、AF3和Boltz-1四种结构预测工具重新预测每个结合剂-靶点复合物,提取200多个结构、能量和置信度特征,通过平均精度(AP)评估特征区分结合剂与非结合剂的能力,并采用贪心特征选择结合逻辑回归构建预测模型。实验中,AF3的ipSAE_min平均精度较ipAE提升1.4倍,结合ipSAE_min与Rosetta ∆G/∆SASA等特征后预测性能进一步优化;简单线性模型基于少量AF3特征即可跨数据集良好泛化,且提出的ipSAE_min排序结合结构过滤的策略能提升筛选精度。该论文为从头结合剂设计的体外优先级排序提供了可解释、靶标无关的筛选策略,同时发布的完整数据集为社区提供了基准资源,助力相关领域研究加速。
5. Benchmarking Pretrained Molecular Embedding Models For Molecular Representation Learning
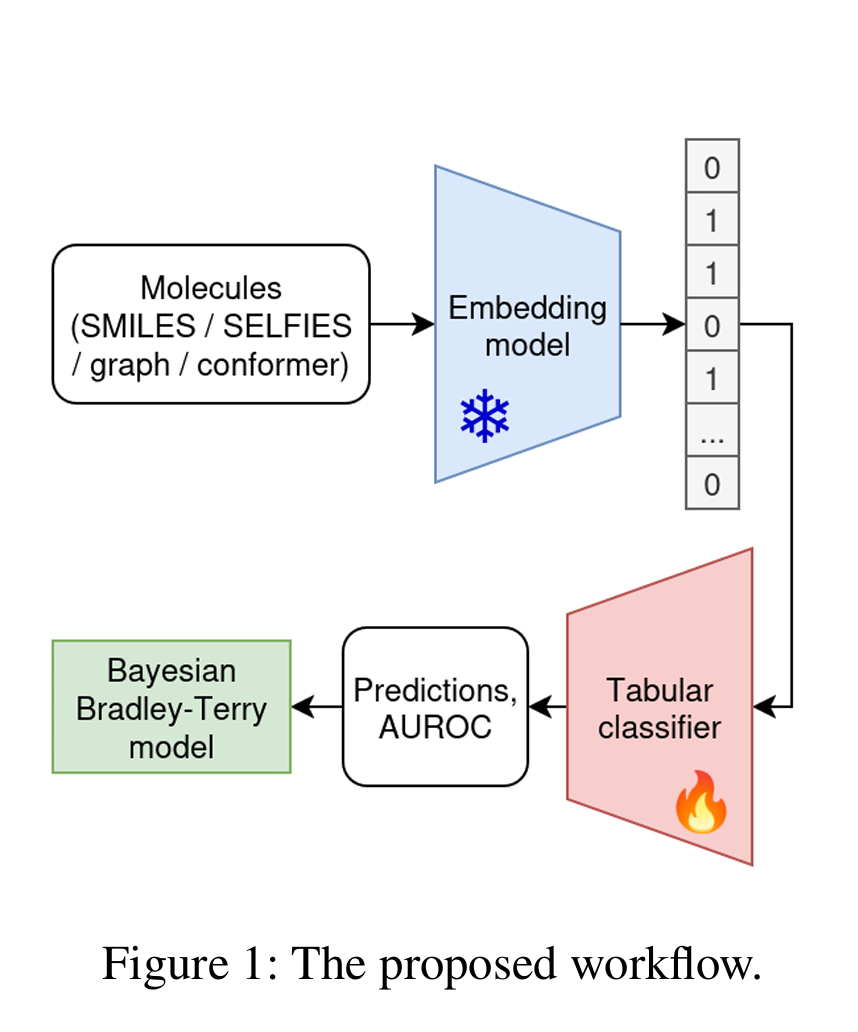
期刊: arXiv 链接: https://arxiv.org/abs/2508.06199 简介: 该论文的要点是对预训练分子嵌入模型进行大规模基准测试,创新点在于采用公平对比框架和分层贝叶斯Bradley-Terry模型,发现多数神经模型相较ECFP分子指纹无显著优势,仅基于分子指纹的CLAMP模型表现更优。方法上,研究评估25个涵盖不同模态、架构和预训练策略的模型,在25个数据集(7个来自MoleculeNet、18个来自TDC)上提取冻结嵌入,训练随机森林、逻辑回归和k近邻分类器,以AUROC为指标,用ECFP计数指纹作基线,通过贝叶斯测试分析模型性能。实验中,CLAMP模型平均AUROC达82.55%,是唯一统计上显著优于ECFP的模型;多数图神经网络(GNN)和SELFIES基于的文本Transformer表现较差,仅少数Transformer(如R-MAT、MolBERT)表现接近ECFP;在25个数据集中,5个数据集无模型优于ECFP,8个数据集仅1-2个模型优于ECFP。该论文为分子表示学习领域提供了严谨的基准测试结果,揭示现有预训练模型的局限,为从业者选择模型提供实用建议,也为后续模型开发指明需融入领域知识的方向。
6. Helixvs: Deep Learning Enhanced Structure-Based Virtual Screening Platform For Hit Discovery
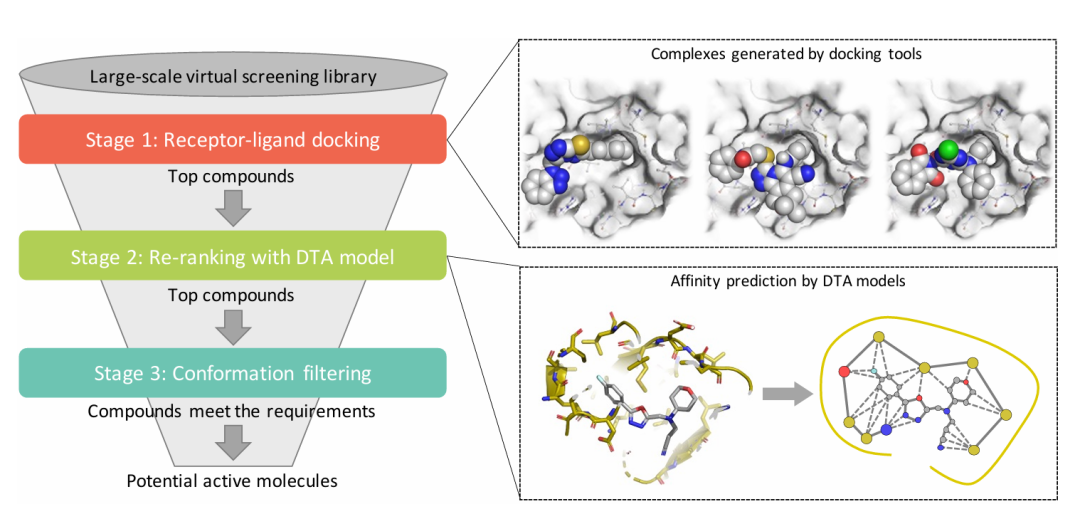
期刊: arXiv 链接: https://arxiv.org/abs/2508.10262 简介: 该论文的要点是提出深度学习增强的基于结构的虚拟筛选平台HelixVS,创新点在于将精确的深度学习姿态评分模型与姿态筛选模块整合到多阶段筛选流程中,在提升筛选性能的同时大幅加快速度并降低成本。方法上,HelixVS采用三阶段筛选流程:第一阶段用AutoDock QuickVina 2进行分子对接并保留多构象;第二阶段通过基于RTMscore改进的深度学习亲和力评分模型重新排序;第三阶段可选构象筛选并聚类确保结果多样性,同时依托百度云CPU和CHPC高性能计算资源实现高效筛选。实验中,在DUD-E数据集(含102个蛋白靶点)上,HelixVS的0.1%和1%富集因子(EF)分别达44.205和26.968,较Vina平均多发现159%活性分子且速度快近15倍,成本低至每千分子1元;在四个药物研发管线(CDK4/6、TLR4/MD-2、cGAS、NIK)中,筛选出的化合物经湿实验验证,超10%在微摩尔甚至纳摩尔水平显活性。该论文开发的HelixVS平台为药物发现提供高效、低成本的虚拟筛选工具,其公开在线版本和私有部署方案可满足不同用户需求,助力加速药物研发进程。
7. Driving Accurate Allergen Prediction with Protein Language Models and Generalization-Focused Evaluation
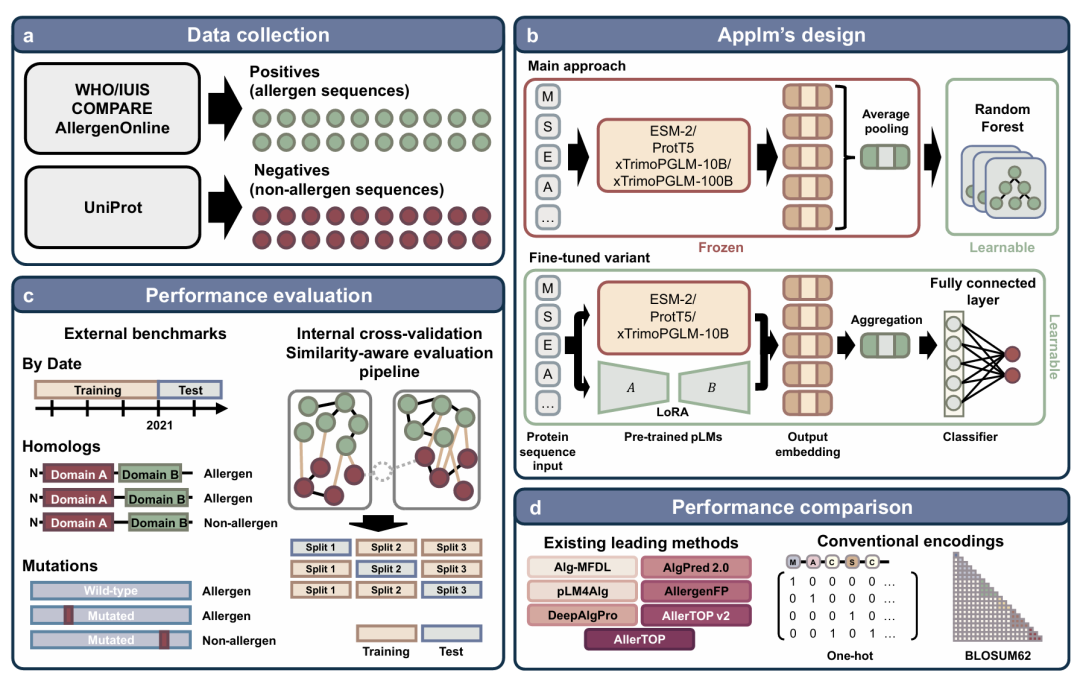
期刊: arXiv 链接: https://arxiv.org/abs/2508.10541 代码: https://github.com/brianwongsh/Applm 简介: 该论文提出了计算框架Applm,其创新点在于首次将1000亿参数的蛋白质语言模型xTrimoPGLM应用于过敏原预测,并构建了注重泛化能力的评估体系,以解决现有方法在真实场景中性能不佳的问题。方法上,Applm采用两阶段流程,先通过xTrimoPGLM、ESM-2等四种预训练蛋白质语言模型生成蛋白质的冻结嵌入,再输入随机森林分类器进行过敏原预测,同时开发了相似性感知评估流水线控制训练集与测试集(组间)、过敏原与非过敏原(类间)的序列相似性。实验中,在六个外部测试集(涵盖“按时间”“同源蛋白”“突变”三种真实场景)上,Applm对比七种主流方法,平均AUROC达0.872、平均AUPRC达0.700,显著优于竞品,且xTrimoPGLM-100B因参数规模大表现最佳;消融实验显示蛋白质语言模型嵌入比传统编码(如OHE、BLOSUM62)更具优势,训练集难度匹配、序列长度分布平衡等因素也影响模型性能。该论文不仅建立了过敏原预测的新基准,还提供了开源软件和基准数据集,其相似性感知评估方法也可应用于其他蛋白质分类任务。
8. A Guide to Bayesian Optimization in Bioprocess Engineering
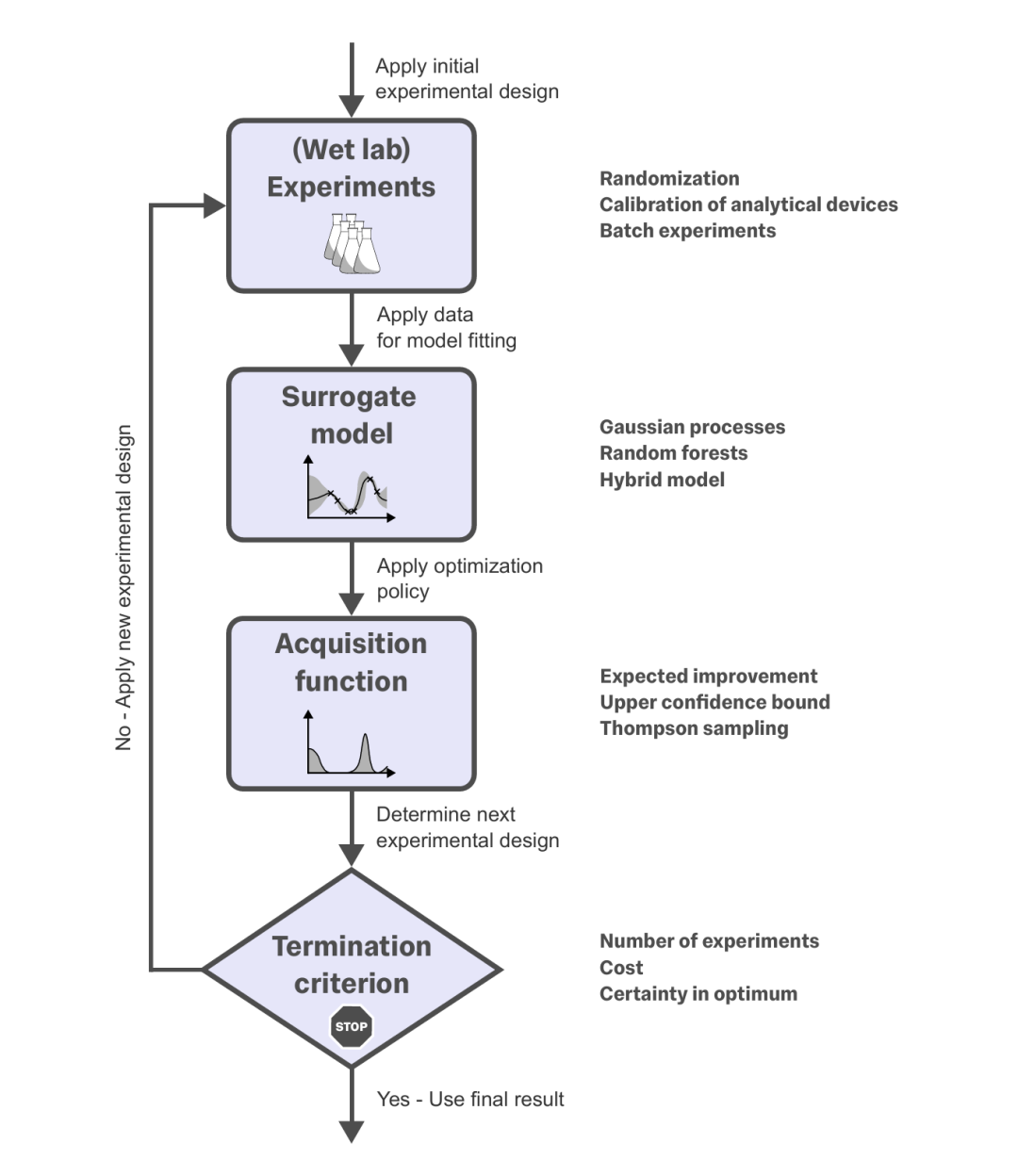
期刊: arXiv 链接: https://arxiv.org/abs/2508.10642 代码: https://github.com/lhelleckes/BO_Empirical_Examples 简介: 该论文聚焦贝叶斯优化(BO)在生物过程工程中的应用,创新点在于针对生物系统实验的复杂性(如高不确定性、系统偏差),提供了直观实用的BO方法介绍,并指出该领域未被充分探索的应用方向与算法挑战,降低了非统计背景研究者的使用门槛。方法上,BO以概率模型(常用高斯过程)为代理模型,结合采集函数(如期望改进EI、上置信界UCB、汤普森采样)平衡探索与利用,通过迭代选择实验条件,同时介绍了批量BO、多保真度BO等进阶方法以适配生物过程需求。实验中,以生物催化中粗细胞提取物反应速率优化(pH为变量)为例,展示了BO的实施流程,对比朴素高斯过程与融合反应动力学的混合模型,证明混合模型能更好处理批次效应等偏差;还通过模拟案例验证了超参数先验、核函数选择对模型性能的影响。该论文为生物过程工程师提供了BO实施的实用指南,也为机器学习研究者指出了生物过程工程中的特定挑战,助力BO成为该领域实验设计的标准工具。
9. FROGENT: An End-to-End Full-process Drug Design Agent
期刊: arXiv 链接: https://arxiv.org/abs/2508.10760v1
简介: 该论文提出了端到端全流程药物设计智能体Frogent,创新点在于通过大语言模型(LLM)和模型上下文协议(MCP),整合动态生化数据库、可扩展工具库与任务专用AI模型,解决了现有药物发现工具碎片化、需人工衔接的问题。方法上,Frogent包含数据库层(如PubMed、RCSB PDB、DrugBank等)、工具层(如RDKit、QVina、代码解释器等)和模型层(如P2Rank用于结合位点发现、ADMET-ai用于ADMET预测、多种扩散模型用于分子生成、DirectMultiStep用于逆合成),通过MCP实现各组件标准化通信与灵活扩展。实验中,在八个涵盖药物发现全流程的基准任务(如知识检索、性质预测、虚拟筛选、分子设计等)上,Frogent对比GPT-4o、Qwen3-32B及四种ReAct风格基线模型,在命中发现任务上性能为最佳基线的3倍,在相互作用分析任务上为2倍,且在逆合成规划等复杂任务中表现突出;案例研究中,成功完成充血性心力衰竭相关药物(以PPARγ为靶点)的端到端设计及碳酸酐酶II抑制剂的先导优化。该论文首次实现小分子药物设计的全流程自动化,显著提升药物研发效率,为后续结合湿实验验证奠定基础。
10. Performance of universal machine-learned potentials with explicit long-range interactions in biomolecular simulations
期刊: arXiv 链接: https://arxiv.org/abs/2508.10841v1 代码: https://github.com/nec-research/ictp 简介: 该论文系统评估了带有和不带有显式长程色散与静电作用的等变消息传递架构(以ICTP模型为代表)在生物分子模拟中的性能,创新点在于探究模型大小、训练数据构成和静电处理对模拟结果的影响,填补了通用机器学习势在生物分子模拟应用中的研究空白。研究采用在扩展的SPICE-v2数据集上训练的不同大小ICTP模型,结合解析对势(如D4色散校正、库仑势)处理长程相互作用,并通过均方根误差(RMSE)等指标评估模型在分布内/外基准数据集及多种生物相关系统(体相液态水、NaCl水溶液、丙氨酸三肽、Trp-cage迷你蛋白、Crambin蛋白)中的表现。实验发现,更大模型虽提升基准数据集精度,但未持续改善模拟属性;训练数据构成影响预测结果,长程静电作用对多数系统无系统性影响,仅对Trp-cage提升构象变异性。该论文表明,当前不平衡的数据集和不成熟的评估方法限制了通用机器学习势在生物分子模拟中的应用,为后续模型优化和数据构建提供了方向。
11. IBEX: Information-Bottleneck-EXplored Coarse-to-Fine Molecular Generation under Limited Data
期刊: arXiv 链接: https://arxiv.org/abs/2508.10775v1 简介: 该论文提出IBEX框架,旨在解决基于结构的药物设计中蛋白质-配体复合物数据稀缺的问题,创新点在于利用PAC贝叶斯信息瓶颈理论量化样本信息密度,发现骨架跳跃(SH)任务比从头生成(DN)和侧链修饰(SC)任务具有更高信息密度和更好迁移性能,且采用粗到精的生成-物理优化范式提升分子生成质量。研究保留TargetDiff架构和超参数进行粗生成,再通过L-BFGS优化器,基于五个物理项和六个自由度对分子构象精细优化。实验在CBGBench基准上进行,使用CrossDocked2020数据集,结果显示IBEX将零样本对接成功率从53%提升至64%,平均Vina分数从-7.41 kcal/mol提升至-8.07 kcal/mol,在100个口袋中57个实现最佳中位Vina能量,同时QED提升25%,有效性和多样性达当前最佳,且显著降低外推误差。该论文为数据稀缺场景下的结构基药物设计提供了理论与实践基础,证明信息瓶颈理论与物理优化结合可高效提升分子生成性能。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-16,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

