J. Cheminf. | PoLiGenX:精准控制的AI配体生成新范式,潜空间条件扩散驱动的先导化合物优化
J. Cheminf. | PoLiGenX:精准控制的AI配体生成新范式,潜空间条件扩散驱动的先导化合物优化
用户1151118
发布于 2026-01-08 13:32:57
发布于 2026-01-08 13:32:57
在AI药物发现的浪潮中,如何在保证配体与蛋白结合能力的同时,兼顾形状相似性与化学多样性,一直是先导化合物优化的核心挑战。来自辉瑞机器学习研究团队的最新研究 PoLiGenX,通过潜空间条件扩散技术,创造性地将参考分子的结构信息注入生成过程,实现了对配体生成的精准控制。实验表明,该方法生成的配体不仅保持了与参考分子高达 0.87 的形状相似度,还展现出更优的结合姿态、更低的应变能和更好的成药性,为靶向药物设计开辟了新路径。

一、背景:先导化合物优化的困境与机遇
现有方法的局限
近年来,基于结构的药物发现(SBDD)与深度学习的结合取得了显著进展。TargetDiff、DiffSBDD、EQGAT-diff 等等变扩散模型通过对蛋白口袋的条件约束,能够从头生成具有高结合亲和力的配体分子。这些方法在技术上确实令人印象深刻——它们利用 E(3) 等变图神经网络处理三维坐标,通过去噪扩散概率模型(DDPM)逐步雕刻出配体结构。
然而,这类无约束生成方法存在一个根本性矛盾:它们虽然能探索广阔的化学空间,却难以精准命中特定的设计目标。生成的分子往往缺乏对已知活性片段的借鉴,可能面临合成困难、成药性差、毒性风险等现实问题。更关键的是,在药物发现的先导化合物扩增(Hit Expansion)阶段,研究者需要的不是漫无目的的探索,而是基于已验证的苗头化合物进行有方向的优化——既要保留核心药效团的结构特征,又要通过化学修饰改善 ADMET 性质。
精准控制的必要性
设想这样一个场景:高通量筛选已经为你找到了一个具有中等活性的苗头化合物,它能与靶标蛋白结合,但存在溶解度差、代谢稳定性低等问题。传统的先导化合物优化依赖药物化学家的经验和直觉,通过手工设计侧链修饰来改进性质,这个过程往往耗时数月甚至数年。而现有的 AI 生成模型虽然能产出数千个全新分子,但它们与你的苗头化合物可能毫无关联——这种创造性过剩反而成了负担。
PoLiGenX 正是为解决这一痛点而生。它的核心理念是:让AI理解并延续参考分子的设计意图。通过将参考分子的结构和化学信息编码为潜变量,模型能够在保持形状相似性的前提下,探索周边的化学空间,生成既熟悉又新颖的候选分子。
二、方法:潜空间引导的精密生成机制
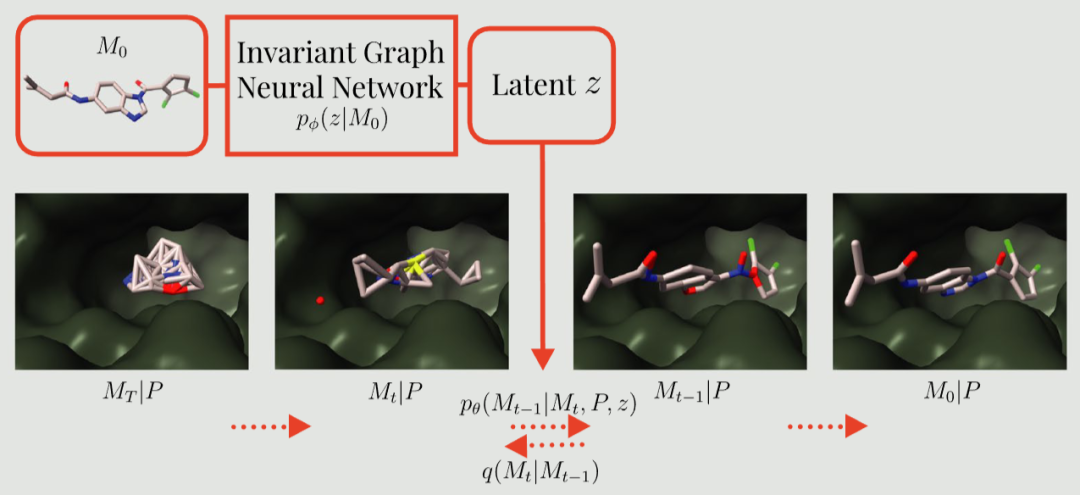
PoLiGenX 模型框架图 - 展示编码配体 z 如何作为输入引导扩散模型 pθ 生成新配体 M0
PoLiGenX 模型框架图 - 展示编码配体 z 如何作为输入引导扩散模型 pθ 生成新配体 M0
2.1 技术架构:三位一体的设计哲学
PoLiGenX 的技术创新可以概括为三个核心模块的协同工作:
模块一:不变图编码器(G-Invariant Encoder)
首先需要将参考配体 M₀ 的三维结构"压缩"成一个固定维度的潜向量 z ∈ R¹²⁸。这个编码器采用 EQGAT 消息传递层,经过 8 轮信息聚合后,通过 Softmax 注意力池化机制将所有原子的嵌入整合为全局表示。关键在于,这个编码器必须满足 SO(3) 等变性和排列不变性——无论分子如何旋转、平移或原子编号如何排列,输出的潜向量都应保持一致。
这个潜向量 z 不仅仅编码了分子的几何形状,还隐含了原子组成、化学键连接等丰富信息。可以理解为,z 是参考分子的"DNA指纹",浓缩了其结构精髓。
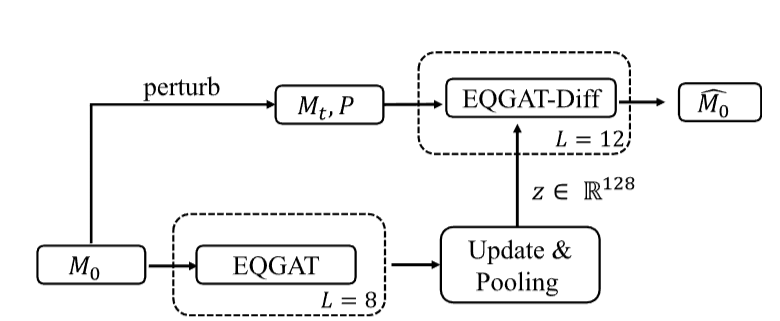
不变图编码器输入真实配体 M0 并输出潜嵌入 z,上方展示 EQGAT-diff 如何输入扰动配体 Mt、蛋白口袋及潜表示 z 来预测未损坏的配体 M0
不变图编码器输入真实配体 M0 并输出潜嵌入 z,上方展示 EQGAT-diff 如何输入扰动配体 Mt、蛋白口袋及潜表示 z 来预测未损坏的配体 M0
模块二:自适应层归一化(AdaLN)条件注入
仅仅得到潜向量还不够,关键是如何将其"注入"到扩散生成过程中。PoLiGenX 采用了受计算机视觉风格迁移启发的自适应层归一化技术。在 EQGAT-diff 的每一层消息传递之后,节点嵌入 H 不再使用固定的归一化参数,而是根据潜向量 z 动态计算仿射变换的尺度(scale)和偏移(shift)参数:
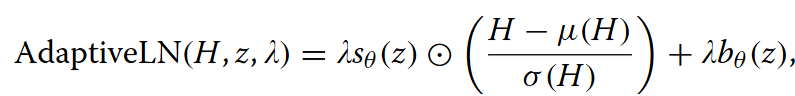
这里的 α ∈ (0,1] 是可调节的控制强度参数——α 接近 1 时,生成分子高度相似于参考分子;α 趋近 0 时,模型退化为无条件的 EQGAT-diff。这种设计赋予了研究者在相似性与多样性之间灵活权衡的能力。
模块三:联合训练的变分框架
PoLiGenX 采用变分推断框架,通过证据下界(ELBO)联合优化编码器 qφ(z|M₀) 和生成器 pθ(M|P,z)。损失函数包含三个部分:
- 重构损失:确保生成的配体能准确匹配原始数据的坐标、原子类型和化学键
- 潜空间正则化:使用最大均值差异(MMD)损失使潜向量分布接近标准正态分布
- 辅助预测任务:让编码器同时预测分子的原子数,增强潜表示的语义有效性
值得注意的是,PoLiGenX 对不同数据模态采用了自适应噪声调度器:坐标的噪声衰减系数设为 2.5,化学键为 1.5,原子类型为 1.0。这种差异化处理反映了一个深刻的化学直觉——分子的几何结构比离散的原子标签更需要精细控制。
2.2 与前人工作的本质区别
SQUID 和 ShapeMol 也尝试过基于形状的配体生成,但它们使用的是等变表面编码,且未将蛋白受体信息整合进生成过程。相比之下,PoLiGenX 的潜空间条件机制更加简洁高效,同时天然支持蛋白口袋的约束。
更重要的是,PoLiGenX 不仅保留了参考分子的形状,还通过联合训练确保潜变量捕获了化学相似性。实验表明,生成的配体与参考分子的 ECFP4 指纹 Tanimoto 相似度达到 0.33,这意味着在保持大致轮廓的同时,化学结构已发生了实质性的多样化变异——这正是先导化合物优化所追求的"熟悉中的创新"。
三、结果:全方位性能提升的实证
3.1 形状保留与化学探索的完美平衡
在 CrossDocked2020 数据集的 100 个蛋白-配体复合物测试中,PoLiGenX 展现出卓越的形状控制能力。平均形状相似度达到 0.87,远超无条件的 EQGAT-diff(0.64)。但更令人惊喜的是化学多样性的保持:平均化学相似度为 0.33,而 EQGAT-diff 仅为 0.12。
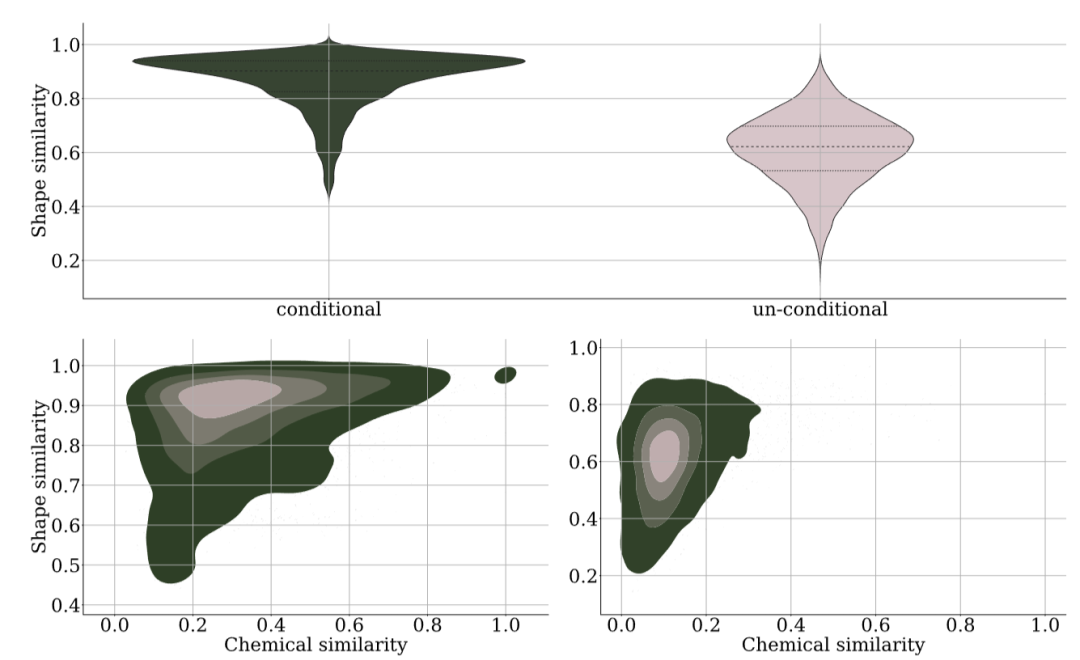
顶部展示 Tanimoto 形状相似度的小提琴图,对比 PoLiGenX(左)与 EQGAT-diff(右);底部为热力图直方图,展示形状相似度与化学相似度的联合分布,颜色越亮表示分子数量越多
顶部展示 Tanimoto 形状相似度的小提琴图,对比 PoLiGenX(左)与 EQGAT-diff(右);底部为热力图直方图,展示形状相似度与化学相似度的联合分布,颜色越亮表示分子数量越多
这组数据揭示了 PoLiGenX 的核心价值:既不是简单的分子复制,也不是盲目的随机探索,而是沿着参考分子的演化路径进行有方向的创新。从热力图可以看到,PoLiGenX 生成的分子集中在高形状相似度、中等化学相似度的区域,这正是药物化学家期望的骨架跃迁效果。
3.2 结合性能:更优的亲和力与构象质量
对接分析显示,PoLiGenX 生成的配体平均 QuickVina2 得分为 -6.88 ± 2.12 kcal/mol,Top 10% 样本更是达到 -7.77 ± 2.61 kcal/mol,优于参考数据集的 -6.85 ± 2.33 kcal/mol。但这只是表面的成功——真正的技术突破体现在配体姿态的质量上。
DiffSBDD 和 TargetDiff 虽然能生成"看起来合理"的配体,但它们的构象往往存在严重的物理缺陷——超高的应变能意味着分子内部存在扭曲的化学键,大量的立体冲突则表明原子间的空间排斥未得到妥善处理。这些问题在分子动力学模拟或实验验证中会暴露无遗。
相比之下,PoLiGenX 生成的配体具有接近天然配体的构象合理性。应变能降低了 3-4 倍,立体冲突接近参考水平,氢键恢复率接近 50%——这些指标共同表明,潜空间条件引导确实帮助模型学习到了更符合物理化学规律的生成策略。
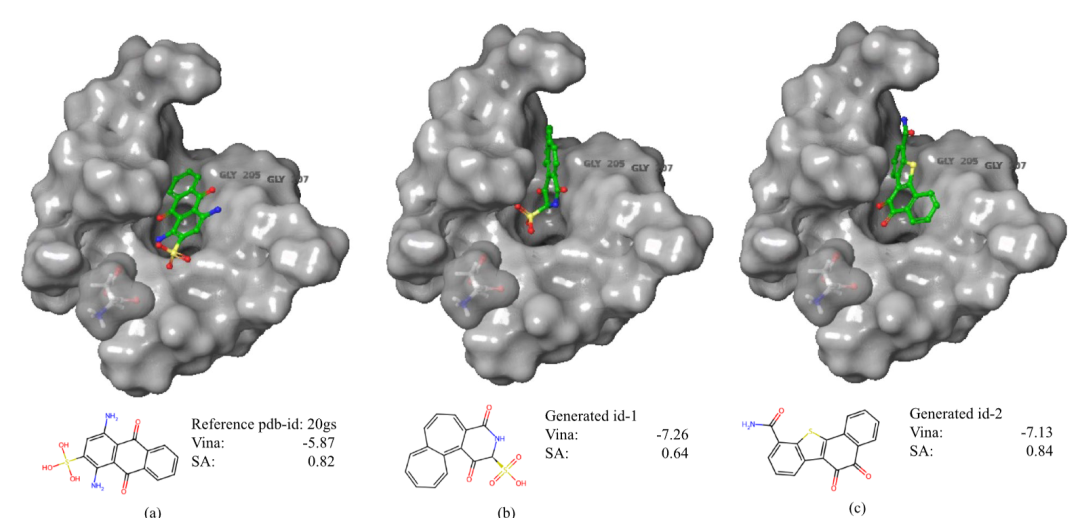
PDB ID 20gs 蛋白口袋中的可视化对比 - (a)参考配体,(b)-(c)两个 PoLiGenX 生成样本,其中生成配体-1 具有最低 Vina 分数但合成可及性略低于参考,生成配体-2 在 Vina 分数和 SA 分数上均优于参考配体
PDB ID 20gs 蛋白口袋中的可视化对比 - (a)参考配体,(b)-(c)两个 PoLiGenX 生成样本,其中生成配体-1 具有最低 Vina 分数但合成可及性略低于参考,生成配体-2 在 Vina 分数和 SA 分数上均优于参考配体
3.3 成药性评估:符合 Lipinski 五倍律
药物开发不仅要考虑活性,还要确保分子能被人体吸收、分布和代谢。Lipinski 五倍律是评估口服药物成药性的经典标准。统计显示,PoLiGenX 生成的分子在分子量、氢键供体/受体数量、logP 等关键参数上均表现良好,平均违反规则的数量仅为 4.28,优于参考数据集的 3.35。
更值得关注的是合成可及性(SA)评分:PoLiGenX 的平均 SA 得分为 0.68(越接近 1 越容易合成),显著优于 DiffSBDD(0.58)和 TargetDiff 的结果。当引入 SA 引导的重要性采样算法后,SA 得分进一步提升至 0.74——这意味着生成的分子不仅理论上有效,还更容易在实验室中真正合成出来。
3.4 潜空间的可解释性
为了验证潜变量 z 的有效性,研究团队对 10 个随机选择的测试靶标各采样 100 个配体,并将其潜嵌入通过 UMAP 降维可视化。结果显示,不同靶标的配体在潜空间中形成了清晰分离的聚类,这表明编码器成功捕获了配体与特定蛋白口袋的上下文关系。
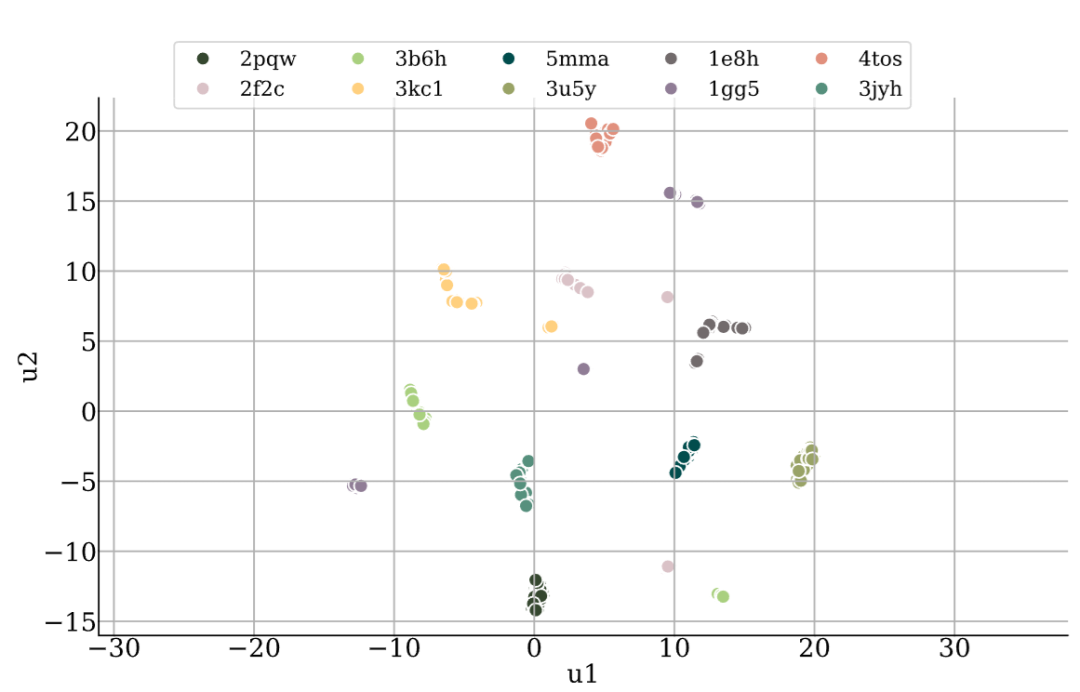
UMAP 降维图 - 展示 10 个随机测试集靶标各 100 个采样配体的潜嵌入二维投影,不同靶标用不同颜色标记,形成明显的聚类
UMAP 降维图 - 展示 10 个随机测试集靶标各 100 个采样配体的潜嵌入二维投影,不同靶标用不同颜色标记,形成明显的聚类
这一发现具有重要意义:它意味着潜空间不是一个随机的数值容器,而是一个结构化的表征空间,其中相似的化学结构和相似的蛋白结合模式在空间中彼此接近。这种可解释性为未来的模型改进提供了方向——例如,可以尝试在潜空间中进行插值或定向搜索,探索特定性质的优化路径。
3.5 可控性验证:α 参数的调节效应
PoLiGenX 的一个独特优势是通过参数 α 实现生成过程的精细控制。实验显示,当 α 从 0.2 增加到 1.0 时,生成配体与参考分子的化学相似度呈现单调上升趋势——α = 0.2 时相似度约 0.25,α = 1.0 时达到 0.40。
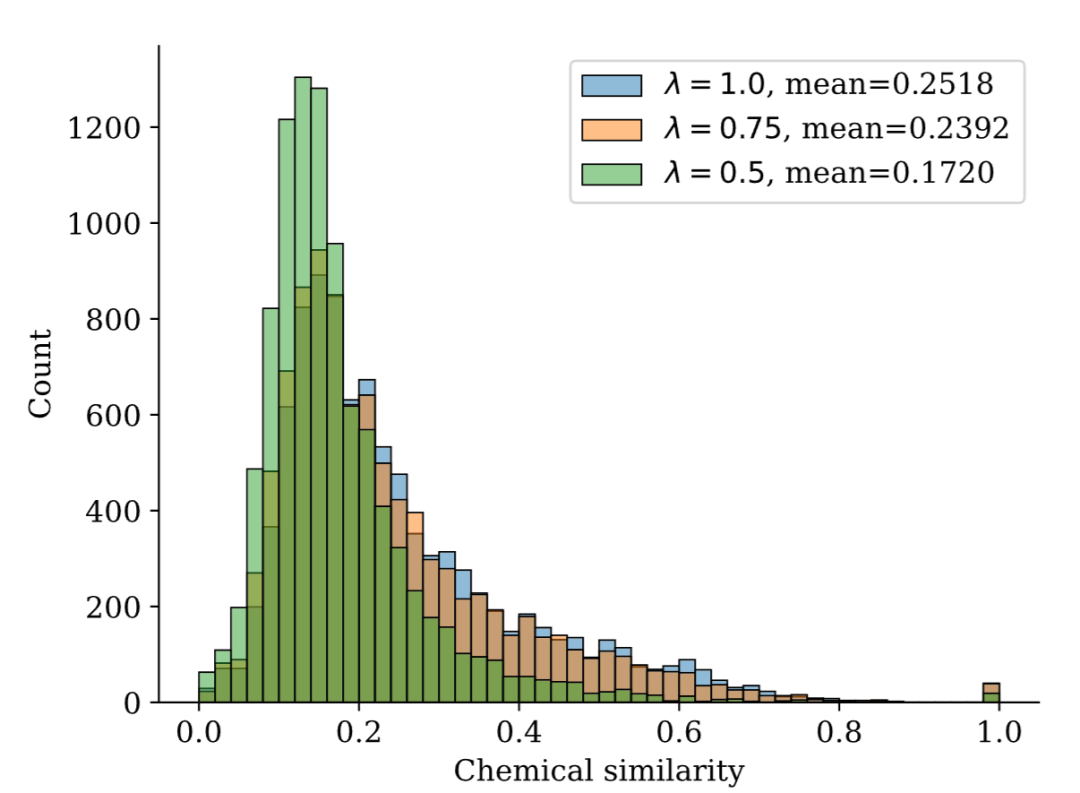
化学相似度密度图 - 展示不同 α 控制参数下生成配体的化学相似度分布,随着 α 增大,分布峰值右移
化学相似度密度图 - 展示不同 α 控制参数下生成配体的化学相似度分布,随着 α 增大,分布峰值右移
这种可调节性在实际应用中极具价值。在先导化合物优化的早期阶段,研究者可能希望进行大胆的骨架跃迁,此时可设置较小的 α;而在后期精修阶段,则可提高 α 以保留已验证的药效团结构,仅对外围基团进行微调。
四、局限与启示
当前方法的不足
尽管 PoLiGenX 在多个维度上取得了显著进步,但仍存在改进空间:
刚性蛋白假设:目前的模型将蛋白视为静态结构,未考虑侧链和主链的动态变化。然而真实的蛋白-配体结合是一个诱导契合过程——配体结合会引发蛋白构象的调整。未来的改进方向可能包括引入多构象蛋白口袋或与分子动力学模拟耦合。
稀有骨架的探索深度:虽然 PoLiGenX 能在参考分子周围进行有效探索,但对于发现全新骨架类型的能力仍有限。这可能需要结合更先进的采样策略,如主动学习或强化学习框架,在保证质量的前提下扩大探索半径。
计算成本:联合训练编码器和扩散模型需要较大的计算资源(8 块 A100 GPU,300 个 epoch)。对于资源受限的研究团队,如何降低训练成本或开发迁移学习方案值得探索。
启示
PoLiGenX 的成功揭示了几个关键设计原则:
条件信息的表征方式至关重要:相比直接使用表面编码或显式约束,潜变量的隐式引导更加灵活,能在保留核心特征的同时给予模型足够的创造空间。这种"软约束"哲学值得推广到其他生成任务中。
联合训练优于两阶段训练:PoLiGenX 同时优化编码器和生成器,使得潜变量的学习与扩散过程的重构损失形成闭环反馈。这避免了预训练编码器可能产生的表征偏差,确保了两个模块的协同工作。
物理约束应嵌入模型设计:通过评估应变能和立体冲突,研究团队展示了生成模型不应仅追求统计上的似然性,还需符合物理化学规律。将 Vina 打分、氢键分析等物理验证纳入评估体系,是推动 AI 药物设计走向实用的必要步骤。
五、展望:迈向智能化药物设计的下一步
PoLiGenX 代表了结构辅助药物发现从"盲目生成"到"精准设计"的范式转变。它不再是简单地向化学空间撒网捕鱼,而是像经验丰富的药物化学家一样,基于已有的知识进行系统性的结构优化。
在实际应用场景中,可以设想这样的工作流程:高通量筛选识别出初始苗头化合物 → PoLiGenX 生成数百个形状相似但化学多样的类似物 → 通过 ADMET 预测和对接打分筛选 Top 候选 → 实验验证并反馈数据 → 迭代优化生成策略。这种人机协同的闭环设计有望大幅缩短新药研发周期。
从技术演进的视角看,PoLiGenX 提出的潜空间条件机制具有很强的可扩展性。未来可以将更多约束整合进潜变量:例如编码多个参考分子的共识特征,或引入药效团约束、反应性约束等先验知识。这些方向都值得深入探索。
最终,AI 药物设计的终极目标不是取代药物化学家,而是增强其创造力——让机器承担繁重的结构枚举和性质预测工作,将人类的洞察力解放出来,专注于关键决策和实验验证。PoLiGenX 的工作为我们勾勒出了这一未来的轮廓。
值得思考的问题
Q1: 潜空间条件与传统的基于片段的药物设计有何本质区别?
传统 FBDD 通常通过刚性对接将预定义的分子片段组装到蛋白口袋中,这是一个离散的组合优化过程。PoLiGenX 的潜空间条件则是连续的、端到端的生成过程——它不依赖片段库,而是通过学习参考分子的隐式表征,在连续空间中生长出新分子。这种方法能探索片段库之外的化学空间,且生成的分子保证了结构的完整性和合理性。
Q2: 如何理解"应变能"和"立体冲突"在评估生成配体质量中的重要性?
应变能反映了分子内部偏离平衡几何构型的程度——高应变能意味着化学键被过度拉伸或压缩,这样的分子在真实环境中很不稳定。立体冲突则指原子间距小于范德华半径之和,产生强烈的排斥作用。这两个指标共同决定了分子构象的物理合理性。PoLiGenX 在这些指标上的优势表明,通过参考分子的引导,模型学会了生成更符合化学直觉的低能构象,这对后续的分子动力学模拟和实验验证至关重要。
Q3: PoLiGenX 的潜空间是否可以直接用于分子性质预测或虚拟筛选?
这是一个很有前景的方向。由于潜变量 z 是通过不变图神经网络编码的,它天然包含了分子的结构和化学信息。论文中已经展示了将其用于辅助任务(预测原子数)的可行性。理论上,可以在潜空间上训练额外的回归或分类模型,预测溶解度、膜透性等性质,或直接在潜空间中进行最近邻搜索以实现快速虚拟筛选。这种端到端的表征学习避免了手工特征工程,可能比传统的分子指纹更具表达力。
Q4: 对于靶标未知的孤儿配体(Orphan Ligands),PoLiGenX 的方法是否适用?
PoLiGenX 的核心优势在于蛋白口袋条件和参考分子条件的联合作用。如果缺乏蛋白结构信息,模型退化为纯粹基于参考分子的生成,此时失去了靶向设计的能力。不过,可以考虑以下变通方案:①使用同源建模或 AlphaFold 预测蛋白结构;②采用基于药效团的虚拟口袋表征;③训练一个不依赖蛋白条件的变体模型,专注于分子性质优化。这些都是值得探索的研究方向。
Q5: 如何将 PoLiGenX 的思想应用到其他分子生成任务,如反应产物预测或材料设计?
潜空间条件生成的核心思想——通过编码参考样本引导生成过程——具有普适性。在反应产物预测中,可以将反应物和反应条件编码为潜变量,引导生成器预测产物结构;在材料设计中,可以基于已知性能的材料结构进行改进优化。关键在于设计合适的编码器和条件注入机制,使其适应特定领域的对称性和约束。PoLiGenX 展示的自适应层归一化和变分框架提供了一个通用的技术模板。
参考文献:Le, T., Cremer, J., Clevert, DA. et al. Equivariant diffusion for structure-based de novo ligand generation with latent-conditioning. J Cheminform 17, 90 (2025). https://doi.org/10.1186/s13321-025-01028-x
代码数据:
https://github.com/pfizer-opensource/e3moldiffusion/tree/poligenX
欢迎在评论区分享您对潜空间条件扩散方法的看法或应用经验。如果您对 AI 药物发现和结构辅助药物设计感兴趣,欢迎关注 MindDance 公众号获取更多前沿内容,也欢迎将文章分享给您的同行。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-07,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

