TarPass:靶标感知分子生成模型的全面基准测评与启示
TarPass:靶标感知分子生成模型的全面基准测评与启示
用户1151118
发布于 2026-01-08 13:33:51
发布于 2026-01-08 13:33:51
在AI驱动药物发现的浪潮中,靶标感知分子生成模型被寄予厚望——它们理论上能精准锚定蛋白质靶标特征,高效生成具有潜在活性的新型化合物,大幅缩短早期药物研发周期。但一个关键问题始终悬而未决:这些模型是真正掌握了蛋白质-配体相互作用(PLIs)的核心原理,实现了理性设计,还是陷入了Texas Sharpshooter,仅通过事后合理化包装输出结果?浙江大学团队近期在ChemRxiv发表的研究,通过构建名为TarPass的全面基准体系,对15种主流模型进行了系统性测评,为解答这一问题提供了权威答案,也为领域发展指明了清晰方向。
本文针对当前靶标感知分子生成模型评估的核心痛点——缺乏统一标准、评估维度片面、难以区分真实性能与表面效果——构建了包含18个特征明确靶标的TarPass基准数据集。研究团队从PLIs、分子合理性和类药性三个核心维度,对3D原位生成、非3D生成和优化型生成三大范式的模型进行了全面且严格的测评。结果表明,现有模型在捕捉PLIs核心原理上仍存在显著不足,但预训练模型和优化策略能有效提升分子质量;基于测评洞察设计的多层虚拟筛选流程,可高效富集潜在候选化合物,为实验验证提供有力支撑。总体而言,TarPass基准揭示了当前模型与真正理性设计工具的差距,同时也明确了其探索新型化学空间的独特价值与未来改进方向。


研究背景
早期药物发现面临成本高、效率低的严峻挑战,传统高通量筛选需遍历海量化学库,却常因化学空间覆盖不足而收效甚微。生成模型的出现为解决这一问题提供了新路径,其中靶标感知生成模型凭借以靶标为导向的核心优势,成为基于结构的药物发现的热门工具——它们不再是盲目探索化学空间,而是将蛋白质靶标信息(如结构、序列)作为约束条件,理论上能大幅提升生成化合物的生物合理性。
当前靶标感知分子生成主要形成三大技术范式:3D原位模型直接基于蛋白质口袋结构,通过扩散、流匹配等方法构建3D配体,能编码空间互补性但计算成本高、对结构输入质量要求严苛;非3D模型不依赖 explicit 结构几何,而是基于序列嵌入等模态生成2D分子图或化学字符串,适用性广、效率高但缺乏结构基础,可解释性与结合模式真实性不足;优化型模型则在上述两种模型基础上引入强化学习、偏好对齐等后处理目标,能引导采样向理想化学空间偏移,但易受评分函数缺陷影响,面临多目标优化的权衡难题。
尽管领域发展迅速,但模型评估体系的滞后严重制约了技术落地。现有评估多依赖对接分数和类药性指标等粗糙替代物,难以全面反映PLIs的真实性与分子有效性;同时,选择性案例分析的普遍存在,使得模型性能容易被“事后合理化”,掩盖了实际缺陷。现有基准要么侧重结合亲和力而忽略相互作用模式,要么局限于单一范式,缺乏跨范式的统一评估框架,导致不同模型的性能缺乏可比性,领域难以形成统一的发展共识。正是在这一背景下,TarPass基准的构建与应用具有了重要的现实意义。
核心方法:TarPass基准的设计与评估体系
基准数据集的构建逻辑
TarPass基准的核心优势在于其数据的完整性、代表性与评估的全面性,具体构建围绕四个关键部分展开。在靶标选择上,研究团队从POKMOL-3D数据集筛选出20个结构(涵盖18个靶标),包括激酶、GPCRs、核受体、非激酶酶等多个家族,所有靶标均有已批准药物或临床候选物支持,且通过严格的序列与结构相似性分析,确保与CrossDocked2020、PDBbind等常用训练集的直接重叠度极低(序列一致性多低于1%),避免了数据泄露导致的评估偏差。
关键交互信息的整理遵循严格的科学原则:仅纳入与天然配体结合相关的保守残基与相互作用、有晶体学证据或突变实验支持的相互作用,以及通过PLIP工具在不同结构条目间一致识别的相互作用,主要包括氢键、疏水作用、盐桥、π-堆积和卤素键等,确保了PLIs评估的针对性与可靠性。参考化合物方面,从BindingDB收集实验验证的活性化合物,同时从ChEMBL随机选取1000个分子作为基线,通过t-SNE降维分析验证了活性化合物的靶标特异性聚类特征,为评估提供了坚实的对照基础。
统一评估流程的设计则保障了测评的公平性:模型针对每个靶标生成1000个独特分子,经对接模块计算分数、构象和相互作用后,通过MoleEval(分子性质评估)、DockEval(PLIs分析)和综合分析模块完成多维度评估。其中,MoleEval模块计算理化描述符、类药性指标、结构复杂性等参数,DockEval模块量化关键相互作用匹配率、结合模式相似性等核心指标,综合分析模块则进行靶标水平的统计与跨模型对比,形成了一套闭环的评估体系。
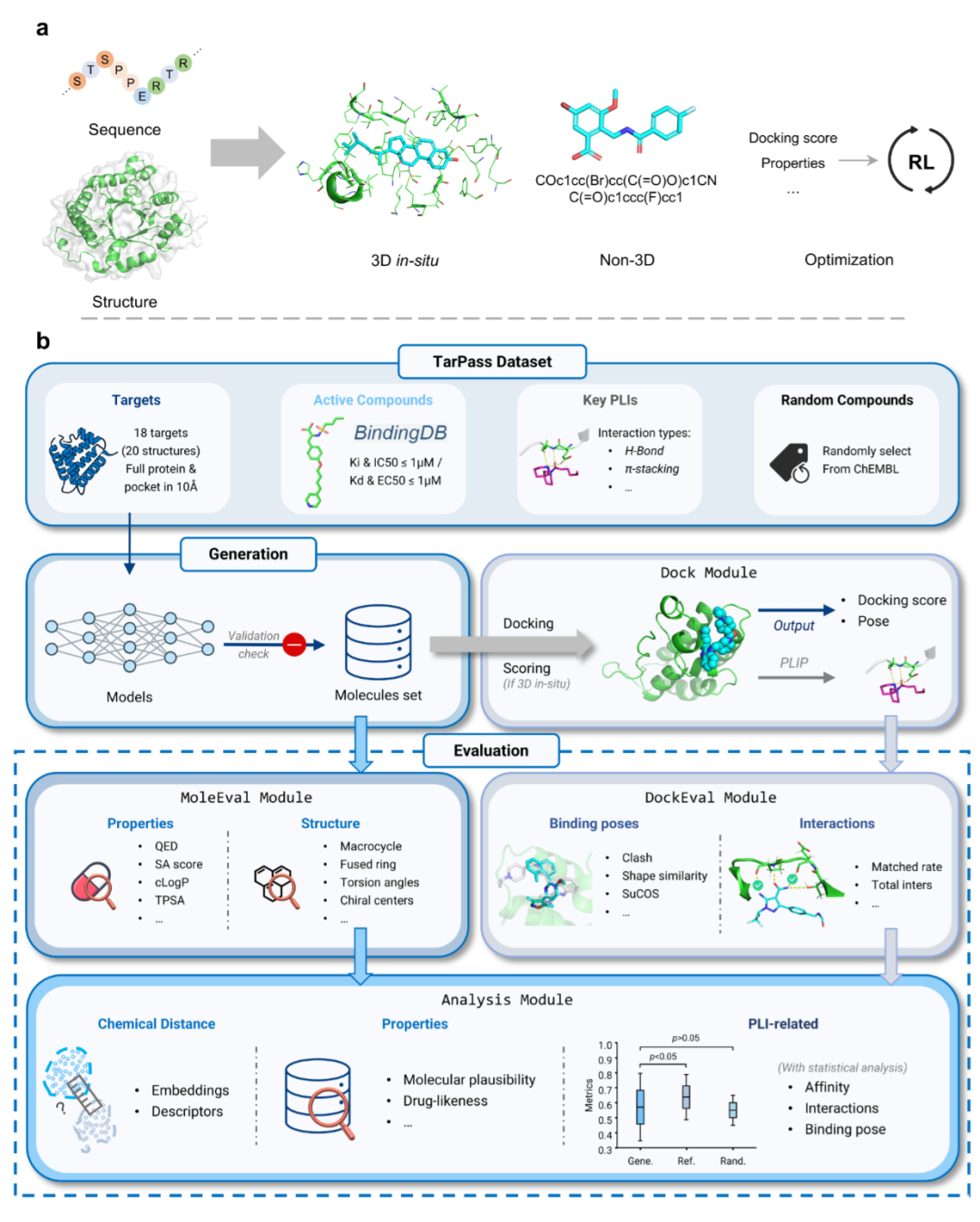
靶标感知分子生成范式与 TarPass 基准工作流
靶标感知分子生成范式与 TarPass 基准工作流
多维度评估指标体系
研究构建的评估体系全面覆盖了模型性能的核心维度,确保结果的科学性与实用性。基础性能评估包括分子有效性(结构合法性)、唯一性(避免重复)和生成速度,直接反映模型的工程化应用潜力;PLIs相关评估是核心重点,涵盖结合亲和力(对接分数与重打分结果)、关键相互作用匹配率(完全匹配与部分匹配)、构象无冲突率、结合模式相似性(SuCOS值)等指标,深入刻画模型对分子结合机制的捕捉能力;分子合理性与类药性评估则关注结构完整性、立体化学特征、环系统复杂性、QED值、Lipinski规则符合度、合成可及性(SA Score)及结构警报(PAINS、SureChEMBL)等,确保生成分子的实际可开发价值;化学空间距离评估通过内部多样性(IntDiv、#Circle)、嵌入距离(Tanimoto相似度、FCD)和描述符距离(Wasserstein距离),量化生成分子与活性化合物的化学空间重叠度;靶标特异性评估则从蛋白质家族水平区分能力与结构相似靶标的识别能力(apo-holo对、位点特异性对)两个层面,检验模型的靶向精准性。
关键结果:模型性能的全面解构
基础性能
不同技术范式的模型在基础性能上呈现出鲜明差异。非3D模型展现出显著的速度优势,生成单个分子的时间普遍低于0.1秒,其中REINVENT的生成速度最快,仅需0.011秒/分子,远超其他范式;3D原位模型的生成效率则相差较大,流基方法需数秒/分子,而扩散模型和自回归模型的生成时间常超过10秒,SurfGen的平均生成时间更是达到51.051秒/分子,这一差异直接限制了3D原位模型在大规模筛选场景的应用。
在有效性与唯一性方面,多数模型的分子有效性较高(>0.96),但DrugFlow系列模型表现不佳,有效性仅0.831;值得注意的是,3D原位模型的唯一性普遍低于非3D模型,MolCraft的Uniq@1k仅为0.498,反映出其生成多样性不足的问题,而DeepBlock、DRAGONFLY等非3D模型的唯一性均达到1.0,展现出更优的化学空间探索能力。部分模型存在特定结构处理缺陷,如SimpleSBDD无法处理HDAC6中的锌离子,TamGen不能接受AlphaFold预测的5-HT2A结构,这些细节也为实际应用中的模型选择提供了重要参考。
PLIs建模:核心瓶颈尚未突破
PLIs建模作为靶标感知分子生成的核心目标,仍是当前模型的主要短板。在结合亲和力方面,仅5个未优化模型在超过50%的靶标上显著优于随机分子,3D原位模型的平均表现略优于非3D模型,但多数模型的性能并未超过随机基线。MolCraft和DrugFlow是唯一两类持续优于非3D方法的3D原位模型,重打分后中位分数低于-7.5,而部分低性能模型甚至出现正的对接分数,表明其生成的分子与靶标的结合能力极差。
关键相互作用匹配率的表现更为严峻,参考配体的完全匹配率(51.4%)是随机基线(22.3%)的两倍多,而多数模型的匹配率与随机分子相当,仅DrugFlow、MolCraft及其优化变体接近参考水平。模型对相互作用先验的利用效果差异显著:Lingo3DMol凭借有效的相互作用先验设计,性能超过多个3D原位模型,而IPDiff虽引入相互作用先验,表现却低于随机基线,凸显了先验设计的重要性。3D原位模型的初始构象普遍存在空间冲突问题,无冲突率多低于85%,经优化后可提升至90%以上;构象重置对接实验进一步表明,初始构象的空间准确性有限,虽模型性能排名保持一致,但整体指标均出现下降,说明3D原位模型的构象预测能力仍需大幅提升。
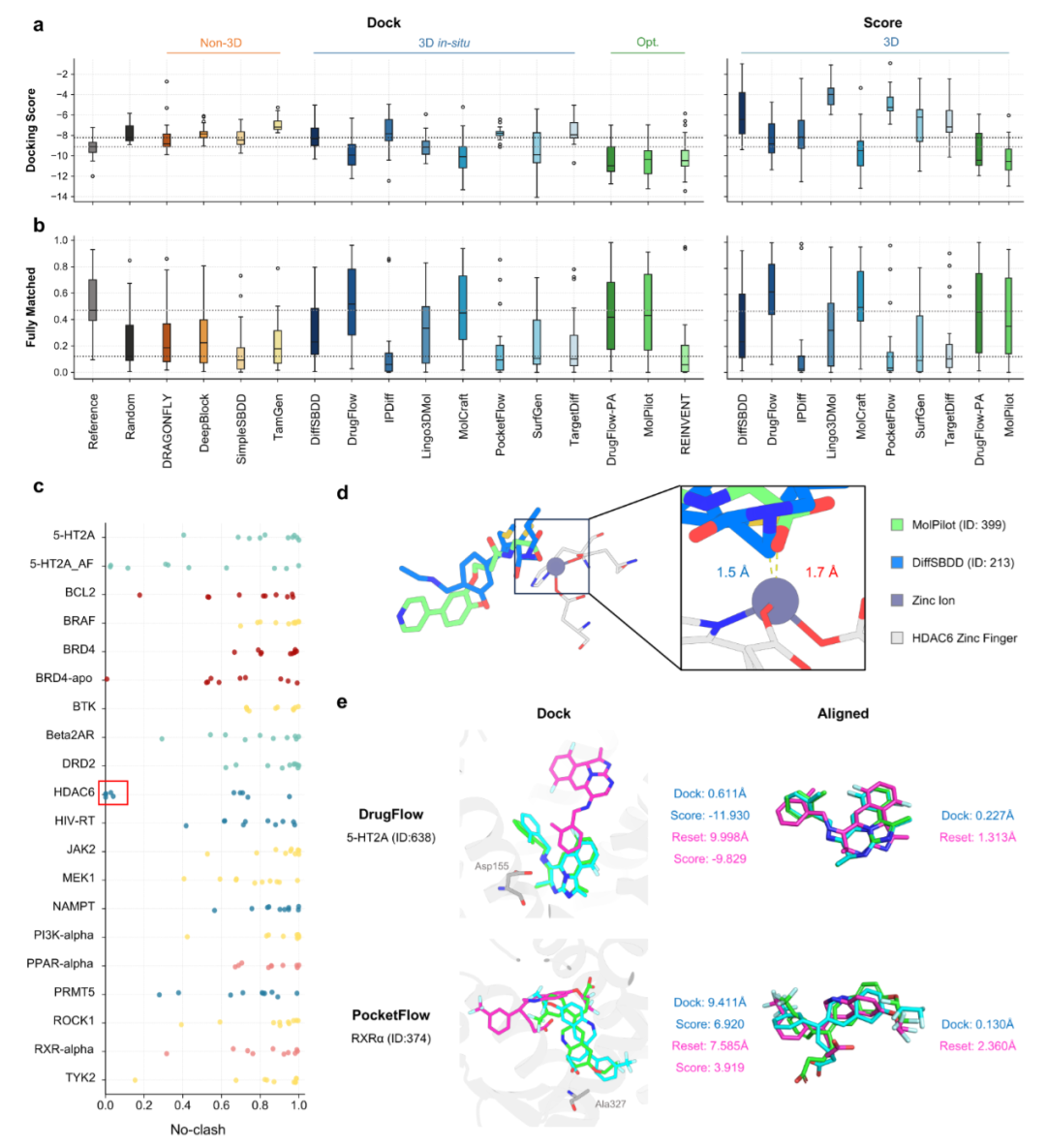
模型在结合亲和力、相互作用及构象质量上的性能分布
模型在结合亲和力、相互作用及构象质量上的性能分布
分子合理性与类药性
分子合理性与类药性的评估结果呈现出明显的范式分化。非3D模型(如DRAGONFLY、DeepBlock)得益于在大规模药物样分子语料库上的预训练优势,生成分子的立体化学特征更接近真实化合物(手性中心<1个),类药性表现更佳(QED多>0.6),结构警报更少,合成可及性更高(SA Score多≤3.0)。相比之下,多数3D原位模型易产生过量手性中心(>3.5个)和高度稠环系统,如SurfGen有74%的分子包含高度稠环,导致其合成可及性较差(SA Score多>3.5),难以满足实际药物开发需求。
优化型模型展现出针对性改善单一属性的能力,例如DrugFlow-PA的SA Score较基础模型提升0.9,SureChEMBL结构警报数量减少超过三倍,但这种优化往往以牺牲其他属性为代价,如DrugFlow-PA在氢键受体数量、杂原子计数等指标上与药物样空间的偏差显著扩大,反映出多目标优化的内在挑战。结构警报分析显示,3D原位模型的SureChEMBL警报数量比参考化合物高出约50%,而除SimpleSBDD外的所有非3D模型均表现优于真实化合物,进一步验证了预训练在提升分子质量上的关键作用。
靶标特异性与化学空间
靶标特异性评估结果表明,现有模型已具备一定的蛋白质家族水平区分能力。激酶和GPCRs等保守性较高的家族中,同一家族靶标的分子相似性显著高于不同家族,RXR-alpha与同家族的PPAR-alpha之间的FCD值明显低于与其他家族靶标的差异,这一趋势与参考分子的表现基本一致。双因素方差分析结果显示,模型和靶标对 docking 分数与相互作用匹配率均存在显著的主效应及交互效应,说明模型确实能部分捕捉靶标特异性特征。
但在结构相似靶标的精细区分上,模型表现仍不尽如人意。针对5-HT2A和BRD4的apo-holo对(RMSD分别为0.402 Å和0.304 Å)以及JAK2-TYK2位点特异性对(RMSD为1.657 Å)的测试显示,仅DeepBlock、DrugFlow和PocketFlow能遵循“apo对FCD值低于位点特异性对”的预期模式,且DrugFlow的优异表现与原始配体采样高度相关,缺乏普适性。多数非3D模型的FCD值集中在跨靶标平均水平,部分3D原位模型(如DiffSBDD、IPDiff)甚至呈现相反趋势,交叉对接实验进一步证实,非3D模型难以区分apo-holo对和位点特异性对的PLIs差异,3D原位模型则对局部口袋环境过于敏感,在holo-apo对比中缺乏稳健性。
化学空间分析显示,多数模型生成的分子集合过于分散,#Circle值普遍超过100,部分3D原位模型甚至超过随机基线(517),增加了后续筛选的计算负担与验证成本。预训练模型(尤其是ChEMBL预训练的模型)生成的分子与药物样空间的偏差更小(FCD移位<2.5),而缺乏预训练的模型则偏离显著,说明预训练能有效帮助模型学习药物样分子的结构与性质分布。
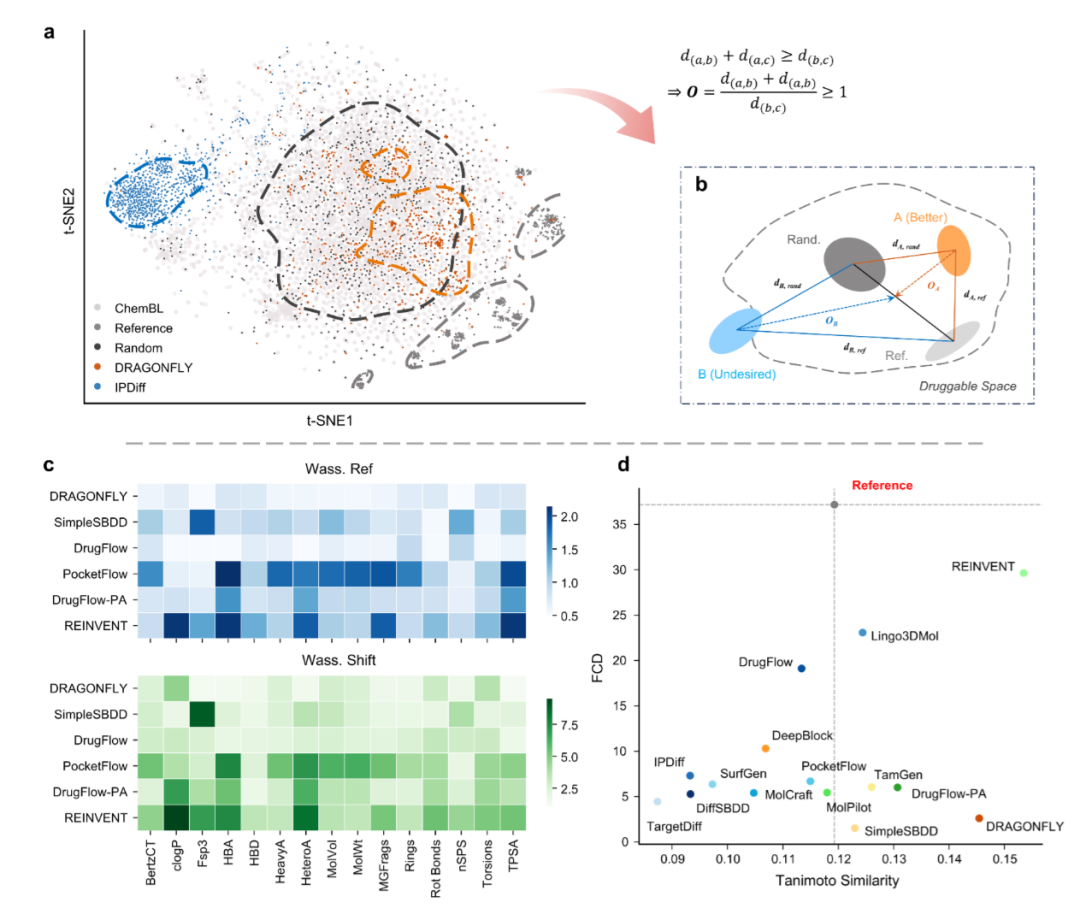
化学空间距离与模型特异性分析。预训练能显著提升分子与药物样空间的一致性,现有模型在跨靶标特异性上表现有限,难以平衡结构多样性与生物相关性。
化学空间距离与模型特异性分析。预训练能显著提升分子与药物样空间的一致性,现有模型在跨靶标特异性上表现有限,难以平衡结构多样性与生物相关性。
虚拟筛选实践:多层流程实现有效富集
尽管模型存在诸多局限,研究团队基于测评洞察设计的多层虚拟筛选流程,仍展现出显著的实用价值。该流程第一阶段通过基于PLIs、结构合理性和类药性的硬筛选,自动剔除大量劣质分子——3D原位模型及其优化变体在PLI水平的通过率超过40%,非3D模型则在结构可行性和类药性上保持优势,最终两类范式均能保留约10%的分子,为后续筛选奠定基础。
第二阶段通过经验性软筛选进一步缩小候选池规模,针对MolCraft(3D原位)和DeepBlock(非3D)的代表模型,结合重对接分数、SuCOS值及理化性质约束(如分子量300-600、clogP 1-6),将候选分子数量缩减至20-30个,达到实验验证的可行规模。对JAK2和TYK2的测试表明,筛选后的候选分子在结合自由能(MM/GBSA)和结构相似性(Tc_max)上均实现有效富集:JAK2的候选分子虽略低于设定阈值,但聚集在“良好结合能+中等新颖性”区域;TYK2则有更多分子同时满足亲和力与相似性标准。所选代表性分子均展现出合理的化学结构与对接构象,虽仍未达到强先导化合物的质量标准(如JAK2候选缺乏额外稳定相互作用,TYK2候选未充分占据选择性口袋),但已初步具备实验验证价值,证明该流程能有效挖掘模型生成结果中的潜在优质分子。
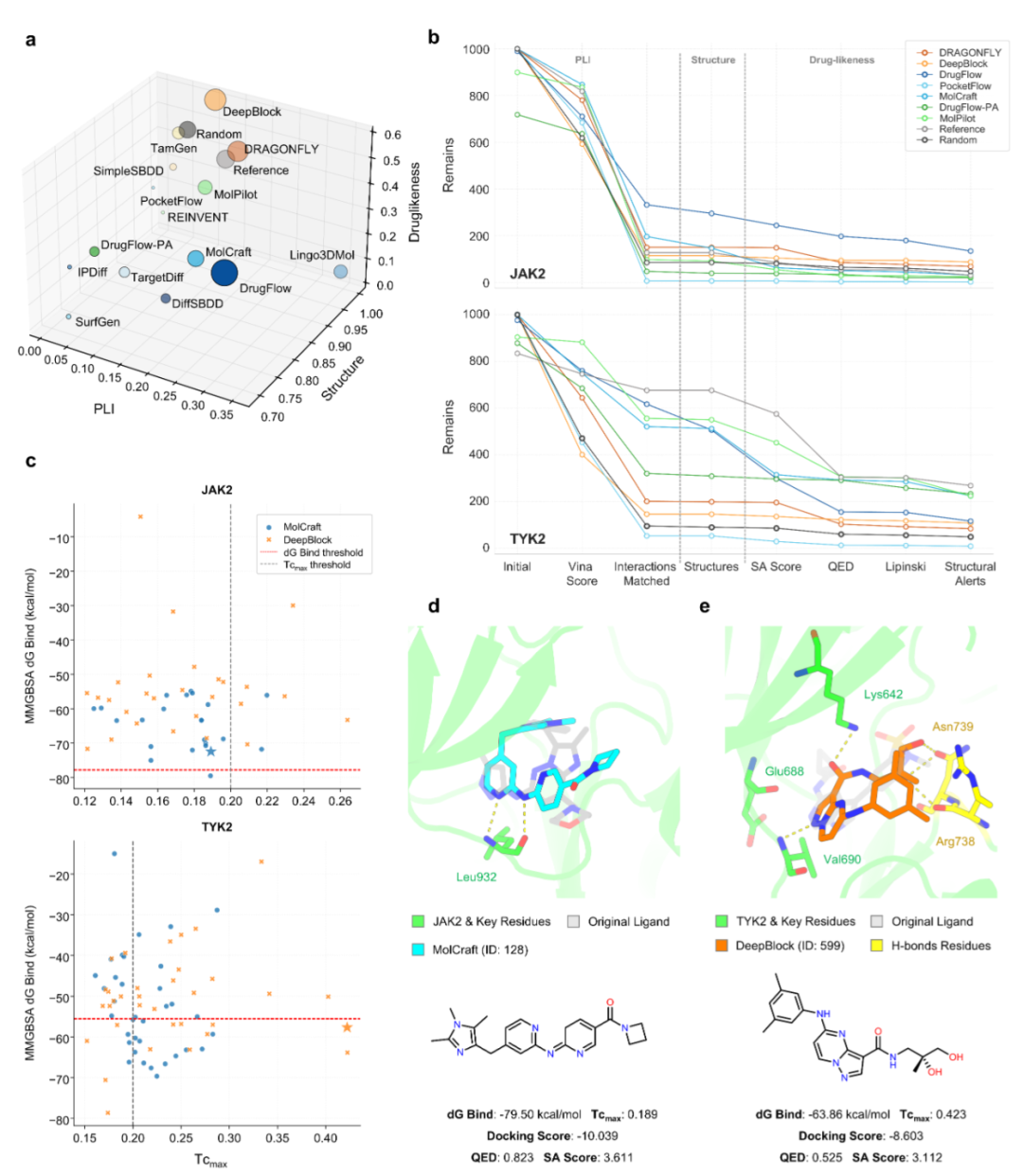
虚拟筛选流程及候选分子富集效果
虚拟筛选流程及候选分子富集效果
研究启示与未来方向
TarPass基准的系统性测评清晰揭示了当前靶标感知分子生成领域的核心现状:现有模型虽具备探索新型化学空间的独特潜力,但距离真正的理性设计工具仍有较大差距。3D原位模型虽能利用空间信息,但其构象预测准确性不足、计算成本高的问题限制了实际应用;非3D模型凭借预训练优势在分子质量上表现更优,但难以有效重构PLIs;优化型模型能定向改善特定属性,却面临多目标优化的权衡难题。靶标特异性方面,现有模型仅能区分蛋白质家族水平的差异,无法精准识别结构相似靶标的细微区别,这一局限严重影响了生成化合物的靶向性与实用性。
从应用视角来看,当前模型更适合作为随机分子生成器,为药物发现提供现有筛选库之外的新型化学实体,而非直接生成可进入临床前研究的先导化合物。实验验证结果显示,模型生成的分子多为小分子、易合成结构,虽易于获取,但富集效率较低,验证后活性多处于微摩尔水平,领域内所谓的模型优越性往往存在事后合理化的偏差,这一认知也为实际应用中的预期管理提供了重要参考。
未来研究应重点聚焦三个核心方向:一是强化PLIs建模的准确性,通过融入相互作用模式、电子密度等先验知识,深入捕捉蛋白质-配体结合的生物物理本质;二是优化预训练策略,扩大化学空间覆盖度,实现多样性与靶向性的平衡,避免过度分散或过度局限;三是拓展基准的评估场景,将分子优化等实际应用需求纳入评估体系,构建更全面的跨范式评估标准。此外,金属离子、保守水分子等辅因子的明确处理,3D原位模型对口袋环境的稳健感知能力提升,以及多目标优化算法的创新,也是提升模型实用性的关键所在。
参考文献:Qin R, Chen Z, Li Y, Fang M, Shen L, Su Y, et al. Revisiting Target-Aware de novo Molecular Generation with TarPass: Between Rational Design and Texas Sharpshooter. ChemRxiv. 2026; doi:10.26434/chemrxiv-2026-dhdqk This content is a preprint and has not been peer-reviewed.
代码数据:
https://github.com/sorui-qin/TarPass
评估结果:
https://doi.org/10.5281/zenodo.17649772待传
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-05,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

