Nat. Mach. Intell. | 消除数据偏差可提升结合亲和力预测的泛化性能
Nat. Mach. Intell. | 消除数据偏差可提升结合亲和力预测的泛化性能
用户1151118
发布于 2026-01-08 14:09:35
发布于 2026-01-08 14:09:35
DRUGONE
计算机辅助药物设计依赖于准确的评分函数来预测蛋白–配体相互作用的结合亲和力。然而,PDBbind 数据库与评分函数比较评估基准(CASF)之间存在严重的训练–测试数据泄漏,使得当前深度学习模型的性能被过度估计。为此,研究人员提出了一个基于结构的过滤算法,构建了一个新的训练集 PDBbind CleanSplit,在消除数据泄漏与冗余后重新训练多种主流模型。结果显示,这些模型的基准性能显著下降,表明其高分主要源自数据泄漏。相比之下,研究人员提出的 GEMS(Graph neural network for Efficient Molecular Scoring) 模型在 CleanSplit 上仍保持高性能,并能泛化至独立测试集。GEMS 结合稀疏图建模与语言模型迁移学习,实现了对蛋白–配体结合的真实理解,为结构基础药物设计提供了鲁棒的泛化框架。

结构基础药物设计(SBDD)旨在设计能高亲和力结合特定靶蛋白的小分子药物。近年来,深度神经网络的引入推动了计算药物设计的革新,从蛋白质折叠(如 RoseTTAFold、AlphaFold3)到去噪扩散模型(如 DiffSBDD),AI 能够生成全新的蛋白–配体结合模式。然而,这些生成的结合构象并不保证具有药物级别的亲和力,因此高质量的评分函数成为关键。传统基于力场或经验的打分方法(如 AutoDock Vina、GOLD)计算开销大且准确度有限。虽然卷积神经网络和图神经网络模型的出现提升了预测能力,但泛化性差仍是主要瓶颈。研究人员发现,许多模型在独立数据集上的表现远低于 CASF 基准,原因在于 PDBbind 与 CASF 数据集之间的高度相似性导致了训练–测试泄漏。这使得模型在测试集上“记忆”训练数据而非学习真实相互作用规律。
方法概要
- 数据集:基于 PDBbind v2020(约 19,443 个复合物),CleanSplit 通过蛋白结构比对(TM-align)、配体指纹相似度(Tanimoto)与位姿 RMSD 过滤获得约 18,600 个样本。
- 特征构建:节点特征包含原子类型、芳香性、氢键数等;蛋白节点补充氨基酸种类与语言模型嵌入;全局特征初始化自 ChemBERTa 生成的配体向量。
- 模型训练:基于 PyTorch Geometric 实现,五折交叉验证,早停机制,GPU 加速(RTX 3090/4090)。
- 模型比较:包括 Pafnucy、GenScore、传统评分函数(AutoDock Vina、GlideScore)以及搜索算法基线。
结果
结构相似性过滤与 CleanSplit 构建
研究人员开发了一种结构层面的聚类算法,结合 蛋白结构相似度(TM-score)、配体化学相似度(Tanimoto 指数) 与 结合位姿相似度(对齐 RMSD) 三个指标来检测数据泄漏。分析发现,PDBbind 与 CASF 间约 49% 的复合物具有高度结构相似性,即存在严重的“训练–测试重叠”。CleanSplit 通过多层筛选规则删除了这些重复结构和高度相似的配体,使训练集与 CASF 严格分离,并进一步减少了训练集内部的冗余(移除约 7.8% 数据),最终形成一个更具多样性的训练基础。
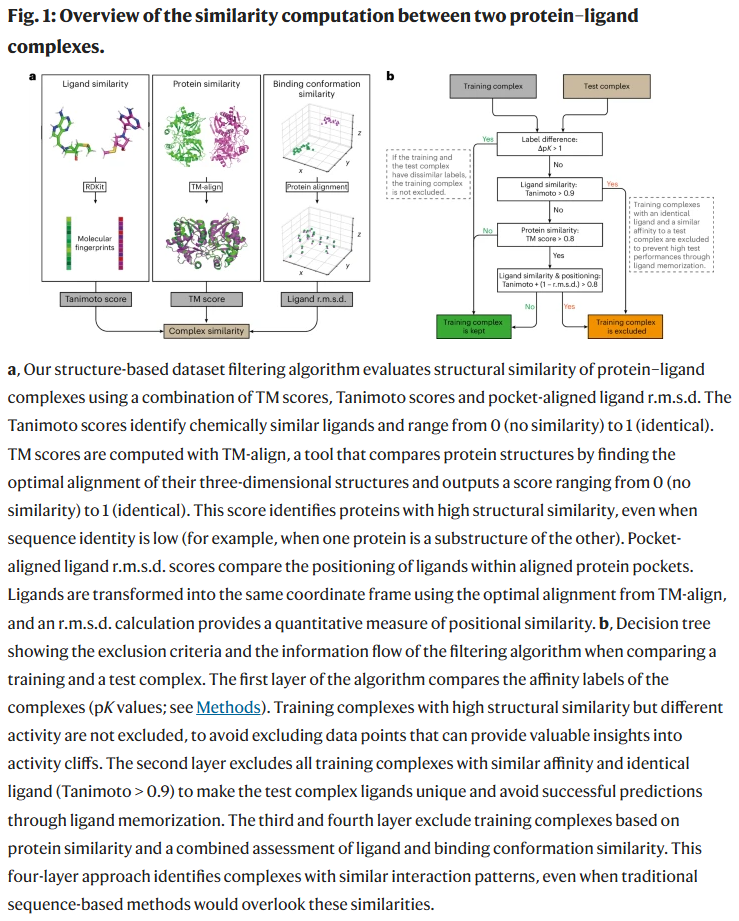
数据泄漏对模型性能的影响
为量化泄漏效应,研究人员设计了两个简单算法:
- 基于最相似复合物的平均打分法;
- 基于最相似配体的平均打分法。
这两个非学习算法在未过滤数据上竟能达到与主流深度学习模型相当的性能,而在 CleanSplit 上性能急剧下降,证明此前高性能主要源于记忆训练数据而非理解化学规律。
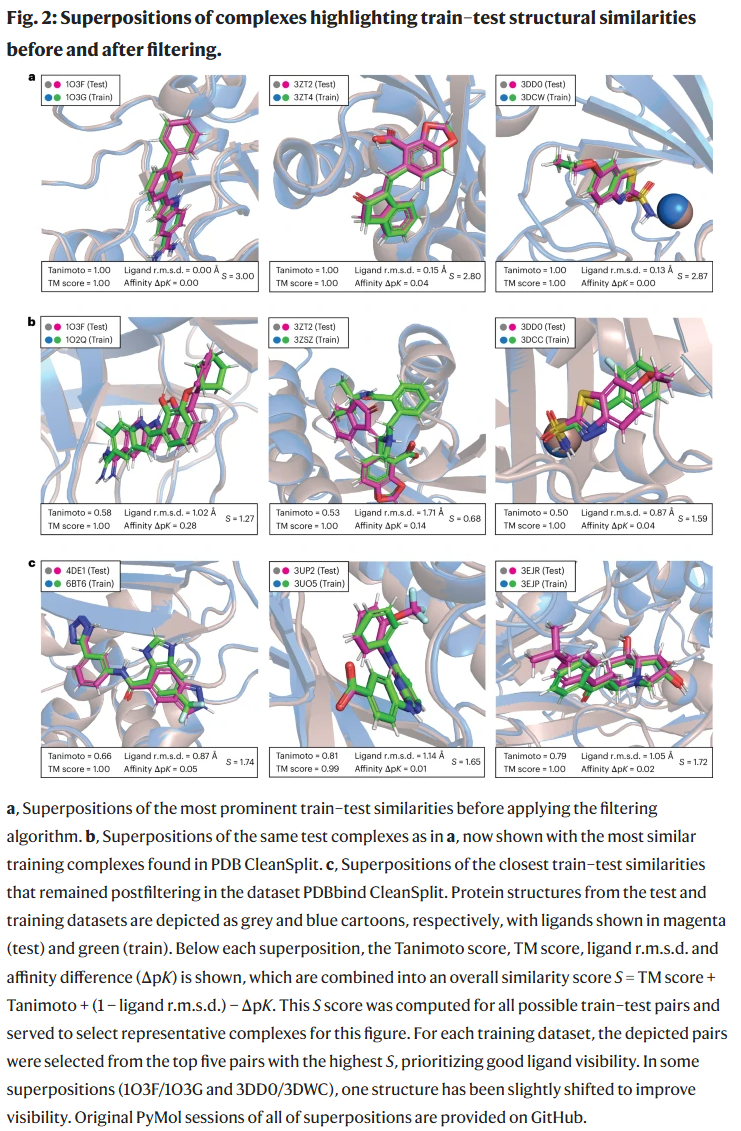
重新训练主流模型(Pafnucy 与 GenScore)
研究人员在 CleanSplit 上重新训练了两个代表性模型:
- Pafnucy(3D CNN 模型)
- GenScore(GNN 模型)
结果显示,二者的性能均显著下降,尤其是 Pafnucy 的 CASF 相关系数降至 0.65 左右,验证了原有结果被数据泄漏严重高估。GenScore 在 CleanSplit 上稍具鲁棒性,但也显示出泛化性能受限。
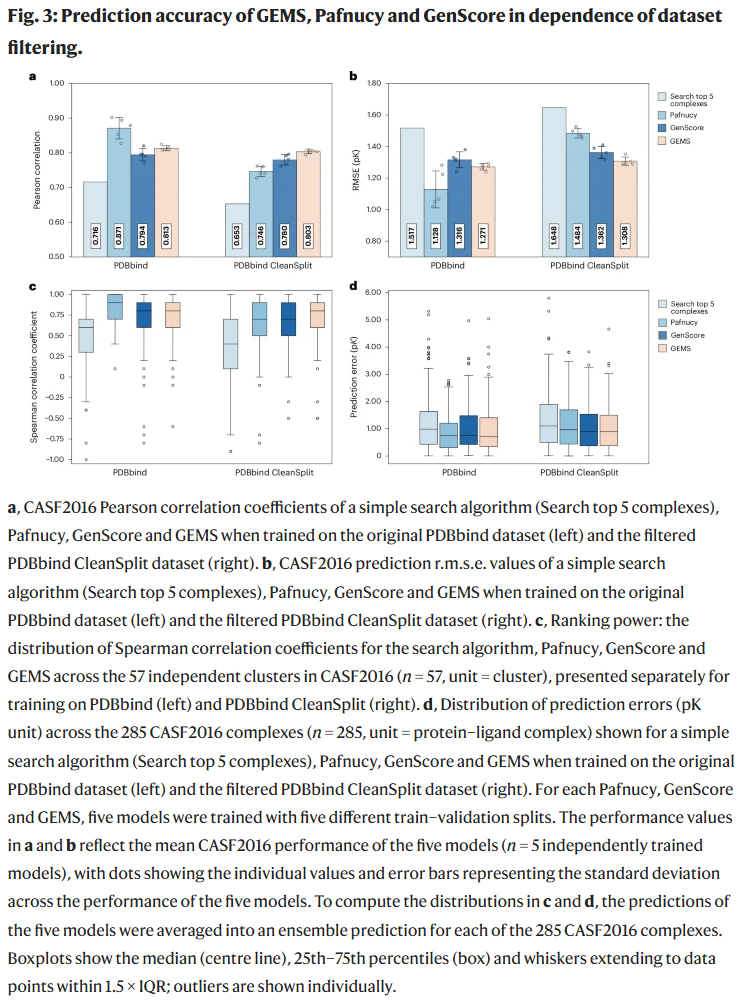
新模型 GEMS 的提出与表现
为克服上述问题,研究人员提出了 GEMS(Graph neural network for Efficient Molecular Scoring) 模型。该模型的核心思路是:
- 使用稀疏图结构同时表示配体原子与蛋白氨基酸节点;
- 在节点特征中引入 语言模型嵌入(ChemBERTa、ESM2、Ankh),捕捉化学与序列语义;
- 采用 多层图注意卷积(GATConv)与边卷积(EdgeConv) 实现特征融合。
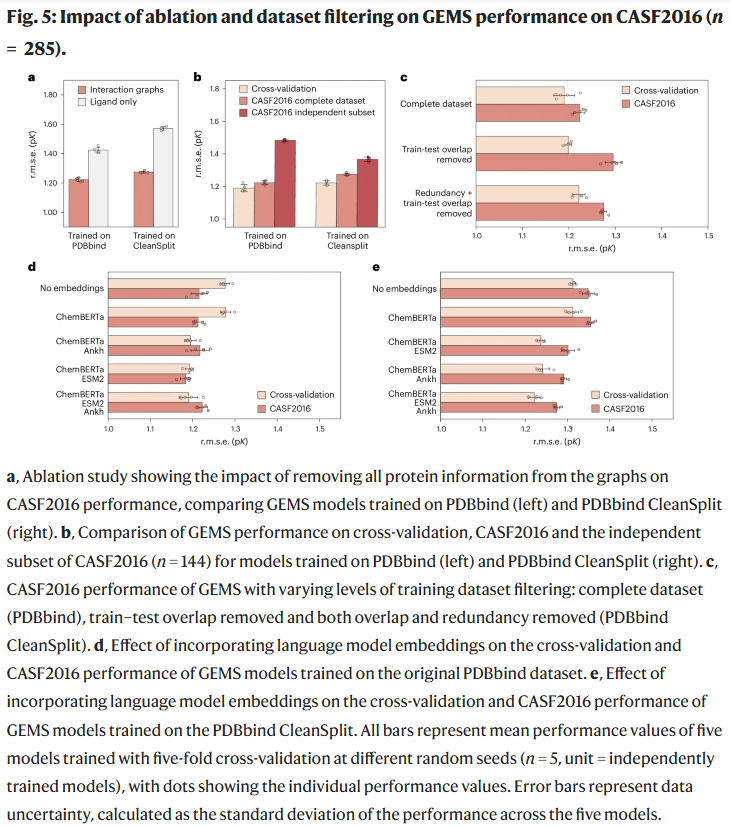
在 PDBbind CleanSplit 上,GEMS 达到 Pearson R = 0.803, RMSE = 1.308,超越所有在无过滤数据上训练的主流模型。更重要的是,当移除蛋白信息后,其性能显著下降,说明模型的预测真正依赖蛋白–配体相互作用而非“配体记忆”。
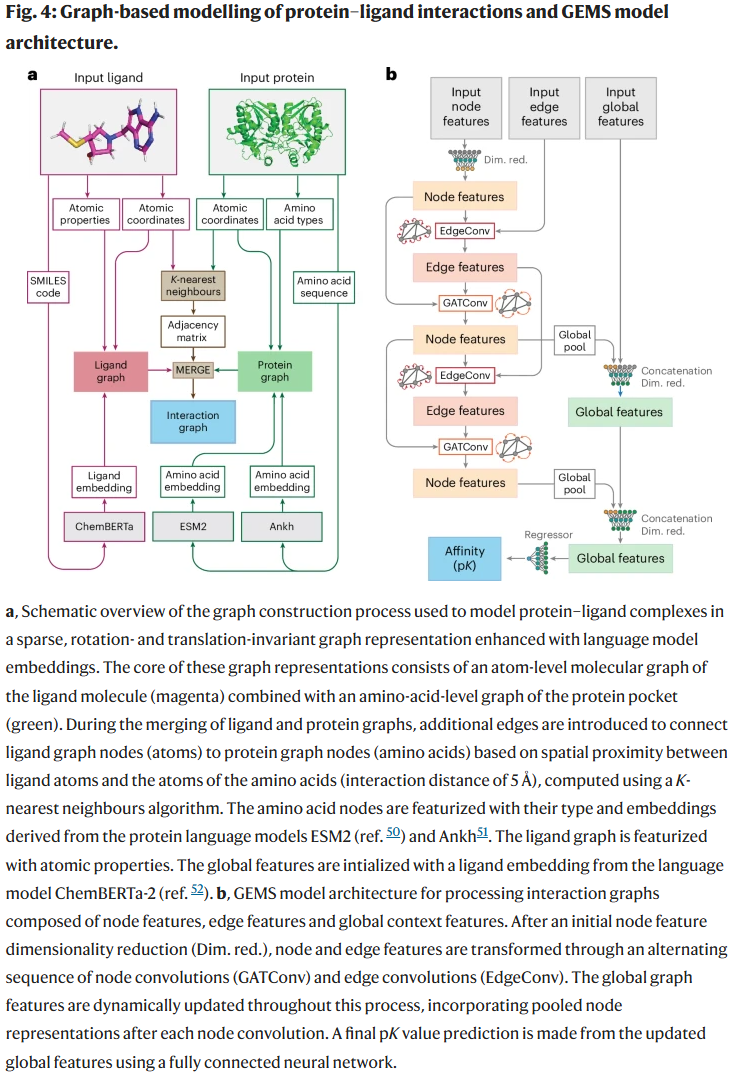
语言模型嵌入的增益与冗余影响
研究人员进一步发现:
- 在未经过滤的 PDBbind 上,增加语言模型特征可提升交叉验证性能但不提升测试集性能,说明模型仍在利用数据泄漏。
- 在 CleanSplit 上,嵌入语言模型后,训练与测试性能均显著提高,表明在消除偏差后模型能真正受益于语义丰富的特征。
此外,移除训练冗余反而提高了测试性能,说明去除过多相似样本可防止过拟合,促进泛化。
讨论
PDBbind CleanSplit 为结合亲和力预测提供了更可靠的训练与评估基础。它消除了常见数据集中的结构重复与泄漏问题,使模型无法依赖“记忆”获得高分,而必须学习蛋白–配体相互作用的本质。
GEMS 在这种环境下仍能保持高准确度和高计算效率(比 Pafnucy 快 25 倍,比 GenScore 快 100 倍),并能泛化到完全独立的 OOD 数据集,展示了强大的实际适用性。
研究人员强调,GEMS 为结构基础药物设计提供了一个关键环节:它可作为生成模型(如 RFdiffusion、DiffSBDD)之后的精准评分工具,帮助识别具有治疗潜力的候选分子。
未来方向包括:
- 引入更高级的配体相似度计算(如 ROCS 评分);
- 增强图卷积模块的可解释性;
- 通过引入对接构象扩充训练数据,以适应虚拟筛选中“构象选择”的任务。
整理 | DrugOne团队
参考资料
Graber, D., Stockinger, P., Meyer, F. et al. Resolving data bias improves generalization in binding affinity prediction. Nat Mach Intell (2025).
https://doi.org/10.1038/s42256-025-01124-5
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-22,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

