Nat. Biotechnol. | 基于深度学习的抗菌化合物虚拟筛选
Nat. Biotechnol. | 基于深度学习的抗菌化合物虚拟筛选
用户1151118
发布于 2026-01-08 14:12:29
发布于 2026-01-08 14:12:29
DRUGONE
多重耐药菌的快速蔓延凸显了新型抗生素的迫切需求。研究人员将小分子高通量筛选(HTS)与深度学习虚拟筛选策略相结合,开发出一种名为 GNEprop 的图神经网络模型,用于预测分子的抗菌活性。研究人员首先针对敏感化的 大肠杆菌 ΔtolC 菌株筛选了约 200 万个小分子,得到数千个活性分子。随后基于这些实验数据训练 GNEprop,并验证其在分布外泛化(out-of-distribution, OOD)与活性断崖(activity cliff)预测方面的鲁棒性。进一步利用该模型在超过 14 亿个可合成化合物上进行虚拟筛选,发现 82 个在实验中表现出抗菌活性的候选分子,命中率比最初的 HTS 提高约 90 倍。值得注意的是,这些分子多数在结构上与已知抗生素差异显著,并表现出针对不同菌株的广谱或选择性效应。后续的生物学表征确认了其具体分子靶点,为抗菌药物发现提供了新的方向。

耐药性细菌的不断进化使抗生素研发面临巨大挑战。传统的药物发现依赖天然产物挖掘、高通量筛选或生物制剂定向进化等策略,但这些方法受到筛选规模、化学空间覆盖率和结构多样性不足的限制。即使当前最广泛的实验筛选也仅能覆盖约 10⁶–10⁷ 个分子,而可药用化学空间被估计高达 10⁶⁰。因而,传统方法常难以发现结构新颖的抗菌先导化合物。特别是针对革兰氏阴性菌的药物开发,更受限于其双层膜结构和高效外排系统。为突破这些瓶颈,研究人员提出结合高通量实验数据与机器学习虚拟筛选的混合策略,以在超大化学空间中高效探索潜在抗菌分子。近年来,图神经网络(GNN)在表征化学结构与预测分子性质方面表现出强大的潜力,为抗生素研究提供了新的技术基础。
方法概述
研究人员首先进行了基于表型的高通量筛选,使用约 198 万个小分子测试其对 E. coli ΔtolC 菌株的生长抑制作用,共鉴定出 5,161 个活性分子(命中率 0.26%)。基于这些实验结果,研究人员构建并训练了深度学习模型 GNEprop。该模型以图神经网络为核心,通过对分子图的层次表征实现抗菌活性预测。模型采用对比学习进行大规模自监督预训练,利用 1.22 亿个未标记分子(来自 ZINC15 数据库)学习可迁移的化学表示,再在 HTS 数据集上进行微调。
为评估模型性能,研究人员设计了严格的“骨架簇划分”(scaffold-cluster splitting)策略,以验证模型在未知化学空间中的泛化能力。随后,GNEprop 被用于对 Enamine REAL 数据库中约 14 亿个可合成分子进行虚拟筛选。
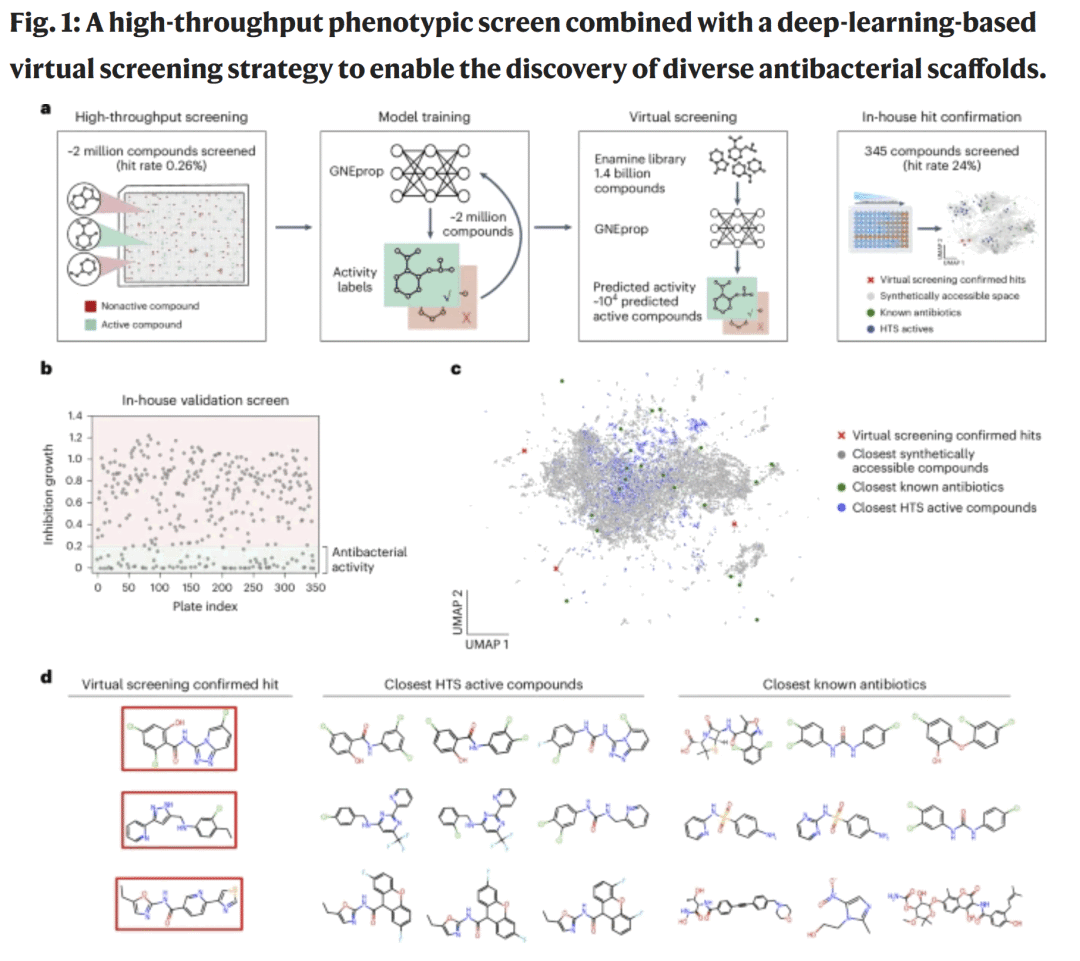
图1. 基于表型高通量筛选与深度学习虚拟筛选的联合策略示意图
结果
高通量筛选揭示丰富的结构–活性关系
HTS 数据集包含约 200 万个化合物,覆盖 18 万多个分子骨架,其中 3,200 多个骨架包含抗菌活性分子。分析发现,同一骨架内的微小结构变化即可导致活性剧烈变化,形成典型的“活性断崖”现象。
研究人员还观察到活性与分子量、疏水性等常规理化性质无显著相关性,说明模型需捕捉更深层的结构–功能关系。最终,研究团队公开了该数据的子集 GNEtolC(含 11.5 万分子、4.5 万骨架),为抗菌研究提供了目前最大规模之一的公开资源。
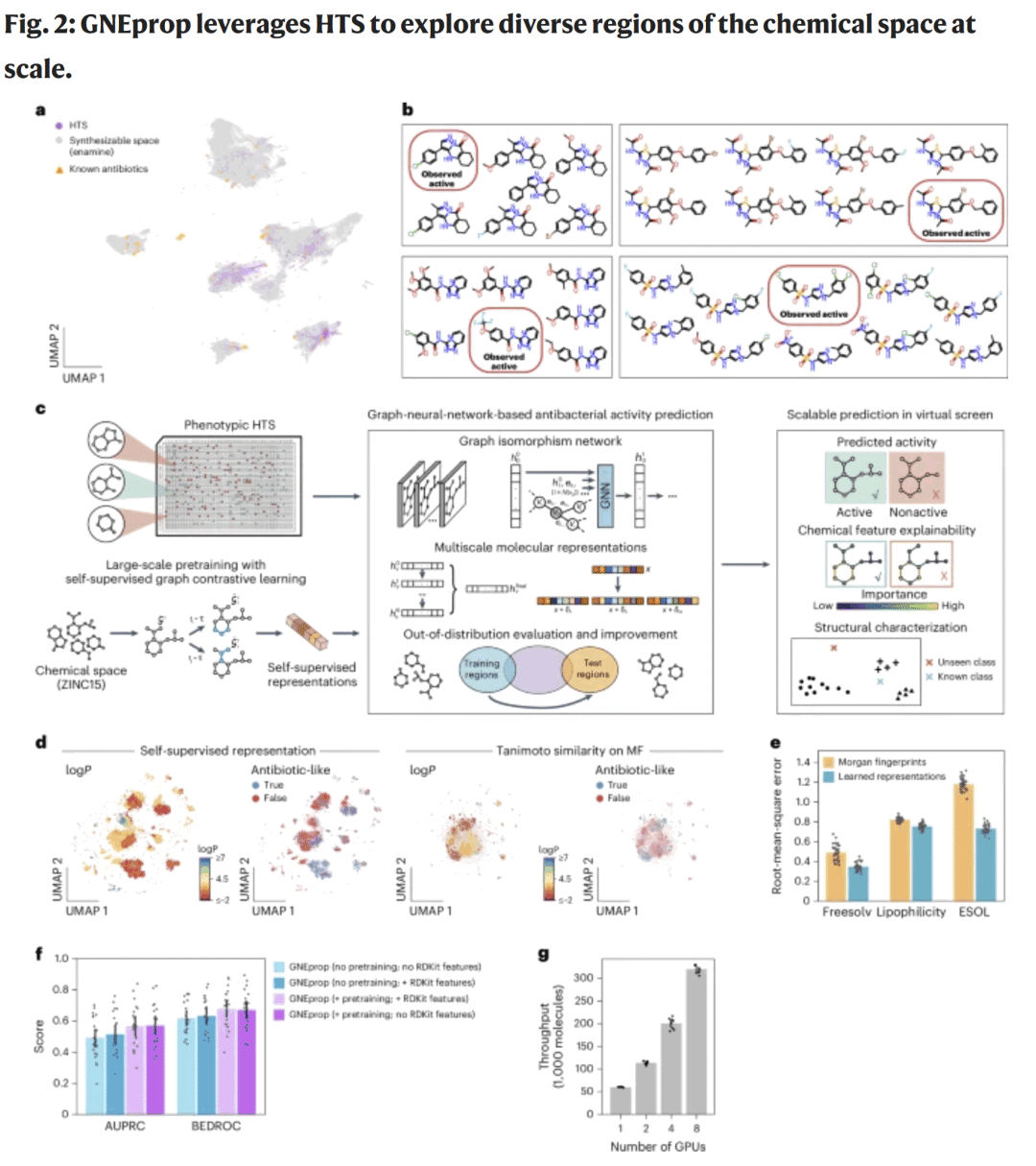
图2. HTS 数据化学空间分布与活性断崖示例
深度学习模型 GNEprop 的设计与验证
GNEprop 基于图同构网络(GIN),结合多层跳跃连接以整合局部与全局特征。模型通过自监督预训练获得具备化学意义的表示空间,这一空间可自发地按分子理化性质聚类。在多个分子性质预测任务上,GNEprop 的自监督表示显著优于传统的 Morgan 指纹。模型可在多 GPU 环境下实现高通量推理,单次可处理数十万分子,满足超大规模虚拟筛选的需求。
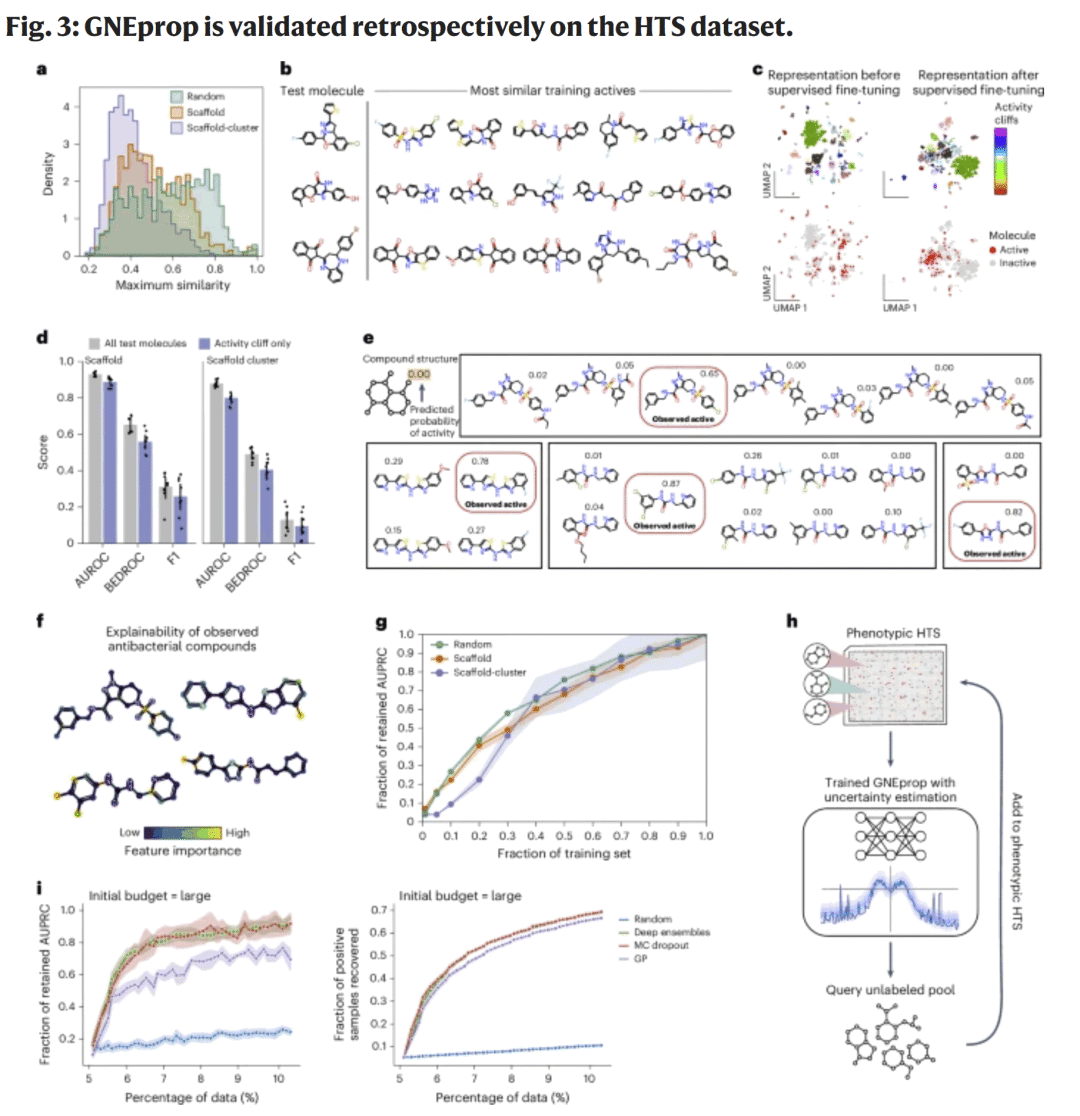
图3. GNEprop 模型架构与性能验证
模型在分布外化学空间的泛化性能
通过“骨架簇划分”,研究人员验证了模型在未知化学结构上的预测能力。GNEprop 在最严苛测试下依然保持高精度(AUROC ≈ 0.88)。在活性断崖预测中,模型能够识别结构微小差异导致的显著活性变化。结合解释性分析(integrated gradients),研究人员生成了分子结构热图,揭示模型关注的关键化学片段与药效团区域,与实际结构–活性关系高度一致。此外,研究人员发现训练数据规模与模型的泛化能力密切相关,大规模数据显著提升性能。通过主动学习(active learning)策略,模型可引导新的分子筛选以持续改进预测能力。
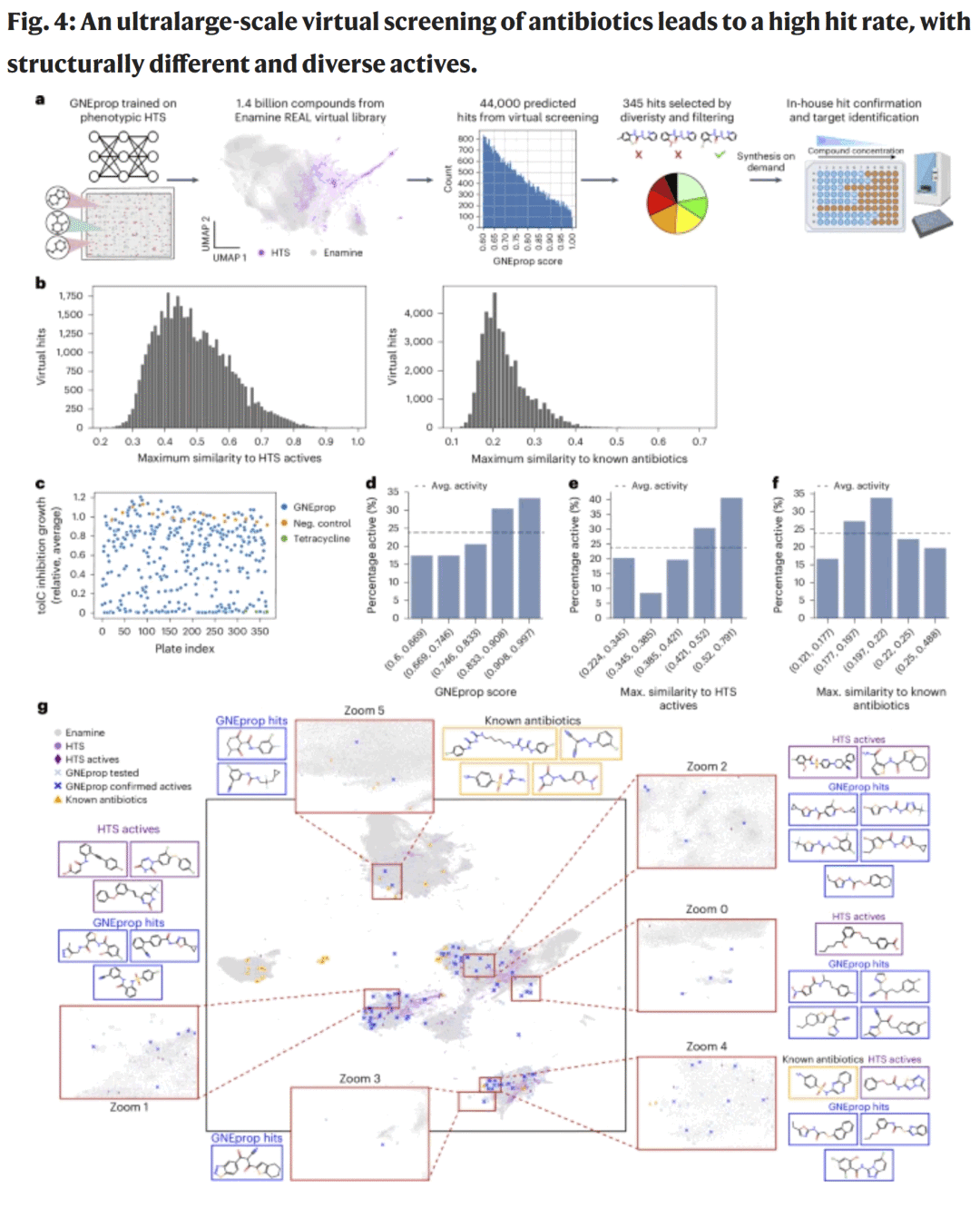
图4. 活性断崖预测与模型解释性分析
超大规模虚拟筛选与实验验证
研究人员利用 GNEprop 对 Enamine REAL 库中的 14 亿个分子进行虚拟筛选,仅用不到 48 小时即完成全部预测,筛选出 44,000 个候选分子。经过聚类与多重筛选后,选取 345 个分子合成验证,结果 82 个表现出显著抗菌活性,命中率高达 23.8%,较初始 HTS 提高约 90 倍。其中约三分之一与训练集中活性分子的结构相似度低于 0.4,绝大多数与已知抗生素差异显著,体现了模型在结构多样性和新颖性上的优势。
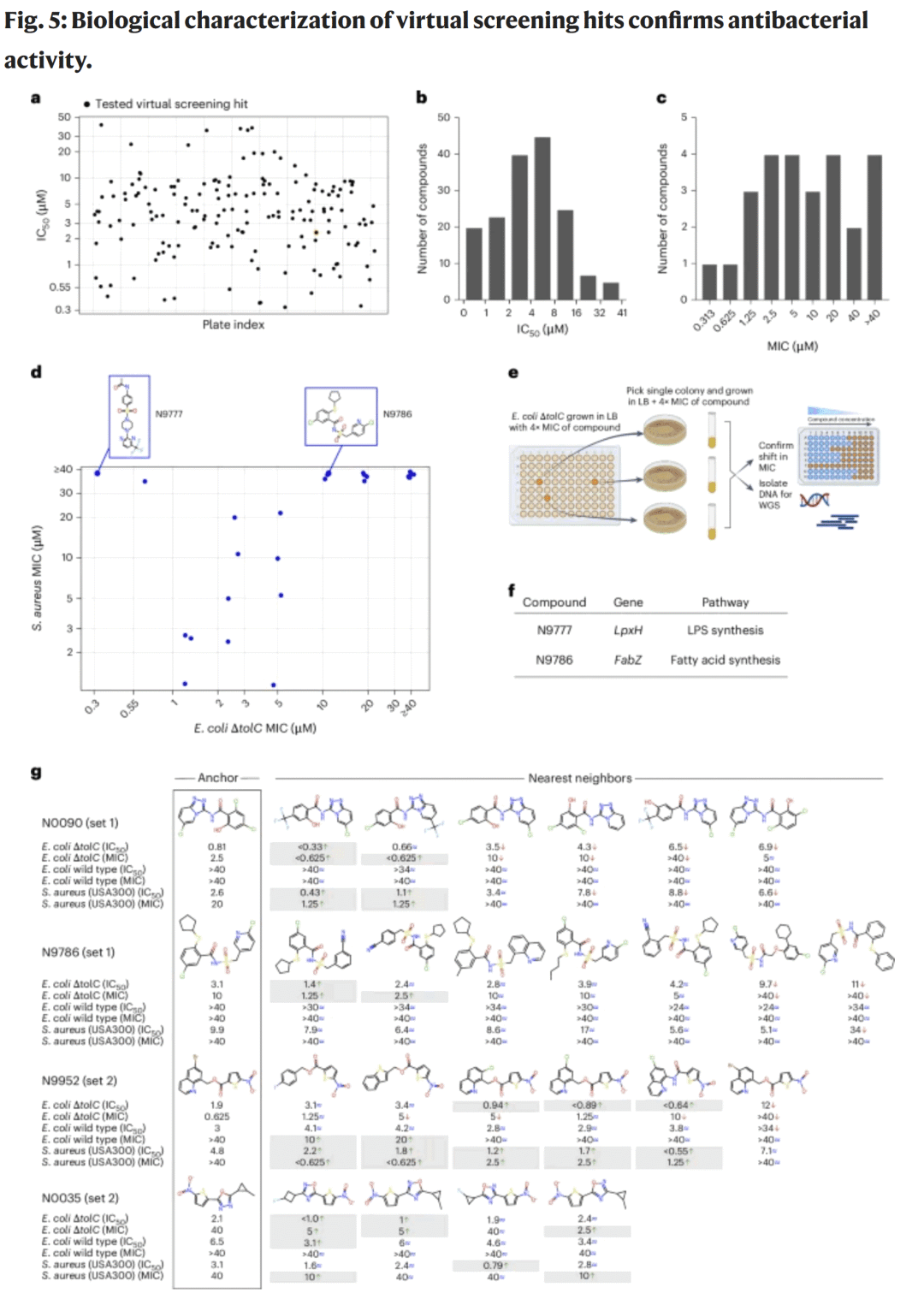
图5. 虚拟筛选流程与候选化合物分布
生物学表征与靶点验证
在 165 个候选分子的剂量–反应测试中,大多数对 ΔtolC 菌株的 IC₅₀ 低于 20 μM,部分对野生型 E. coli 及 金黄色葡萄球菌 也表现出抑菌活性。最具代表性的两个分子(N9777 与 N9786)在基因组测序中被发现分别靶向脂多糖(LPS)合成酶 LpxH 与脂肪酸合成酶 FabZ,确认了其特异性作用机制。此外,基于模型的“最近邻扩展”策略成功发现了新的活性分子,其中部分对野生型 E. coli 的 MIC 显著改善,显示出从虚拟筛选到先导优化的潜力。
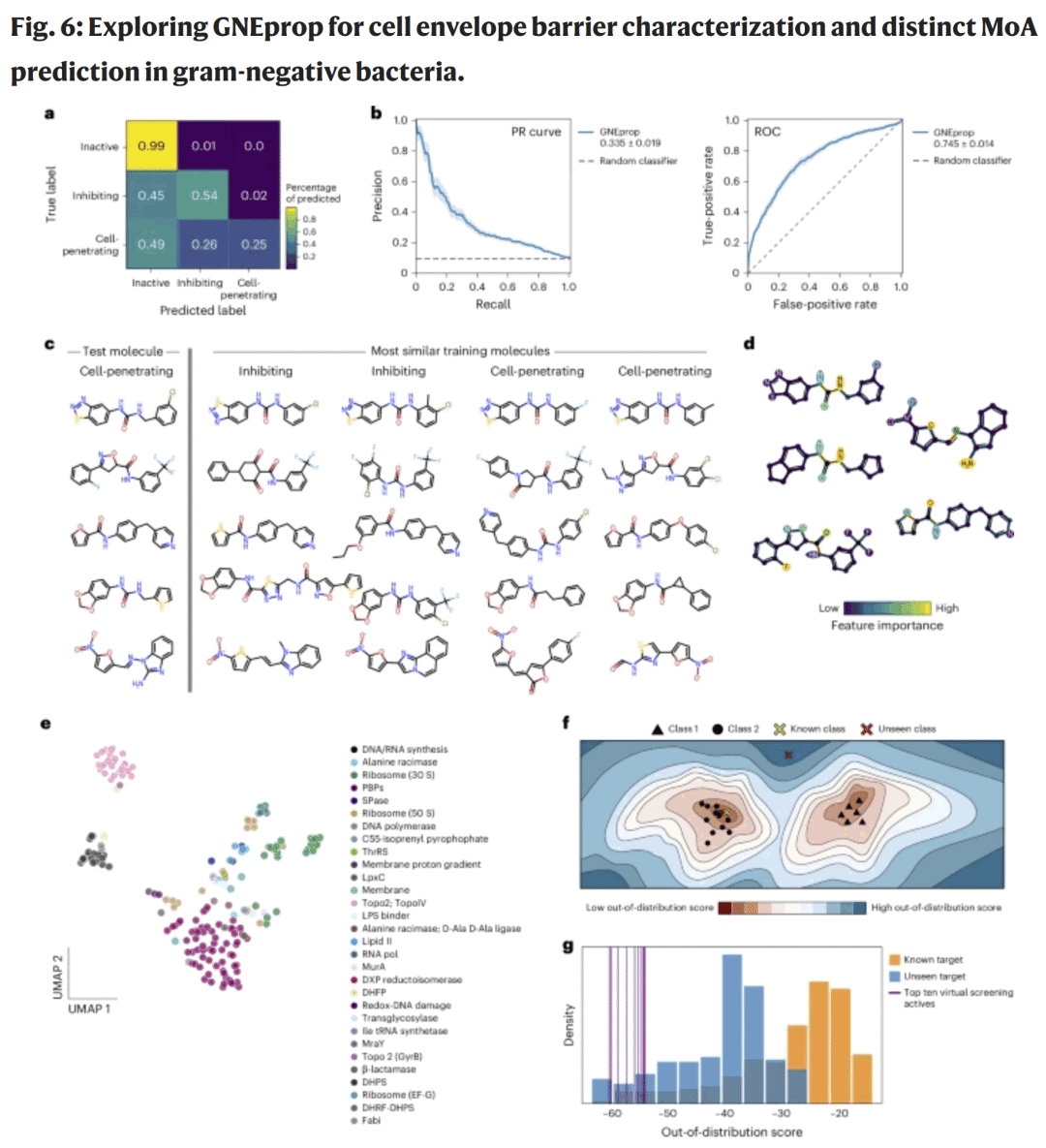
图6. 虚拟筛选命中化合物的生物学验证与靶点分析
讨论
研究结果表明,深度学习虚拟筛选能够在极大程度上拓展抗菌化合物发现的化学空间。GNEprop 模型不仅提高了命中率,还能识别与已知抗生素显著不同的新型骨架,为新作用机制(MoA)的探索提供可能。通过结合 HTS 实验数据、自监督预训练和可解释性分析,研究人员展示了机器学习在抗菌药物研发中的系统化应用路径。未来,随着更大规模的实验数据积累与模型优化,该策略有望加速新抗生素的发现与优化,缓解全球耐药性危机。
整理 | DrugOne团队
参考资料
Scalia, G., Rutherford, S.T., Lu, Z. et al. Deep-learning-based virtual screening of antibacterial compounds. Nat Biotechnol (2025).
https://doi.org/10.1038/s41587-025-02814-6
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-26,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

