ICLR 2026 | AIDD方向全景扫描
ICLR 2026 | AIDD方向全景扫描

MindDance
发布于 2026-02-09 14:53:48
发布于 2026-02-09 14:53:48
ICLR 2026 录用结果公布,共计 5356 篇论文。
对于 AIDD 领域的研究者而言,如何从海量论文中快速定位相关工作,是每年顶会季的必修课。
我们对全部接收论文进行了系统性筛选与分析,整理出 132 篇 AIDD 核心论文,覆盖蛋白质设计、分子生成、分子对接、分子动力学等主流方向。
本文将从论文分布、技术趋势、代表性工作三个维度进行深度解读。
一、筛选方法与数据来源
我们通过三层筛选策略,从 5356 篇论文中提取 AIDD 相关工作:
第一层:关键词初筛(5356 → 538)
基于 40+ 个核心关键词(molecule、protein、drug、docking、binding 等),对论文标题、摘要和关键词进行匹配。
第二层:噪声去除(538 → 210)
排除因歧义词(如 synthesis、atom)误匹配的视频生成、图像编辑等无关论文。
第三层:人工校验(210 → 132)
逐一检查边缘案例,剔除"通用图生成"、"理论证明"等与 AIDD 无直接关联的工作。
二、论文分布:蛋白质设计领跑,分子生成紧随其后
132 篇论文的主题分布如下:
方向 | 数量 | 占比 | 代表性论文 |
|---|---|---|---|
🧬 蛋白质设计与折叠 | 35 | 26.5% | SimpleFold, ProteinAE, La-Proteina |
🧪 小分子生成与优化 | 30 | 22.7% | FragFM, SynCoGen, MolEditRL |
🔗 分子对接与相互作用 | 20 | 15.2% | h-MINT, SigmaDock, PoseX |
⚛️ 分子动力学与力场 | 15 | 11.4% | ATOM, MarS-FM, BioMD |
💊 药物发现应用 | 15 | 11.4% | DrugTrail, SYNC, DeepSADR |
🧬 生物序列建模 | 10 | 7.6% | PatchDNA, AntigenLM |
🔬 其他(单细胞、病理等) | 7 | 5.3% | scDFM, HEIST |
关键洞察:
蛋白质方向论文数量超过小分子方向,这与近年来 AlphaFold 引发的蛋白质 AI 热潮密切相关。ICLR 2026 延续了这一趋势,蛋白质设计、逆折叠、全原子生成成为热门议题。
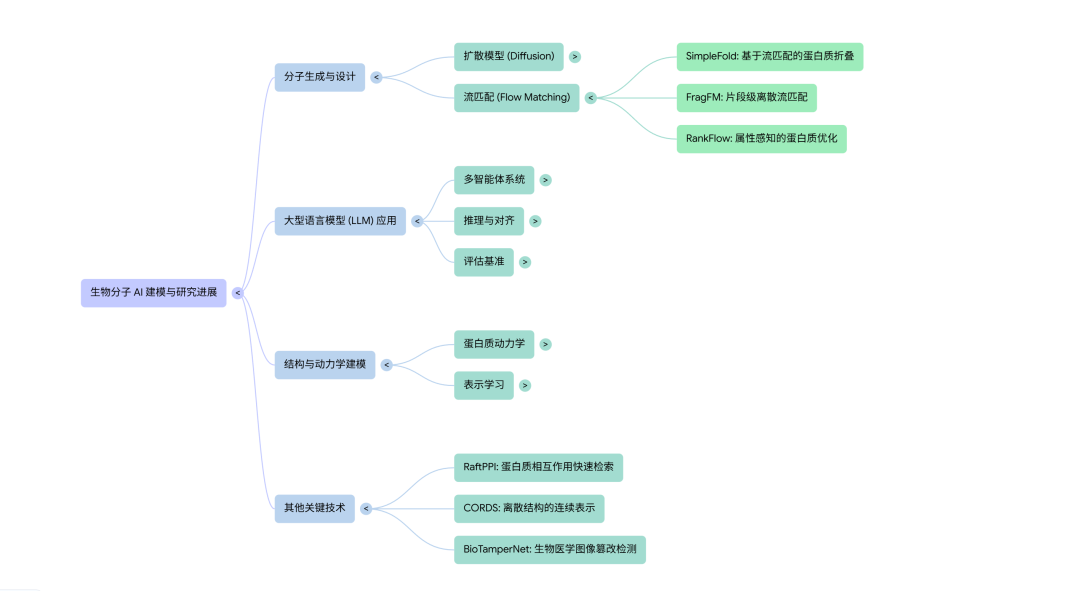
三、技术趋势:扩散模型一统江湖,Flow Matching 异军突起
3.1 扩散模型(Diffusion Models):绝对主力
约 40% 的论文采用了扩散模型,覆盖:
- 分子生成:FragFM, IAGA, MolEditRL
- 蛋白质设计:ProteinAE, Constrained Diffusion, La-Proteina
- 分子对接:SigmaDock, Pallatom-Ligand
- 分子动力学:BioMD, Geometric Graph Neural Diffusion
扩散模型的优势在于生成质量高、多样性好,但采样速度较慢。多篇论文提出了加速策略,如 FragFM 的片段级生成、DriftLite 的轻量化推理。
3.2 Flow Matching:新兴替代方案
Flow Matching 作为扩散模型的“近亲”,在本届 ICLR 有 15+ 篇论文采用:
- MarS-FM:分子动力学的马尔可夫状态建模
- La-Proteina:部分隐空间的蛋白质生成
- RankFlow:性质感知的蛋白质优化
相比扩散模型,Flow Matching 的训练和采样更高效,正在成为分子建模的新宠。
3.3 图神经网络(GNN):稳定发挥
约 25% 的论文使用了 GNN 进行分子表征:
- h-MINT:层次化分子交互网络
- TetraGT:四面体几何驱动的图 Transformer
- KGOT:知识图谱与最优传输结合
值得关注的是,SE(3) 等变网络(如 E(3)NN、EGNN)在分子建模中已成为标配,几乎所有涉及 3D 结构的工作都采用了等变架构。
3.4 大语言模型(LLM)进军化学
约 15 篇论文将 LLM 应用于化学/生物领域:
- ChemEval:LLM 化学能力评测基准
- SpectraLLM:多光谱分子结构解析
- MolLangBench:语言驱动的分子识别与编辑
- CellDuality:LLM 的生物推理能力激活
LLM 在分子理解、化学推理、结构解析等任务上展现出潜力,但在精确的结构生成任务上仍不如专用模型。
四、重点论文速览
4.1 蛋白质方向 Top 5
论文 | 亮点 |
|---|---|
SimpleFold | 简化版蛋白质折叠,证明复杂架构并非必需 |
La-Proteina | 部分隐空间 + Flow Matching 的原子级蛋白质生成 |
Constrained Diffusion | 支持硬约束的蛋白质设计 |
Multi-state Protein Design | DynamicMPNN:多状态蛋白质设计 |
Scaling Atomistic Protein Binder Design | 预训练 + 测试时计算扩展 |
4.2 分子生成 Top 5
论文 | 亮点 |
|---|---|
FragFM | 片段级离散 Flow Matching,高效分子生成 |
SynCoGen | 联合反应与坐标建模,生成可合成分子 |
MolEditRL | 扩散 + 强化学习的分子编辑 |
SYNC | 可合成性评估新基准 |
Refine Drugs | 片段精修而非从头生成 |
4.3 分子对接 Top 5
论文 | 亮点 |
|---|---|
h-MINT | 层次化分子交互网络 |
SigmaDock | 片段级 SE(3) 扩散对接 |
PoseX | 超越物理方法的交叉对接 |
SAIR | 合成数据集助力蛋白质-配体交互学习 |
Towards All-Atom Foundation Models | 全原子结合亲和力预测基础模型 |
4.4 分子动力学 Top 5
论文 | 亮点 |
|---|---|
ATOM | 多任务分子动力学预训练神经算子 |
MarS-FM | 马尔可夫状态模型 + Flow Matching |
BioMD | 全原子生物分子动力学生成模型 |
Geometric GNN Diffusion | 稳定分子动力学模拟 |
Learning Boltzmann Generators | 约束质量传输的 Boltzmann 生成器 |
五、值得关注的新基准
基准 | 任务 | 论文 |
|---|---|---|
SYNC | 分子可合成性评估 | SYNC: Measuring Synthesizability |
3DCS | 分子表征的构象敏感性 | 3DCS: Datasets and Benchmark |
GeomMotif | 蛋白质生成的几何保持能力 | GeomMotif: A Benchmark |
PepBenchmark | 多肽机器学习标准化评测 | PepBenchmark |
ChemEval | LLM 化学能力评测 | ChemEval |
六、趋势总结与展望
6.1 本届 ICLR 的 AIDD 关键词
- 全原子建模(All-Atom):从粗粒度走向原子级精度
- 可合成性(Synthesizability):生成模型开始关注实际可行性
- 预训练 + 微调:大模型范式进入分子领域
- 等变网络:SE(3) 等变已成为 3D 分子建模的标配
6.2 未来值得关注的方向
- 蛋白质-配体共设计:同时优化蛋白质和配体
- 多模态分子表征:结合序列、结构、光谱等多源信息
- 主动学习与实验闭环:AI 设计 → 实验验证 → 反馈迭代
- 可解释性:从"黑盒生成"走向"可理解设计"
七、用 NotebookLM 快速生成领域综述
面对 132 篇论文,逐篇精读不太现实。我们推荐使用 Google NotebookLM 进行快速分析。
使用方法
- 下载全部 132 篇论文 PDF(使用我们提供的脚本)
- 访问 NotebookLM,创建新笔记本
- 上传 PDF 文件(每次最多 50 个,分批上传)
- 使用结构化提示词进行提问




有一说一,总结的真不错
有一说一,总结的真不错
推荐提问流程
第一步:总体概览 → 了解主要研究方向和趋势
第二步:分主题分析 → 深入蛋白质/分子生成/对接等子领域
第三步:技术对比 → 横向比较扩散模型/Flow Matching/GNN
第四步:数据集基准 → 了解评测标准和 SOTA
第五步:未来展望 → 把握研究趋势
第六步:生成综述 → 输出完整报告
示例提示词
请根据我上传的论文,总结 ICLR 2026 AIDD 领域的主要研究方向。
哪些技术方法应用最广泛?有哪些新兴趋势?
请引用具体论文标题作为支撑。
NotebookLM 会基于上传的论文内容回答问题,并给出具体引用,适合快速建立领域全景图。
八、资源获取
完整 132 篇论文列表和筛选脚本已开源:
👉 GitHub:
https://github.com/minddance-x/ICLR2026-AIDD
仓库包含:
- 论文元数据(标题、摘要、关键词、PDF 链接)
- 筛选脚本(可复用于其他会议)
- 批量下载脚本
- NotebookLM 提示词指南
欢迎 Star 和 Fork。如有遗漏或建议,欢迎 Issue 反馈。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-30,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

