Nature子刊 | 通过深度学习预测RNA靶向CRISPR工具的靶标和脱靶活性
Nature子刊 | 通过深度学习预测RNA靶向CRISPR工具的靶标和脱靶活性

转录组工程在具有RNA靶向CRISPR效应物的活细胞中的应用取决于对目标活性和脱靶避免的准确预测。近日,《Nature Biotechnology》发表了一个卷积神经网络模型——TIGER(通过向导RNA设计靶向抑制基因表达),从引导序列和序列环境预测靶标效果。

在这项研究中,研究人员生成了一个庞大的Cas13d数据集,测量了在多个人类细胞系中约20万个引导RNA(gRNA)的活性,并进行了对Cas13d gRNA靶标和脱靶活性的全面评估:gRNA触发Cas13d核酸酶活性的能力取决于gRNA内的排列位置、核苷酸身份和靶位点背景;导致G-U摆动配对的错配比其他单碱基错配的容忍度更高。使用这个大规模数据集,研究人员训练了一个卷积神经网络——TIGER。
TIGER是什么?

TIGER是一种用于预测gRNA功效的深度学习模型,其采用了卷积神经网络(CNN)结构,通过对序列的一次性编码来预测gRNA的功效。与之前用于Cas9脱靶预测的CNN类似,TIGER有两个卷积层,随后是一个最大池化层,然后交织三个dropout和dense层,总共有两个隐藏层和一个输出层。除了之前研究中使用的结构之外,TIGER还具有以下两个架构增强:在23 nt目标位点的侧面有额外的序列背景,以及在第一个密集层中可以灵活地输入非序列特征矢量。TIGER的预测结果可以在不同的筛选模式(细胞增殖和表面标记物表达)和靶向基因(必需和非必需基因)之间泛化。
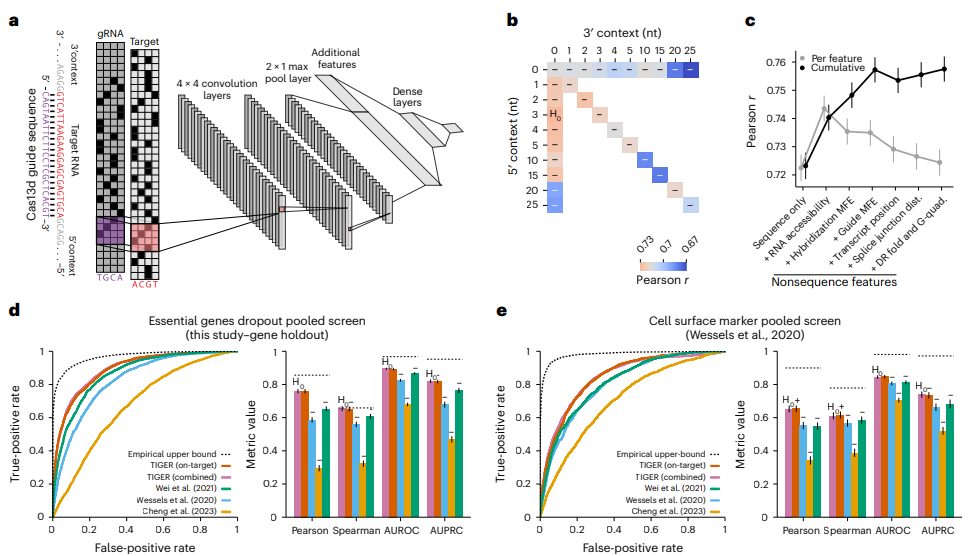
预测最佳Cas13d gRNA的深度学习模型
TIGER的性能及应用

与现有的Cas13d靶标模型(包括具有较大训练集的模型)相比,TIGER在Cas13d的靶标活性方面具有很强的性能。对于理解整个转录组的影响,TIGER模型是一个令人信服的尝试,旨在对Cas13d脱靶结合和核酸酶激活进行建模。最后,开发团队应用TIGER平台开发了一种精确和大规模并行检测基因剂量的方法。
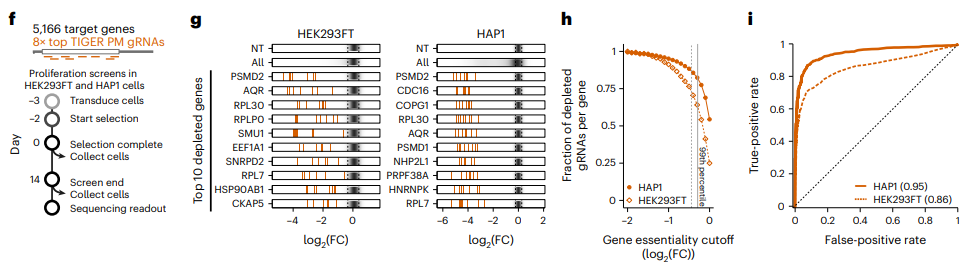
TIGER持续预测高活性的gRNA
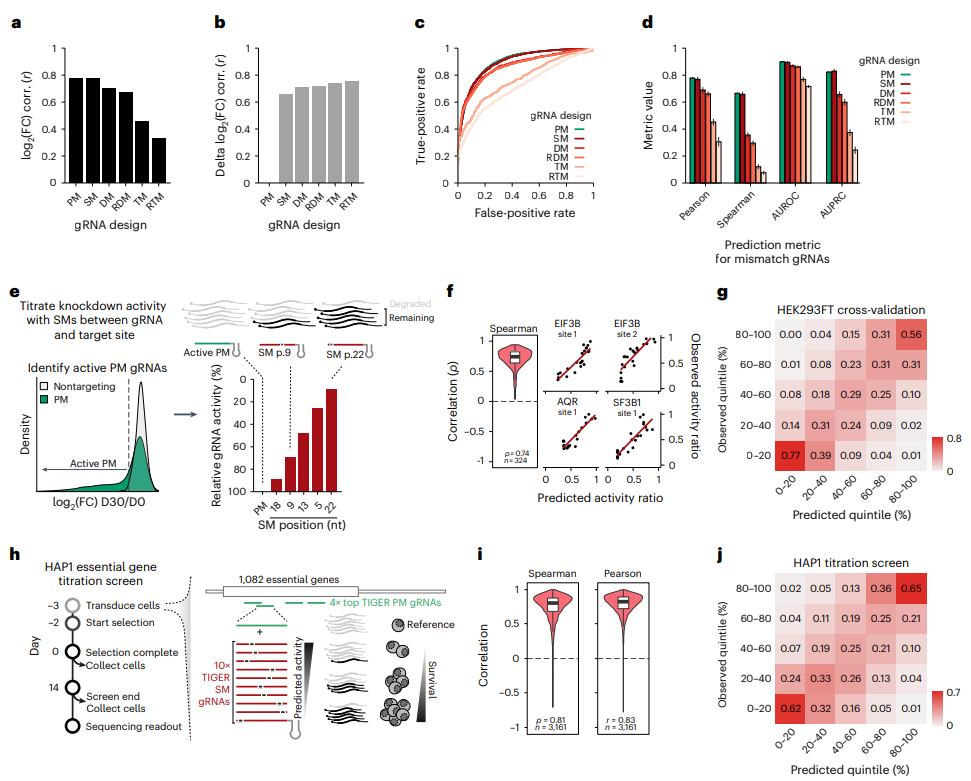
使用带错配的gRNA训练TIGER,可以预测带SMs的gRNA的脱靶活性和转录本调节作用
高精度是RNA靶向CRISPR治疗剂安全性的关键。TIGER预测将实现排序并最终避免不必要的脱靶结合位点和核酸酶激活,进一步促进RNA靶向治疗药物的开发。区分密切相关的靶位点的能力可能使我们能够靶向等位基因变异和其他几乎不可药用的靶点,如融合基因产物。此外,我们的模型可以用于大规模精确调节靶RNA敲除。
TIGER模型代码已存入Github(👉 点击阅读原文访问):https://github.com/daklab/tiger.
// 参考文献
Wessels H H, Stirn A, Méndez-Mancilla A, et al. Prediction of on-target and off-target activity of CRISPR–Cas13d guide RNAs using deep learning[J]. Nature Biotechnology, 2023: 1-10.
