单细胞RNA测序分析人高级别浆液性卵巢癌的组织结构
单细胞RNA测序分析人高级别浆液性卵巢癌的组织结构
生信技能树jimmy
发布于 2024-01-02 17:01:25
发布于 2024-01-02 17:01:25
文章概述
文章标题:《Single-Cell RNA Sequencing Reveals the Tissue Architecture in Human High-Grade Serous Ovarian Cancer》
发表日期和杂志:2022年发表在Clinical Cancer Research上
在线阅读链接:https://doi.org/10.1158%2F1078-0432.CCR-22-0296
之前整理过单细胞测序分析进展性高级别浆液性卵巢癌核心原型表型的演变,这篇文章分析了人高级别浆液性卵巢癌的组织结构
实验设计
为了系统地研究HGSOC的肿瘤内异质性,采用深层单细胞RNA测序技术(scRNA-seq)对7例初治HGSOC早期和晚期患者及5例年龄匹配的非恶性卵巢组织标本进行肿瘤分析。
共获得59,324个HGSOC和非恶性卵巢组织的单细胞,其中,33264个细胞(56%)来⾃HGSOC肿瘤患者组织,26060个(44%)来⾃⾮恶性卵巢组织标本。
单细胞转录组数据情况
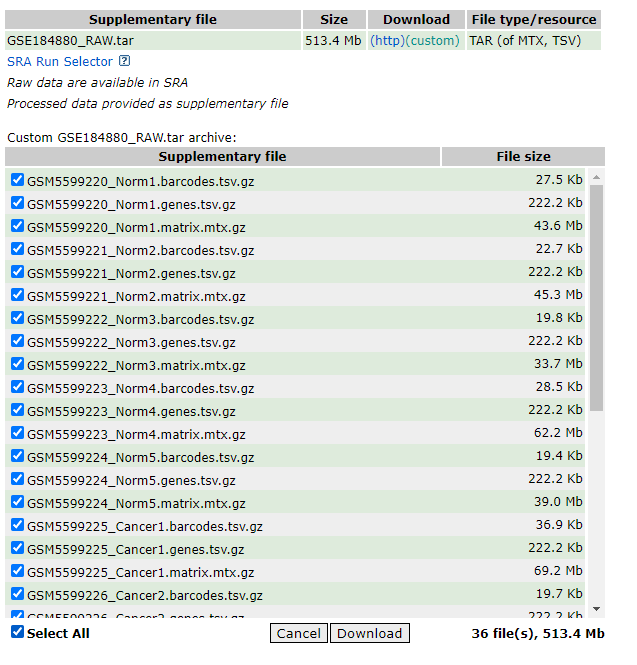
数据链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE184880
GSM5599220 Normal ovarian tissue1
GSM5599221 Normal ovarian tissue2
GSM5599222 Normal ovarian tissue3
GSM5599223 Normal ovarian tissue4
GSM5599224 Normal ovarian tissue5
GSM5599225 Cancer_HGSOC1
GSM5599226 Cancer_HGSOC2
GSM5599227 Cancer_HGSOC3
GSM5599228 Cancer_HGSOC4
GSM5599229 Cancer_HGSOC5
GSM5599230 Cancer_HGSOC6
GSM5599231 Cancer_HGSOC7
提供了10X标准格式的三个文件,不过我们直接下载后按照数据集整理对应的文件,使用Read10X()函数读取即可。
数据整理:下载数据,然后按照数据集名称对应的整理
fs=list.files('./','features.tsv.gz')
fs
samples1=gsub('-features.tsv.gz','',fs)
samples1
library(stringr)
samples2= gsub('-filtered','',samples1)
samples2
lapply(1:length(samples2), function(i){
x=samples2[i]
y=samples1[i]
dir.create(x,recursive = T)
file.copy(from=paste0(y,'-features.tsv.gz'),
to=file.path(x, 'features.tsv.gz' ))
file.copy(from=paste0(y,'-matrix.mtx.gz'),
to= file.path(x, 'matrix.mtx.gz' ) )
file.copy(from=paste0(y,'-barcodes.tsv.gz'),
to= file.path(x, 'barcodes.tsv.gz' ))
})
读取数据,创建seurat结构进行后续的分析。
###### step1:导入数据 ######
dir='GSE184880_RAW/outputs/'
samples=list.files( dir )
samples
# samples = head(samples,10)
sceList = lapply(samples,function(pro){
# pro=samples[1]
print(pro)
sce =CreateSeuratObject(counts = Read10X(file.path(dir,pro )) ,
project = gsub('^GSM[0-9]*_','',pro) ,
min.cells = 5,
min.features = 500 )
return(sce)
})
names(sceList)
# gsub('^GSM[0-9]*','',samples)
sce.all=merge(x=sceList[[1]],
y=sceList[ -1 ],
add.cell.ids = gsub('_gene_cell_exprs_table.txt.gz','',gsub('^GSM[0-9]*_','',samples) ) )
as.data.frame(sce.all@assays$RNA$counts[1:10, 1:2])
head(sce.all@meta.data, 10)
table(sce.all$orig.ident)
# 确认分组信息,通常是隐含在文件名,样品名字里面
sce.all$group=toupper( substring(colnames(sce.all),1,4))
table(sce.all@meta.data$group)
table(sce.all@meta.data$orig.ident)
在批量读取数据之前,一般要先读取一个数据用来进行测试,确认无误后,再使用循环批量读取全部的数据用于后续的分析。
后面就是标准分析啦,对读取进来的数据进行质控、harmony整合以及细分亚群定义等。
第一层次降维聚类分群
使用UMAP方法对细胞进行可视化,并根据建立的基因标记表进行注释
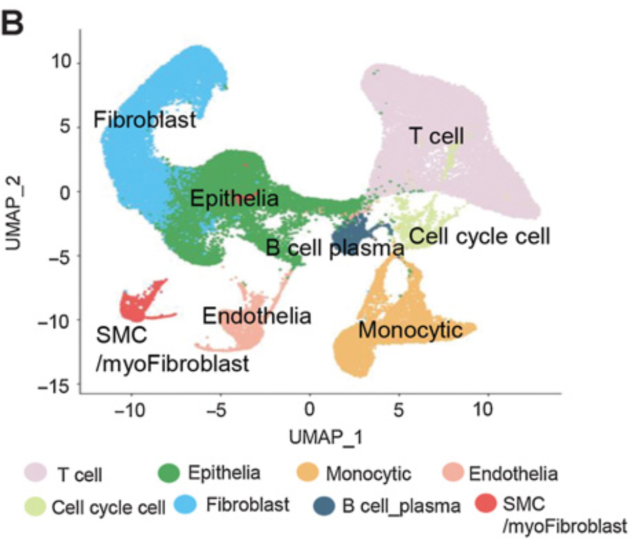
- t细胞(以CD3D、CD3E和CD8A为标记)
- 上皮细胞(以KRT18、EPCAM、CD24和KRT19为标记)
- 单核细胞(CD14和C1QA)
- 内皮细胞(PECAM1和CLDN5)
- 细胞周期细胞(MKI67和TOP2A)
- 成纤维细胞(DCN和OGN)
- B细胞和浆细胞类型(CD79A和JCHAIN)
- 平滑肌细胞/肌成纤维细胞(以ACTA2、MYH11和TAGLN为标记)

HGSOC肿瘤和非恶性卵巢的细胞亚群类型相似,但细胞分布有很大不同。例如,非恶性卵巢以成纤维细胞为主,这与老年卵巢纤维化的增加相一致,而肿瘤中含有更多的T细胞或上皮细胞。
将细胞团与临床分期等肿瘤特征联系起来,发现T细胞团的比例随着肿瘤分期的进展而降低

其它加分项
生态系统上皮细胞在不同肿瘤分期的特征
在所有组织中共收集14636个卵巢上⽪细胞,并将其分为12个簇,包括8192个来⾃HGSOC肿瘤的细胞和6444个来⾃⾮恶性卵巢组织的细胞。
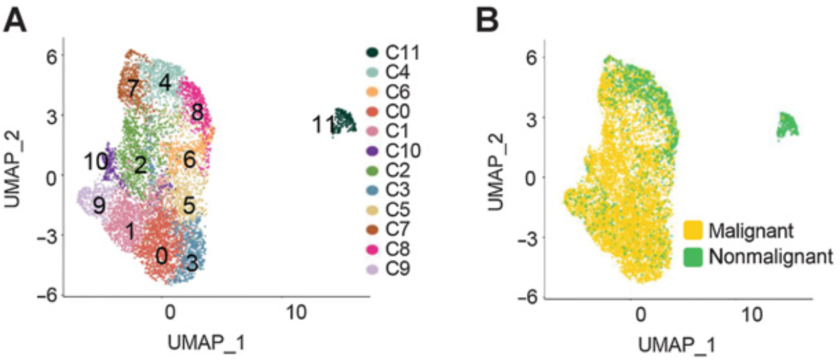
应⽤CytoTRACE预测这些上⽪细胞的分化状态,并识别HGSOC中的静⽌⼲细胞。
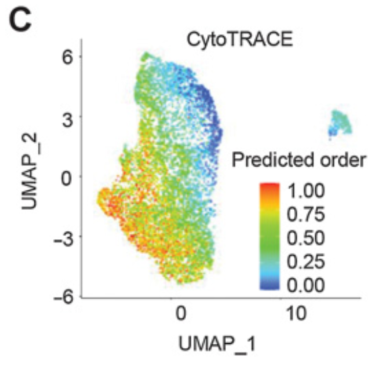
将Monocle算法应⽤于恶性上⽪细胞的拟时分析,以预测上⽪细胞在肿瘤发展中的发展轨迹。

使⽤TCGA和GEO在线分析和可⽤的OS结果评估了TCGA HGSOC数据集、GEO HGSOC数据集和两个浆液性卵巢癌数据集,检查EMT标记物与患者⽣存率的相关性。

分析表明,四个基因包括NOTCH1、SNAI2、WNT11和TGFBR1在这四个批量表达数据集中的⾄少三个队列中与不良预后显著相关。
文章还对基质间充质⼲细胞的多样性和CAFs的特征进行了分析,通过对M1巨噬细胞的富集表明HGSOC早期预后良好
文章小结
- 详细描述了早期和晚期原发性HGSOC肿瘤的全⾯单细胞转录组图谱。
- 利⽤拟时分析描述不同肿瘤分期的上⽪细胞发育层次,并鉴定EMT的特性。
- 发现免疫共抑制受体TIGTI阻断剂可抑制ID8来源的C57BL/6⼩⿏模型的肿瘤⽣⻓。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-12-29,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

