Snakemake — 可重复数据分析框架
工欲善其事必先利其器
1Snakemake
Snakemake是一款流行的生物信息学工作流管理系统,由Johannes Köster及其团队开发。它旨在降低复杂数据分析的复杂性,使生物信息学工作流的创建和执行变得更加容易和可重复。Snakemake的设计灵感来自于Makefile,但它是专门为生物信息学和数据密集型科学工作流设计的,使用Python语言进行工作流的定义,这使得它在生物信息学社区中特别受欢迎。
Snakemake的主要优势包括:
- 易于使用和学习:Snakemake使用简单的、基于Python的语法来定义工作流,这使得它对于具有Python基础的科学家来说非常容易上手。
- 灵活性:Snakemake允许用户以模块化和可重复的方式定义数据分析步骤,易于修改和重用。
- 可扩展性:它可以在各种计算环境中运行,从单个计算机到高性能计算集群,甚至是云环境。Snakemake能够自动化地处理任务分发和并行化,优化资源使用。
- 可重复性:通过使用容器技术(如Docker和Singularity)和Conda环境,Snakemake支持高度可重复的科学分析,确保不同环境下的分析结果一致。
- 集成性:Snakemake可以轻松地与其他生物信息学工具和语言集成,如R和Python,使得复杂分析的步骤更加灵活。
- 社区支持:Snakemake有一个活跃的社区,提供大量的文档、教程和案例,帮助用户学习如何有效使用它。
官网:https://snakemake.github.io/ 文档:https://snakemake.readthedocs.io/en/stable/ github:https://github.com/snakemake

2发表文章
Johannes Köster及其团队在多个场合发表了关于Snakemake的文章,展示了其如何促进科学研究的可重复性和高效性。其中最初的论文是:
题目: Sustainable data analysis with Snakemake 期刊:Bioinformatics 日期:2012/10/1 作者&单位:Johannes Köster & 杜伊斯堡-埃森大学 DOI: 10.1093/bioinformatics/bts480

题目:Sustainable data analysis with Snakemake 期刊:F1000Research DOI:https://doi.org/10.12688/f1000research.29032.2 滚动更新,介绍Snakemake的设计理念、特性以及如何在生物信息学和数据分析中有效应用它,展示了Snakemake确保数据分析可持续性的能力
3如何安装
推荐使用 conda/mamba 安装,简单快捷
## 安装
mamba create -c conda-forge -c bioconda -n snakemake snakemake
## 检查
mamba activate snakemake
snakemake --help
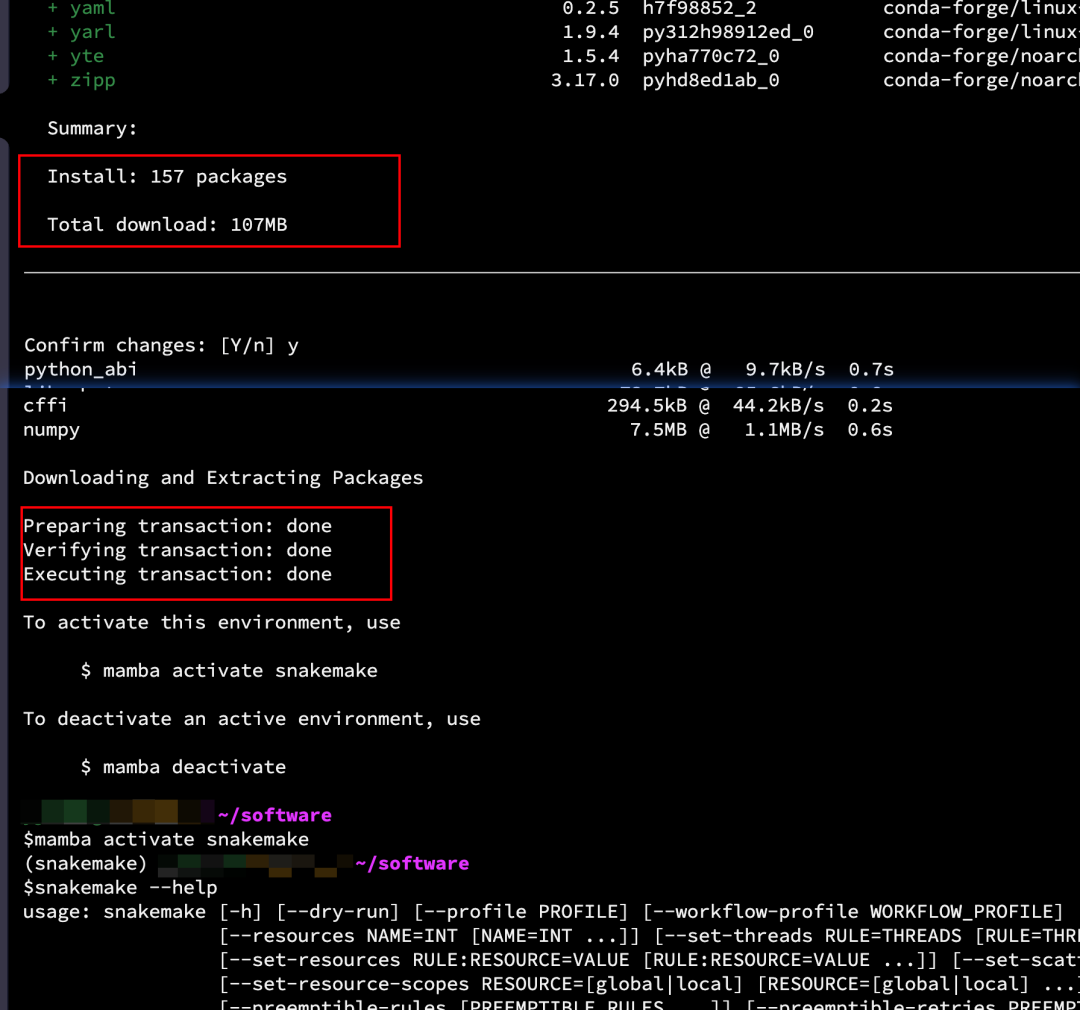
安装完成
4功能简述
Snakemake是一个工作流管理系统,旨在简化和自动化数据分析流程。它允许用户通过简单的Python语法定义分析步骤,管理数据和代码的依赖性。Snakemake支持灵活的规则定义,可以轻松地适应各种计算环境,包括单机、集群和云。它特别强调可重复性和透明性,通过整合软件环境和容器技术,确保分析结果的一致性。此外,Snakemake还支持并行执行和错误处理,使得大规模数据分析更高效、更可靠。
snakemake 的基本组成单位叫“规则”,即 rule;每个 rule 里面又有多个元素(input、output、run等)。工作流是根据规则定义的,这些规则定义了如何从输入文件创建输出文件。规则之间的依赖关系是自动确定的,从而创建可以自动并行化的作业的 DAG(有向无环图)。
5最小化使用
准备工作
## 创建工作目录
mkdir snakemake-tutorial
cd snakemake-tutorial
## 下载示例数据
curl -L https://api.github.com/repos/snakemake/snakemake-tutorial-data/tarball -o snakemake-tutorial-data.tar.gz
tar -xf snakemake-tutorial-data.tar.gz
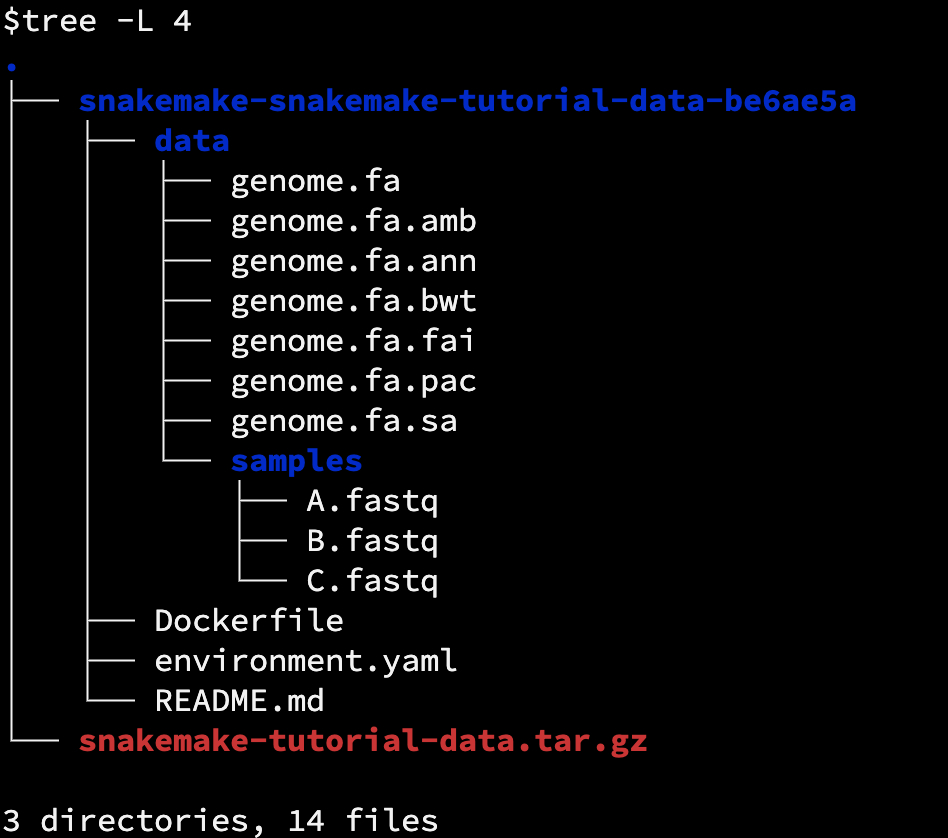
示例数据
## 创建一个测试环境
mamba env create --name snakemake-tutorial --file environment.yaml
## 如果pip安装报错,可以提前设置一下 pip 镜像
pip config set global.index-url https://mirrors.bfsu.edu.cn/pypi/web/simple
## 激活环境
conda activate snakemake-tutorial
snakemake --help
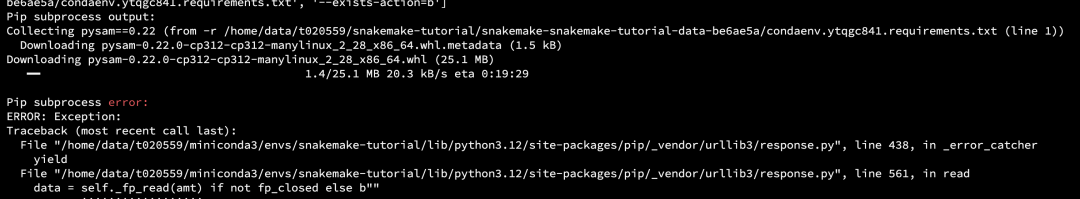
pip安装报错
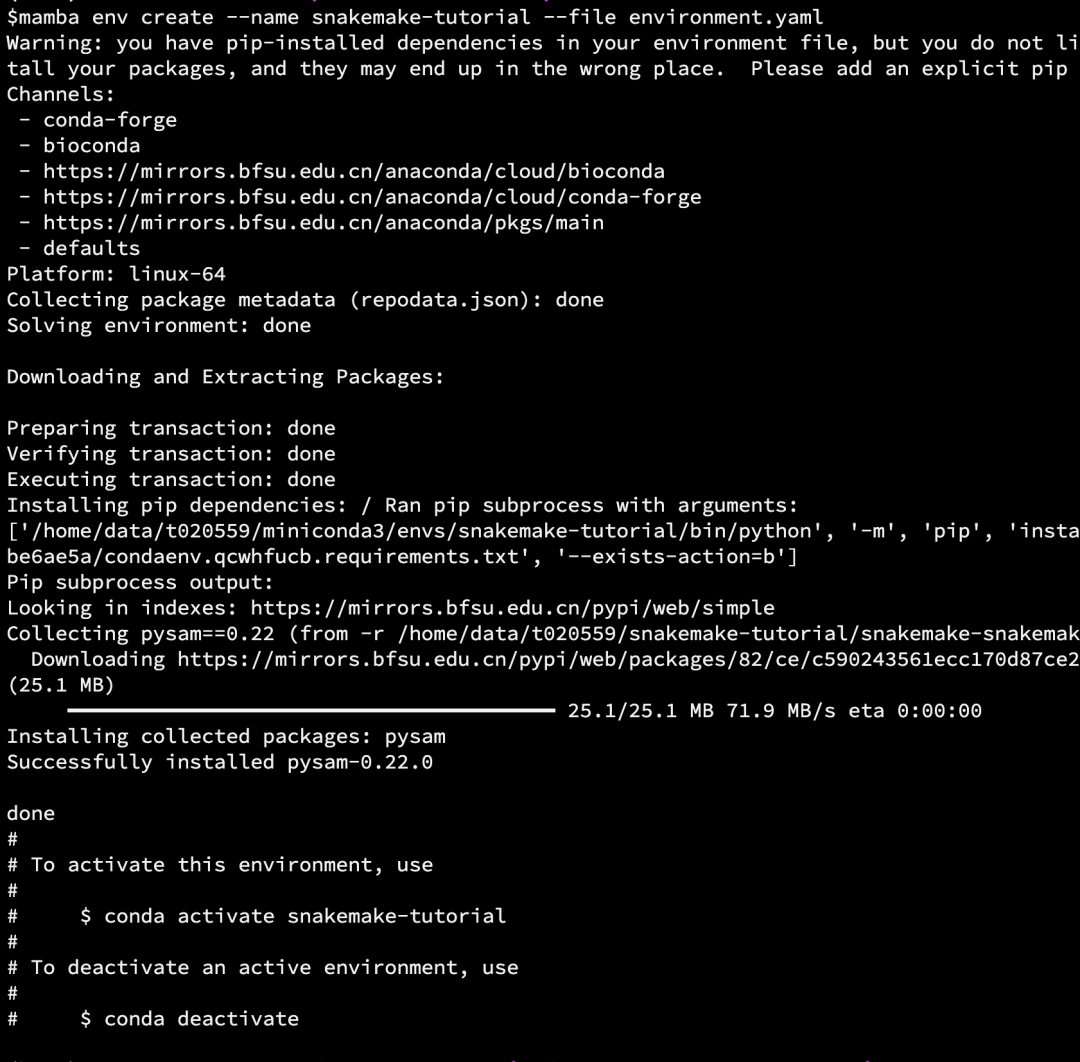
设置镜像后,成功安装
一个简单的 call snp 的示例
##激活环境
conda activate snakemake-tutorial
## 新建一个snakefile
vim snakefile
#####写入以下内容
SAMPLES = ["A", "B"]
rule all:
input:
"plots/quals.svg"
rule bwa_map:
input:
"data/genome.fa",
"data/samples/{sample}.fastq"
output:
"mapped_reads/{sample}.bam"
shell:
"bwa mem {input} | samtools view -Sb - > {output}"
rule samtools_sort:
input:
"mapped_reads/{sample}.bam"
output:
"sorted_reads/{sample}.bam"
shell:
"samtools sort -T sorted_reads/{wildcards.sample} "
"-O bam {input} > {output}"
rule samtools_index:
input:
"sorted_reads/{sample}.bam"
output:
"sorted_reads/{sample}.bam.bai"
shell:
"samtools index {input}"
rule bcftools_call:
input:
fa="data/genome.fa",
bam=expand("sorted_reads/{sample}.bam", sample=SAMPLES),
bai=expand("sorted_reads/{sample}.bam.bai", sample=SAMPLES)
output:
"calls/all.vcf"
shell:
"bcftools mpileup -f {input.fa} {input.bam} | "
"bcftools call -mv - > {output}"
rule plot_quals:
input:
"calls/all.vcf"
output:
"plots/quals.svg"
script:
"scripts/plot-quals.py"
input 定义输入文件
output 定义输出文件
shell 程序运行的shell命令
script 自定义脚本
注意: 1、 输入或输出项之间要有逗号。这是由于 Python 会连接后续字符串,如果没有逗号分割,可能会导致意外行为 2、如果一个规则有多个输出文件,Snakemake 会要求它们全部输出 ,在使用通配符的时候应避免出现完全相同的通配,否则,可能会发生两个工作 并行运行同一规则想要写入同一文件 3、在shell 命令中,我们可以将字符串分成多行,Python 会自动将它们连接成一行。这是一种方便的模式,可以避免 shell 命令行过长。使用它时,请确保每行都有一个尾随空格,但最后一行除外, 以避免参数没有正确分开
$cat plot-quals.py
import matplotlib
matplotlib.use("Agg")
import matplotlib.pyplot as plt
from pysam import VariantFile
quals = [record.qual for record in VariantFile(snakemake.input[0])]
plt.hist(quals)
plt.savefig(snakemake.output[0])
测试流程是否能跑通
## 在snakefile所在的目录下,执行以下命令
snakemake -np > test.log
-p:#打印运行的shell命令
-n:#只展示需要完成的步骤,不运行
$cat test.log
Building DAG of jobs...
Job stats:
job count
-------------- -------
all 1
bcftools_call 1
bwa_map 2
plot_quals 1
samtools_index 2
samtools_sort 2
total 9
Execute 2 jobs...
[Tue Mar 19 21:52:18 2024]
localrule bwa_map:
input: data/genome.fa, data/samples/B.fastq
output: mapped_reads/B.bam
jobid: 6
reason: Missing output files: mapped_reads/B.bam
wildcards: sample=B
resources: tmpdir=<TBD>
bwa mem data/genome.fa data/samples/B.fastq | samtools view -Sb - > mapped_reads/B.bam
[Tue Mar 19 21:52:18 2024]
localrule bwa_map:
input: data/genome.fa, data/samples/A.fastq
output: mapped_reads/A.bam
jobid: 4
reason: Missing output files: mapped_reads/A.bam
wildcards: sample=A
resources: tmpdir=<TBD>
bwa mem data/genome.fa data/samples/A.fastq | samtools view -Sb - > mapped_reads/A.bam
Execute 2 jobs...
[Tue Mar 19 21:52:18 2024]
localrule samtools_sort:
input: mapped_reads/B.bam
output: sorted_reads/B.bam
jobid: 5
reason: Missing output files: sorted_reads/B.bam; Input files updated by another job: mapped_reads/B.bam
wildcards: sample=B
resources: tmpdir=<TBD>
samtools sort -T sorted_reads/B -O bam mapped_reads/B.bam > sorted_reads/B.bam
[Tue Mar 19 21:52:18 2024]
localrule samtools_sort:
input: mapped_reads/A.bam
output: sorted_reads/A.bam
jobid: 3
reason: Missing output files: sorted_reads/A.bam; Input files updated by another job: mapped_reads/A.bam
wildcards: sample=A
resources: tmpdir=<TBD>
samtools sort -T sorted_reads/A -O bam mapped_reads/A.bam > sorted_reads/A.bam
Execute 2 jobs...
[Tue Mar 19 21:52:18 2024]
localrule samtools_index:
input: sorted_reads/B.bam
output: sorted_reads/B.bam.bai
jobid: 8
reason: Missing output files: sorted_reads/B.bam.bai; Input files updated by another job: sorted_reads/B.bam
wildcards: sample=B
resources: tmpdir=<TBD>
samtools index sorted_reads/B.bam
[Tue Mar 19 21:52:18 2024]
localrule samtools_index:
input: sorted_reads/A.bam
output: sorted_reads/A.bam.bai
jobid: 7
reason: Missing output files: sorted_reads/A.bam.bai; Input files updated by another job: sorted_reads/A.bam
wildcards: sample=A
resources: tmpdir=<TBD>
samtools index sorted_reads/A.bam
Execute 1 jobs...
[Tue Mar 19 21:52:18 2024]
localrule bcftools_call:
input: data/genome.fa, sorted_reads/A.bam, sorted_reads/B.bam, sorted_reads/A.bam.bai, sorted_reads/B.bam.bai
output: calls/all.vcf
jobid: 2
reason: Missing output files: calls/all.vcf; Input files updated by another job: sorted_reads/A.bam, sorted_reads/B.bam.bai, sorted_reads/B.bam, sorted_reads/A.bam.bai
resources: tmpdir=<TBD>
bcftools mpileup -f data/genome.fa sorted_reads/A.bam sorted_reads/B.bam | bcftools call -mv - > calls/all.vcf
Execute 1 jobs...
[Tue Mar 19 21:52:18 2024]
localrule plot_quals:
input: calls/all.vcf
output: plots/quals.svg
jobid: 1
reason: Missing output files: plots/quals.svg; Input files updated by another job: calls/all.vcf
resources: tmpdir=<TBD>
Execute 1 jobs...
[Tue Mar 19 21:52:18 2024]
localrule all:
input: plots/quals.svg
jobid: 0
reason: Input files updated by another job: plots/quals.svg
resources: tmpdir=<TBD>
Job stats:
job count
-------------- -------
all 1
bcftools_call 1
bwa_map 2
plot_quals 1
samtools_index 2
samtools_sort 2
total 9
Reasons:
(check individual jobs above for details)
input files updated by another job:
all, bcftools_call, plot_quals, samtools_index, samtools_sort
output files have to be generated:
bcftools_call, bwa_map, plot_quals, samtools_index, samtools_sort
This was a dry-run (flag -n). The order of jobs does not reflect the order of execution.
测试检查无误后,即可提交后台运行
nohup snakemake --cores 4 --keep-going 1>snak.log 2>&1 &
-cores #设定可调用的核数
--keep-going ##如果某一个任务有报错,与其没有依赖关系的任务可以继续跑
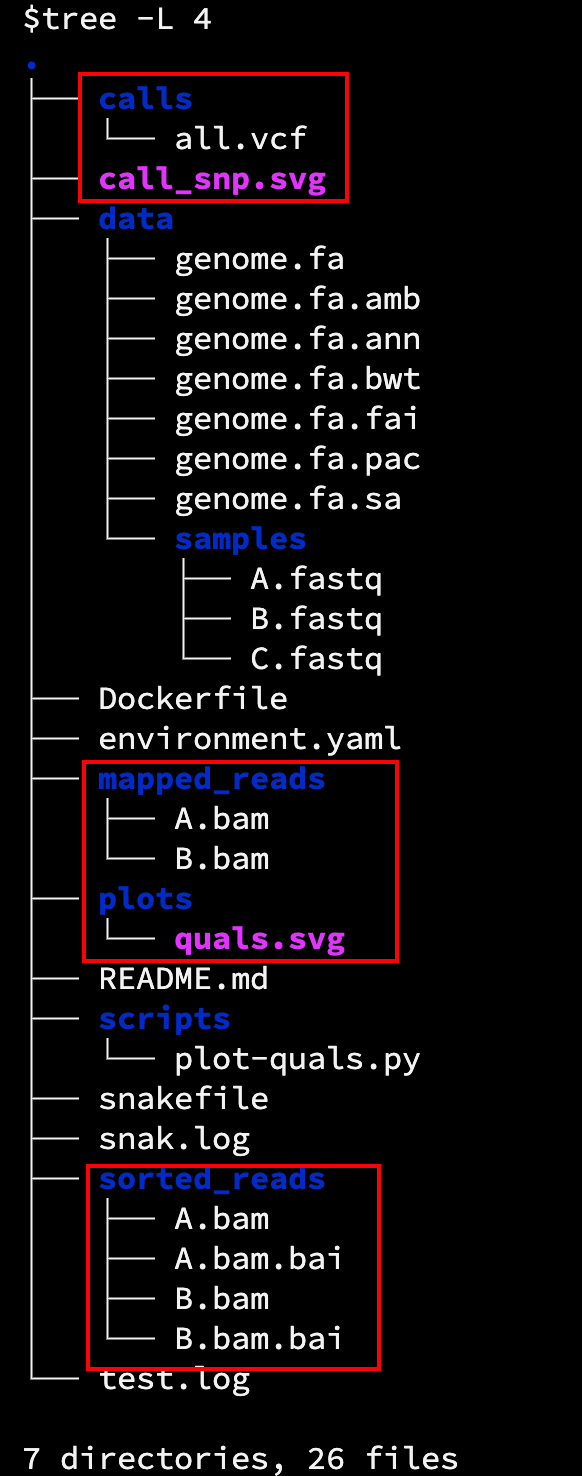
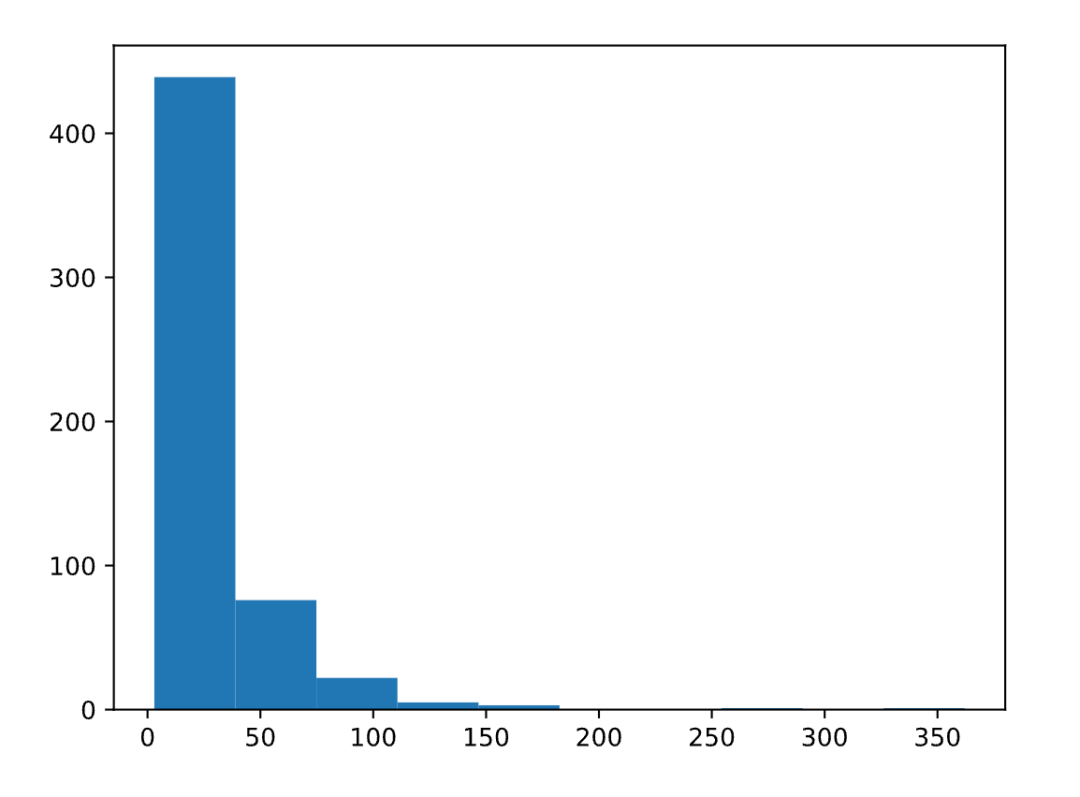
结果图:quals.svg
可视化工作流
snakemake --dag plots/quals.svg |dot -Tsvg >call_snp.svg
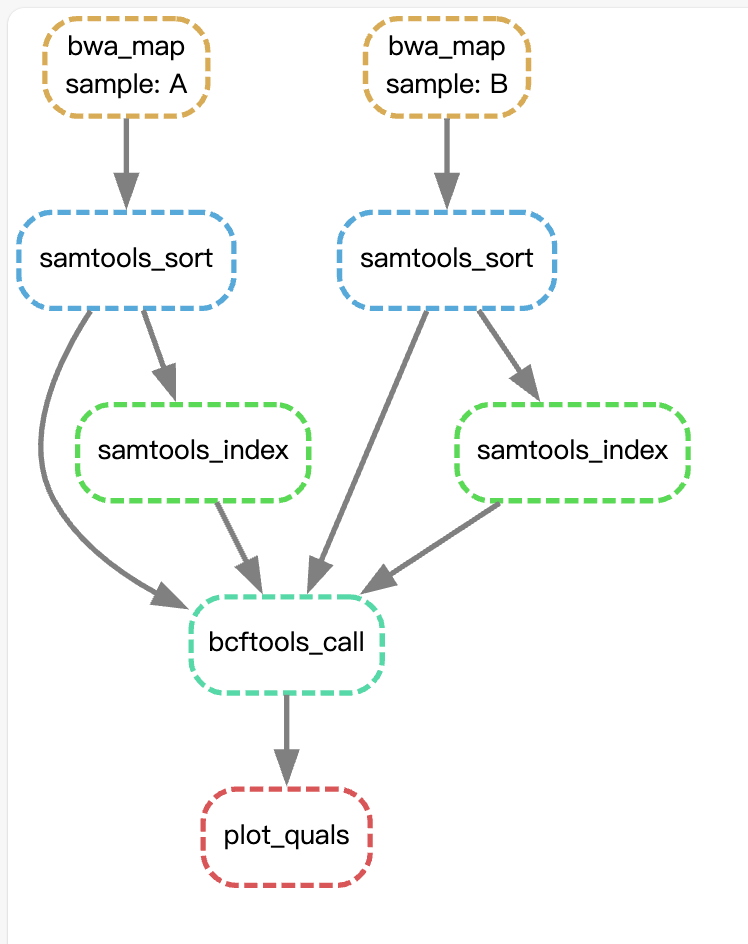
call_snp.svg
更多用法详见:https://snakemake.readthedocs.io/en/stable/index.html
参考:
- https://snakemake.readthedocs.io/en/stable/index.html
- http://www.biomarker.com.cn/archives/19623
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-03-19,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

