Nat Commun|基于化学微扰转录组自监督表征学习的药物筛选
基于表型的药物筛选方法关注化学分子在细胞、组织、整体动物水平上的响应,可以提供对疾病机制的全面理解,在药物研发中发挥重要作用。化学微扰转录组能够提供对药物作用机制更全面的理解,但数据中固有噪声往往掩盖了真正的扰动信号,从中提取有意义的信息具有挑战性,阻碍了化学微扰转录组在药物筛选中的应用。
中国科学院上海药物研究所郑明月课题组提出了基于自监督表征学习的深度生成模型TranSiGen(Transcriptional Signatures Generator),学习化学微扰转录组表征用于药物表型筛选。该模型从细胞基因表达和分子结构以高精度预测化学微扰转录组,相应的微扰表征在基于配体的虚拟筛选、药物响应预测和基于表型的药物重定向等下游任务中展现出有效性。利用TranSiGen筛选胰腺癌候选药物的策略在体外验证中展现出高命中率。相关研究成果已在Nature Communications期刊上发表,题为“Deep representation learning of chemical-induced transcriptional profile for phenotype-based drug discovery”。

背景
基于表型的筛选是药物研发中的重要方法之一,侧重于化合物的更全面的细胞响应,提供对疾病机制更全面的理解,并有可能发现新的药物作用机制和治疗机会。高通量RNA测序技术促进了大规模微扰转录组的产生,例如Integrated Network-based Cell-Signature(LINCS)。虽然化学微扰转录组能够提供对药物作用机制更全面的理解,化合物和细胞系组合的复杂性限制了通过高通量实验进行彻底的探索,促使研究人员发展能够利用大量公共数据构建预测微扰转录组的深度学习模型。然而,转录组数据中固有的高噪声往往掩盖了真正的扰动信号,使得现有机器学习模型难以从中提取有意义的信息。
为解决这一问题,本研究提出基于变分自编码器的TranSiGen模型,利用自监督表征学习进行转录谱的去噪和重建,并且用于推断新的化学微扰转录谱(图1)。TranSiGen同时学习三个分布,包括没有扰动的本底谱、化学微扰转录谱以及它们之间的映射关系。这种自监督方法有效地减少了数据中的噪声,并揭示了潜在的扰动信号。
TranSiGen具有以下关键优势:(1)改进转录谱推断性能:与基准模型相比,TranSiGen在推断本底谱、化学微扰转录谱及对应的差异基因表达(DEGs)方面表现出优越性。(2)可获取细胞和化合物的统一表征:TranSiGen表征有效地捕捉了细胞系和化合物特征,通过可视化分析可证明其区分细胞系和化合物作用机制的能力。(3)TranSiGen表征在各种下游任务中展现有效性,包括:基于配体的虚拟筛选、药物响应预测和基于表型的药物重定向。利用TranSiGen进行抗胰腺癌活性化合物虚拟筛选,体外实验验证结果展现出高命中率,这一结果显示了TranSiGen在药物筛选方面的潜力。
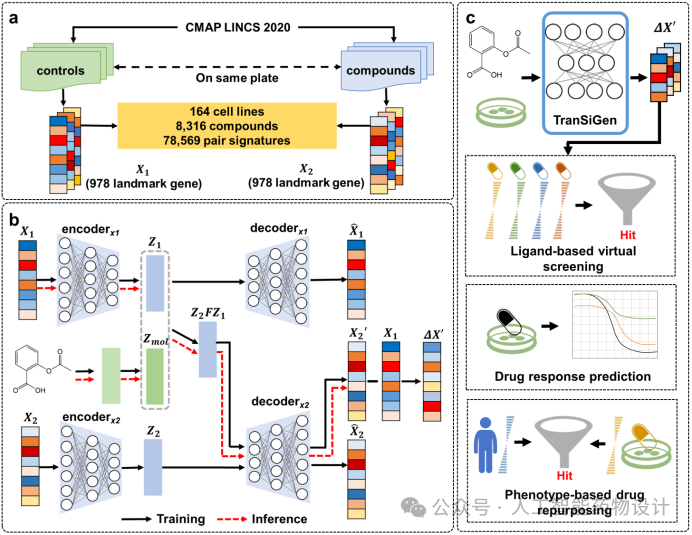
图1 TranSiGen的架构和下游应用。a. TranSiGen的数据处理流程。b. TranSiGen的架构和推理过程。c. TranSiGen表征的下游应用任务。
结果与讨论
1、TranSiGen可以有效表征转录谱
本节评估了TranSiGen在学习本底谱X1,化学微扰转录谱X2以及对应的差异基因表达ΔX方面的表现。结果显示,TranSiGen在重建X1和X2方面表现出色,通过X1和化合物表征预测X2’方面也表现良好(图2a)。同时,分析TranSiGen预测差异基因表达ΔX’的性能与X1~X2相关性的关系,发现随着X1~X2 R2的增加,预测ΔX’的性能也增加。但对于X1~X2 R2>0.8范围内ΔX’的预测性能下降,可能是因为扰动效应过于细微使得模型无法完全捕捉(图2b)。
进一步可视化TranSiGen在学习ΔX’中的细胞和化合物特征方面的有效性来评估其表征能力。相比实验谱ΔX,TranSiGen来源的ΔX’对相同类型的细胞系显示出更清晰的聚集,并且不同细胞类型之间也有更明显的区分(图2c)。这表明TranSiGen表征能够克服高噪声对实验谱的影响,从而提供更有效的细胞类型区分能力。此外,具有相同作用机制的化合物会对转录谱产生类似的影响,TranSiGen来源的ΔX’对相同作用机制的化合物比实验谱ΔX具有更高的相关性(图2d)。
总体而言,TranSiGen的自监督表征学习有助于对转录谱进行去噪和重建,有效地识别和学习数据中有意义的细胞和化合物信息。
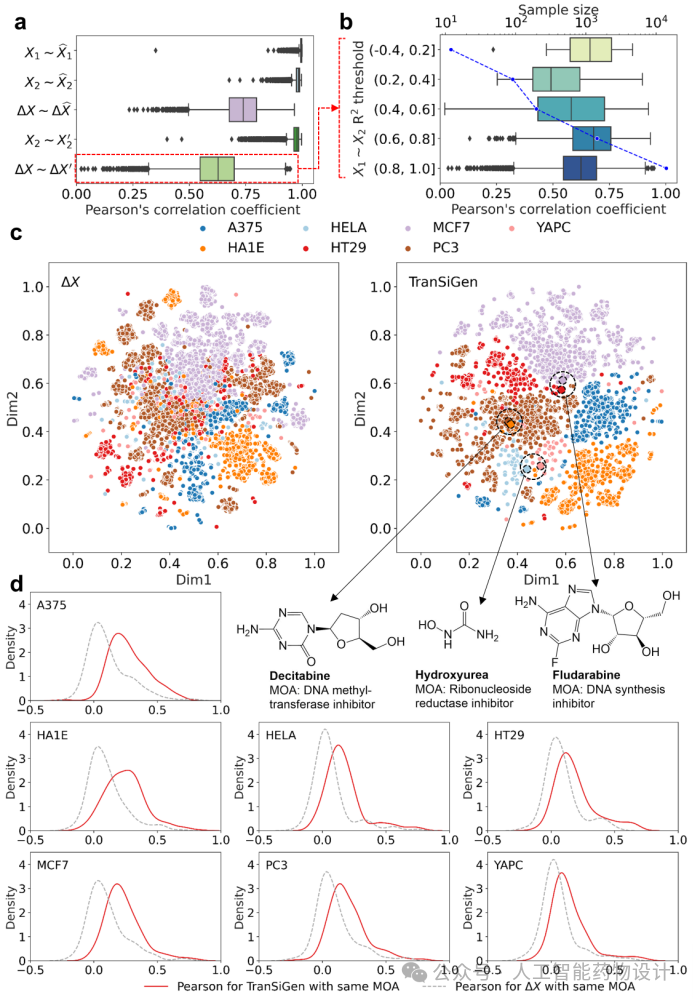
图2 TranSiGen对转录谱的表征学习。a. TranSiGen重建和预测转录谱的性能。b. TranSiGen预测差异基因表达ΔX’的性能与X1~X2相关性的关系。c. 不同细胞系上的实验谱ΔX和TranSiGen来源的差异基因表达ΔX’降维可视化结果。d. 相同作用机制的化合物对应的实验谱ΔX和TranSiGen来源的差异基因表达ΔX’的Pearson相关性分布。
2. TranSiGen与现有模型推断差异表达基因的性能比较
本节评估了TranSiGen与现有基准模型在预测差异基因表达方面的表现,具体包括两种数据划分形式:(1)按照化合物划分,(2)按照细胞系划分(图3a)。在按照化合物划分场景下,TranSiGen在推断未见化合物DEGs方面表现出色,并在完整数据集上获得最优性能(图3b)。在按照细胞系划分场景下,TranSiGen在所有指标上始终优于其他基准模型(图3c)。同时,随着训练细胞数量的增加,TranSiGen在推断未见细胞系差异基因表达方面的性能逐渐提高(图3d)。

图3 数据划分示意图及相应推断差异基因表达性能。a. 分别按照化合物和细胞系划分数据的示意图。b. 按照化合物划分场景下模型性能比较。c. 按照细胞系划分场景下模型性能比较。d. 按照细胞系划分场景下,训练集使用不同数量的细胞系时TranSiGen模型的性能。
3. 下游任务1:基于配体的虚拟筛选
本节研究了TranSiGen表征作为基于配体的虚拟筛选任务中分子表征的潜力。具体来说,使用TranSiGen及其他基准模型在按照化合物和按照细胞系划分场景下预测的差异基因表达作为分子表征,构建随机森林分类器以区分活性和非活性化合物。结果显示,基于TranSiGen表征的模型性能优于基于其他微扰表征的模型(图4a和图4b)。活性/非活性化合物的降维可视化进一步支持了上述结果,TranSiGen表征清楚地区分了两者,而其他微扰表征则表现出两者的重叠(图4c)。
此外,以按照化合物划分场景为例,研究团队进一步探究了融合来自不同细胞系的TranSiGen表征能否提高化合物筛选性能。结果显示,与单独在每个细胞系上的预测性能相比,融合来自不同细胞系的TranSiGen表征可以进一步提高活性化合物筛选性能(图4d)。
作为一种分子表征方式,将TranSiGen表征与其他分子结构表征(分子指纹ECFP4和预训练表征KGPT)进行比较。使用ECFP4计算测试集分子相对于训练集分子的最大谷本相似性,并在不同的最大相似性阈值下评估活性化合物筛选性能。结果显示,对于与训练集不相似的化合物(化学结构相似性∈(0.0, 0.3]),基于TranSiGen表征的模型表现出比基于分子结构表征的模型更好的预测能力(图4e)。这表明使用TranSiGen表征可能在筛选不同于已知化合物结构的新骨架化合物方面具有一定的优势。

图4 基于配体的虚拟筛选性能。a, b. 使用来自按照化合物和细胞系划分场景下模型推断的各种微扰表征进行活性化合物预测性能。c. 基于各种推断微扰表征的活性/非活性化合物降维可视化结果。d. 将不同细胞系的TranSiGen表征进行早期融合和晚期融合后活性化合物预测性能。e. 基于TranSiGen表征和分子结构表征的活性化合物预测性能。
4. 下游任务2:药物响应预测
化学微扰转录谱将分子特征与特定的细胞效应相关联,有利于表征不同细胞的药物响应。本节应用TranSiGen表征来预测化合物在特定细胞系上的剂量反应曲线下面积(AUC)。
为评估是否可以根据TranSiGen表征确定化合物对特定细胞系敏感或耐药,研究团队计算了不同化合物之间的差异基因表达的相似性。同时,使用基于分子指纹ECFP4的谷本相似性比较不同化合物之间的结构相似性。结果表明,TranSiGen表征可以有效区分敏感和耐药化合物(图5a),而分子结构表征无法有效区分两者(图5b)。此外,构建随机森林模型将TranSiGen表征用于药物响应预测,并与结合分子结构和细胞信息的模型(ECFP4+X1和 KPGT+X1)进行性能比较。结果显示,基于TranSiGen表征的模型性能优于其他模型(图5c)。为了评估筛选性能,将化合物根据真实AUC分为敏感或耐药组,并根据预测AUC(AUCspred)进行排序。结果表明,基于TranSiGen表征的模型预测的敏感化合物具有较小的AUCspred和较高的排名(图5d),说明其对敏感化合物具有更好的筛选能力。
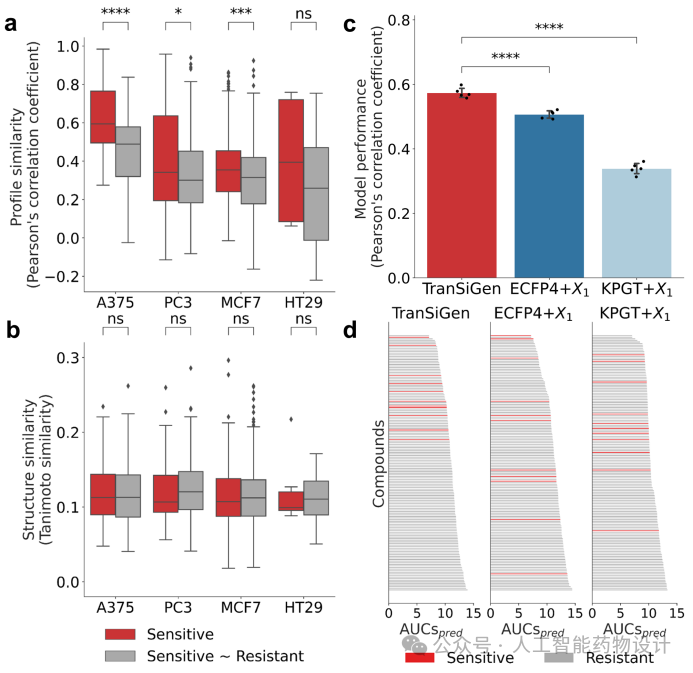
图5 药物响应预测性能。a. 基于TranSiGen表征的敏感化合物组内以及敏感和耐药化合物之间的Pearson相关系数。b. 基于分子指纹ECFP4的敏感化合物组内以及敏感和耐药化合物之间的谷本相似性。c. 使用不同表征的药物响应预测模型性能。d. 使用不同表征的药物响应预测模型根据预测AUCspred对化合物的排序结果。
5. 下游任务3:基于表型的药物重定向
将化学微扰转录谱与疾病联系起来有助于识别治疗特定疾病的潜在化合物。本节中将TranSiGen整合到基于表型的药物重定向流程,用于筛选治疗胰腺癌的候选化合物。首先,评估其从PRISM数据集中优先考虑对YAPC胰腺癌细胞系敏感的化合物的能力。其中,研究团队使用了两种基于表型的筛选策略,并将它们与基于结构相似性的方法进行比较(图6a)。TranSiGen_DRUG使用已批准胰腺癌药物的真实差异基因表达来识别具有类似扰动效应的化合物。相反,TranSiGen_DISEASE寻找可以逆转胰腺癌差异基因表达的化合物。两种策略都使用连通性分数来衡量差异基因表达之间的相关性。为进行比较,ECFP4_DRUG使用基于分子指纹ECFP4的谷本相似性来筛选与已批准胰腺癌药物结构相似的化合物。
三种方法的筛选性能如图6b所示。性能最好的是TranSiGen_DRUG,其次是 TranSiGen_DISEASE,ECFP4_DRUG的预测性能最差。值得注意的是,TranSiGen_DISEASE筛选策略不需要任何化学微扰转录谱,能够模拟疾病缺乏已知治疗药物的情景。这是基于结构相似性的策略无法解决的挑战。同时,基于表型的策略可以识别与已批准药物相似性更低的化合物,例如天然产物thiostrepton和resibufogenin(图6c)。基于表型的策略TranSiGen_DISEASE和TranSiGen_DRUG始终优先考虑它们,而基于结构的策略ECFP4_DRUG将它们分别排在1289和616位(图6d)。
此外,通过TranSiGen_DISEASE和TranSiGen_DRUG对实验室化合物库进行针对YAPC胰腺癌细胞系的表型筛选,分别选择前50个化合物进行体外实验验证,确定这些化合物的细胞增殖抑制活性(图6e和图6f)。结果显示,这两种基于表型的筛选策略展现出了较高的命中率,证实了其识别有效化合物的潜力。
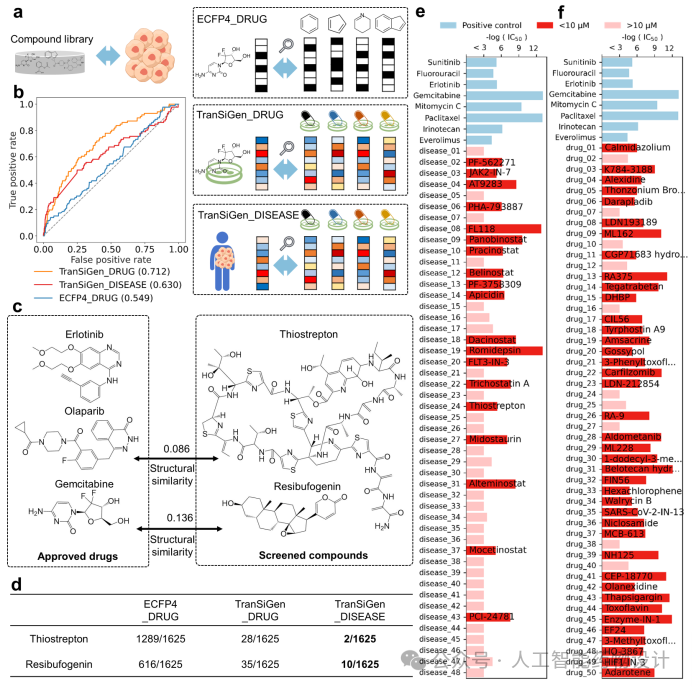
图6 基于表型的药物重定向。a. 化合物筛选策略的流程图。b. 化合物筛选策略的性能。c. 已批准胰腺癌药物与基于表型策略筛选到的化合物的最大结构相似性。d. 不同筛选策略对thiostrepton和resibufogenin的排名。e, f. 分别通过TranSiGen_DISEASE和TranSiGen_DRUG筛选的前50个化合物的细胞增殖抑制活性。
结论
本研究引入深度生成模型TranSiGen,旨在通过自监督表征学习克服转录组中固有噪声和混杂因素的局限,提供了一种标准化方法来表征与细胞环境和化合物效应相关的表型信息。TranSiGen表征在包括虚拟筛选、药物响应预测和基于表型的药物重定向等各种下游任务中展现了有效性,实验验证结果也表明其在药物发现研究具有良好的应用前景。
中国科学院上海药物研究所博士童筱雏为本文的第一作者。中国科学院上海药物研究所郑明月研究员、李叙潼副研究员与张素林副研究员为论文通讯作者。本研究得到了国家自然科学基金、国家重点研发专项、上海药物研究所与上海中医药大学中医药创新团队联合研究项目、中国科学院青年创新促进会会员项目以及上海市科技重大专项资助。感谢国家蛋白质科学研究(上海)设施规模化蛋白质制备系统工作人员提供的技术支持和帮助。
参考文献
Tong et al. Deep representation learning of chemical-induced transcriptional profile for phenotype-based drug discovery, Nat Commun 15, 5378 (2024), https://doi.org/10.1038/s41467-024-49620-3

