如果销售会生信
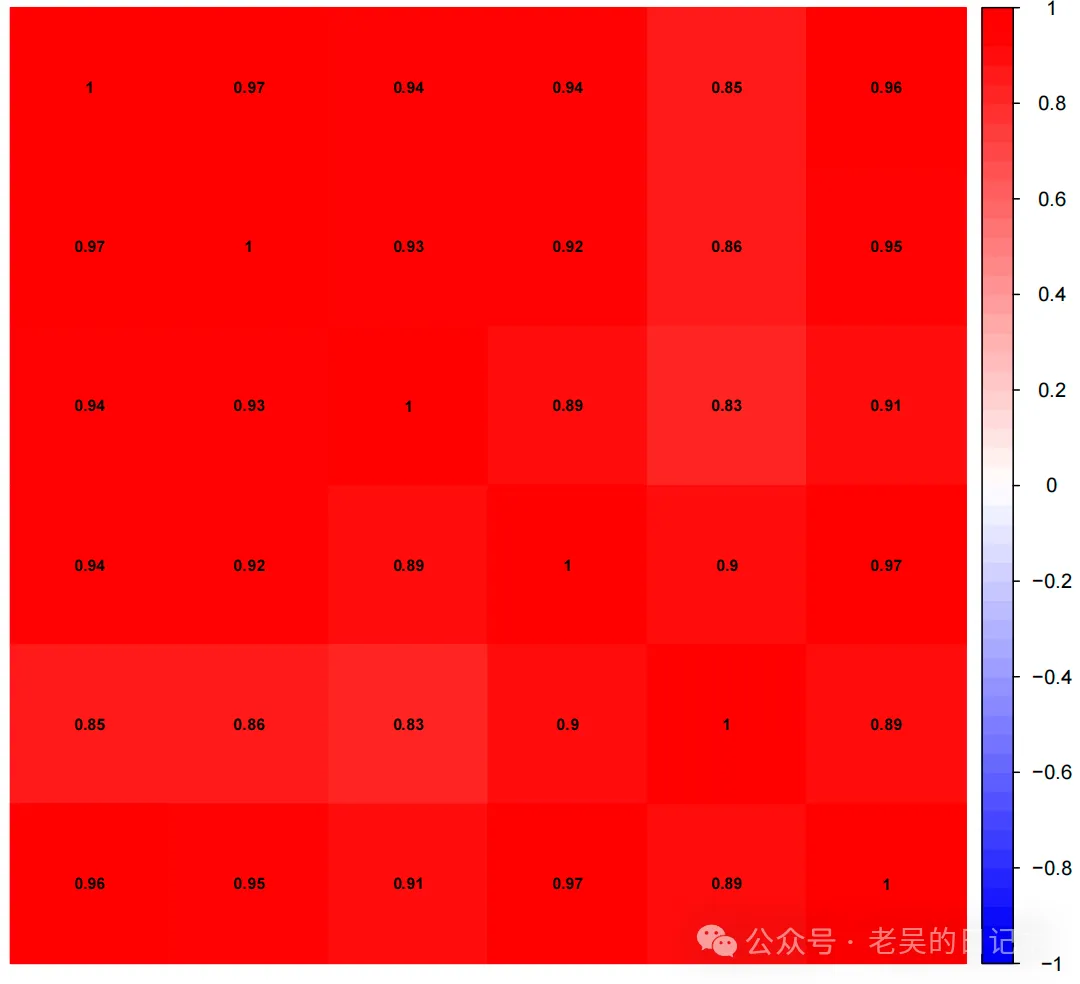
皮尔森相关性系数一直是我对转录组数据评价的重要指标之一,但是最近结题的一个smartseq让我对皮尔森相关性系数有了进一步认识。技术给我发了该项目的结题报告,相关性热图如下:

看到这个热图后,我的第一感觉就是完蛋了,怎么给客户解释…….当天下班后越想越觉得不对,该客户做实验很认真、模式物种的项目、项目启动过程我俩一直保持沟通,他引入这么大差异的可能性很低。虽然是风险样本,但是我感觉是低风险,我们实验员手很稳,也不至于出这么大的纰漏,况且是6个样本都非常低。想到这里,我大概确信这个项目的数据是没有问题的,问题很可能是由于客户的样本细胞偏多导致的部分ncRNA或者低表达基因的随机扩增导致的。于是我回过头查看了一下该项目的基因数目31165个,常规项目大鼠这类模式物种定量的基因很少超过2万,然后我又回过头去查看readscount数目,所有样本居然有3000~6000个为0的。看到这里的时候我基本上觉得找到原因了,于是我尝试读取表达量矩阵然后过滤掉了6个样本中5个为0的数据,使用剩余的数据重新做了相关新热图,果然正常了。
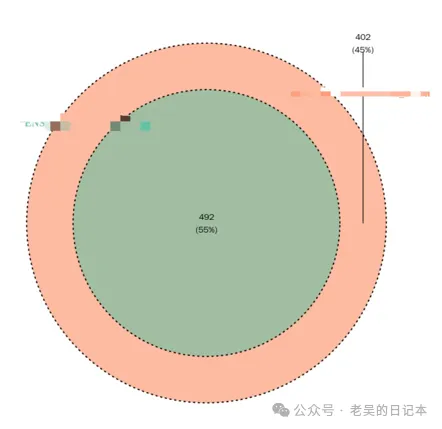
可见样本及数据都是没问题的。那么我进一步的想了想如果数据没有问题,直接采用完整的数据及过滤的数据,两者结果差别大吗?于是我对两者的差异基因做了个韦恩图。我预估两者会有大量重叠,但是我没有想到过滤后的数据差异基因完全是前者的子集。当我把每个样本过滤掉的基因单独求和后发现,过滤掉的基因readscount之和,不到所有基因readscount百分之一。可见这些低表达的基因及ncRNA对转录组定量结果的影响确实很微弱。由于每个基因的P值又是独立的。是子集也就不奇怪了。
这里有很多地方值得思考。一个是对于RNA起始量很低的项目,要留意随机出现的低表达量基因对相关性的影响,很有可能相关性低只是个假象。另外一个就是我想了想之前遇到的项目中会零散的出现个别样本定量的基因很多的现象,当时解释为样本的特殊性,现在看来不是,我估计这个可能是由于oligo(dT)结合的时候发生了非特异性结合,不过由于这部分基因所占的数据非常低,所以对结果的影响很微弱。同时我想到了另外一个问题,由于我们可以认为每个基因的扩增都是独立的,如果遇到相关性低的情况,是不是采用多个内参基因评估,选取一个稳定性最高的内参基因进行标准化结果会更好呢?之前有两个项目给客户这样处理过,结果还可以。那么如果非常明确是测序端导致的相关性低,又不是大量随机出现的低表达基因这种情况,还有什么途径可以把数据给用起来呢?我觉得我们要始终认定,由于每个基因都理想状态下都是独立扩增的,所以如果我们能找到那些接近独立扩增的基因,至少这部分数据可以用起来,有没有可能建立一个随机扩增的核心基因群?或者这种情况直接筛选一个高质量的内参出来,然后用内参进行标准化。
我想如果我们遇到组内相关性差的样本,能否采取其他相关性好的组别位于表达量中间1/2的基因。然后提取相关性差组的这些基因的readscount,剔除异常值,以这些基因的中位数作为内参标准,进行标准化。这里的理论基础我觉得有三点:
- 一、每个基因的扩增理想状态下都是独立的,转录组本质上就是qPCR,找到一个和自身扩增体系相关联的参数进行标准化即可;
- 二、转录组即便受到明显的外源因素影响,大部分基因的独立扩增这个事实依然成立;
- 三、位于readscount中间1/2的基因表达量会更加集中一些,具有一定的缓冲弹性。
分割线
借此机会,给大家一个简单的练习题,看看诸位作为“根正苗红”的生信分析工作者,是否能够从思考深度上面超过一个销售哈:
有一个很简单的表达量芯片数据集(GSE5883):
human lung microvascular endothelial cells in culture were exposed to lipopolysaccharide (LPS), 10 ng, for 4, 8, or 24 hours and changes in mRNA expression were studied using Affymetrix HG U133plus2 gene arrays.
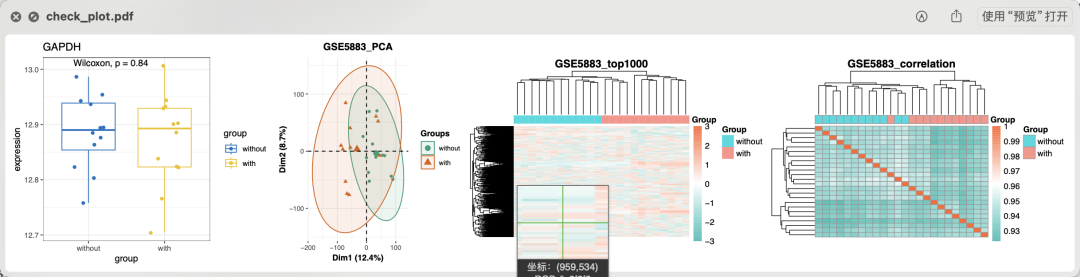
如果是直接读取进去可以看到,处理组和对照组确实是有差异,但是也会被4, 8, or 24 hours 干扰:
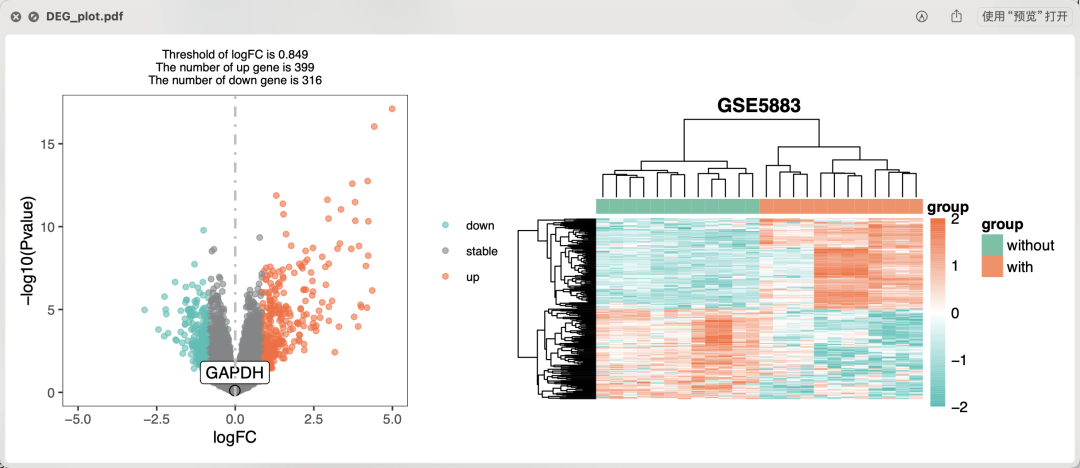
所以总体来说全部的样品的2分组差异分析是可以做的,如下所示
但是也可以在每个时间点(4, 8, or 24 hours )内部进行两分组差异分析,这样的话就会有3次火山图,不同的上下调基因:


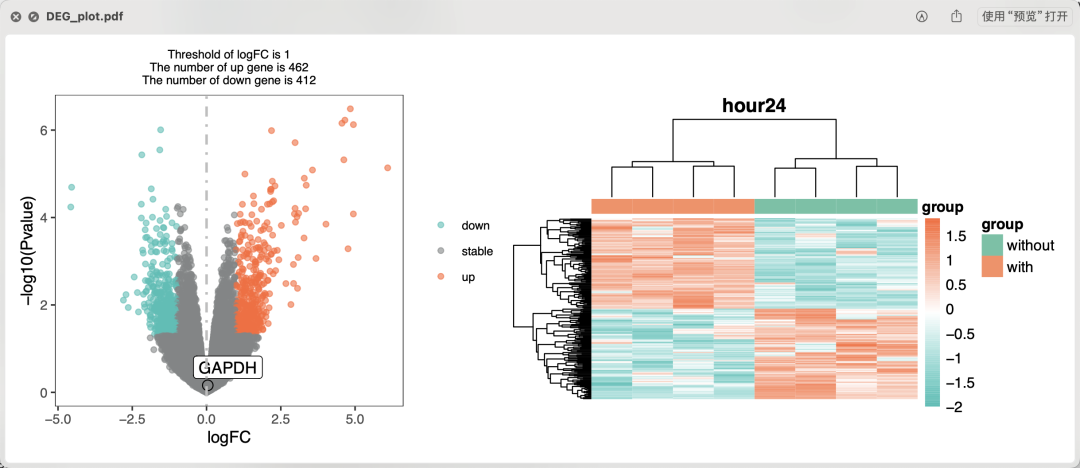
大家需要仔细对比这些统计学显著的上下调基因,说清楚什么样的分析是合理的!
面对这样一个复杂的表达量矩阵,其中涉及人肺微血管内皮细胞在不同时间点暴露于脂多糖(LPS)后的mRNA表达变化,你可以进行多种分析来探究基因表达的变化和生物学意义。以下是一些可能的分析方法:
- 差异表达分析(Differential Expression Analysis):
- 比较LPS处理组和对照组之间的基因表达差异,找出在特定时间点上调或下调的基因。
- 时间序列分析(Time Series Analysis):
- 分析基因表达随时间变化的模式,识别早期响应基因、延迟响应基因或持续响应基因。
- 功能富集分析(Functional Enrichment Analysis):
- 对差异表达基因进行GO(Gene Ontology)和KEGG通路富集分析,了解它们在生物学过程、分子功能和细胞组分中的作用。
- 聚类分析(Clustering Analysis):
- 对基因表达模式进行聚类,识别具有相似表达模式的基因群体,这有助于发现协同调控的基因网络。
