基于单基因SNV对癌症进行分类 | Nature | BRCA2变异的功能评估与临床分类
基于单基因SNV对癌症进行分类 | Nature | BRCA2变异的功能评估与临床分类

Basic Information
- 英文标题:Functional evaluation and clinical classification of BRCA2 variants
- 中文标题:BRCA2变异的功能评估与临床分类
- 发表日期:08 January 2025
- 文章类型:Article
- 所属期刊:Nature
- 文章作者:Huaizhi Huang | Fergus J. Couch
- 文章链接:https://www.nature.com/articles/s41586-024-08388-8
Abstract
Para_01
- 种系 BRCA2 功能缺失变异可以通过临床基因检测识别,这些变异会增加多种癌症的风险。
- 然而,意义不明的变异限制了检测结果的临床应用。
- 因此,需要对所有 BRCA2 变异进行功能表征和临床分类,以帮助管理携带这些变异的个体。
- 我们分析了编码 BRCA2 DNA 结合域热点的所有可能的单核苷酸变异,该区域是致病性错义变异的高发区。
- 为此,我们使用了基于饱和基因组编辑 CRISPR–Cas9 的敲入方法,针对人类单倍体 HAP1 细胞进行了内源靶向研究。
- 该检测通过无义变异和同义变异进行校准,并使用 ClinVar 中的致病性和良性标准以及来自同源定向修复功能检测的结果进行了验证。
- 根据 VarCall 贝叶斯模型,所评估的变异(6,959/6,960)被分为七个致病性类别。
- 编码功能缺失错义变异的单核苷酸变异与乳腺癌和卵巢癌风险增加相关。
- 功能检测结果被整合到 ClinGen、美国医学遗传学与基因组学学院以及分子病理学协会的模型中,用于 BRCA2 变异的临床分类。
- 通过这种方法,91% 的变异被分类为致病性或可能致病性,或者良性或可能良性。
- 这些已分类的变异可用于改善携带 BRCA2 变异个体的临床管理。
Main
Para_01
- BRCA2 是一个已确立的、在临床上具有可操作性的癌症易感基因,并已被广泛用于检测遗传性癌症风险。
- 特别是,BRCA2 的功能丧失型致病变异与69%的终身乳腺癌发病风险以及15%的卵巢癌发病风险相关。
- 胰腺癌或前列腺癌的发病风险也显著增加。
- 目前,致病变异已被用于通过预防、筛查和癌症治疗对携带者进行临床管理。
- 然而,ClinVar 数据库中当前分类为意义不明变异(VUS)的5000多个个体 BRCA2 变异的解读和分类尚未实现。
- 这些主要为错义突变和内含子改变的变异无法有效应用于临床护理。
- 因此,有必要对 BRCA2 变异进行大规模的特征分析和分类。
Para_02
- 最近,美国医学遗传学学院 (ACMG) 和分子病理学协会 (AMP) 发布的指南整合了多种证据来源,包括变异在人群中的频率、基于序列的计算预测和功能数据等,这些指南已被临床检测机构以及 ClinGen BRCA1 和 BRCA2 (BRCA1/2) 变异分类专家小组 (VCEP) 用于变异分类。
- 然而,使用这些模型将变异分类为致病性或可能致病性高度依赖于功能实验的结果。
- 尽管针对 BRCA2 DNA 结合域 (DBD) 错义变异的同源定向修复 (HDR) 功能实验数据已经被整合到 ACMG-AMP 框架中,但这种以及其他低通量功能实验尚未显著解决意义不明变异 (VUS) 的问题。
- 相比之下,多路变异效应分析 (MAVE) 实验能够对大量变异进行功能表征。
- 通过基于细胞的选择和深度测序将基因型与表型联系起来,许多变异可以被功能化,并与已知致病性和良性标准的结果进行比较,正如 BRCA1 和 MSH2 癌症易感基因研究所展示的那样。
- BRCA2 的 MAVE 研究仅限于概念验证工作,这些研究主要集中在 BRCA2 的相对较小区域,并且缺乏验证。
- 在此,我们采用基于 CRISPR-Cas9 敲入的饱和基因组编辑 (SGE) 方法,评估 BRCA2 第 15 至 26 外显子编码 BRCA2 DBD 的所有可能单核苷酸变异 (SNVs) 的功能后果,这是该基因中已知致病性错义变异的唯一位置。
- 结果结合其他遗传和临床证据,纳入 BRCA2 ClinGen-ACMG-AMP 模型,用于将变异分类为致病性或良性,并为携带这些变异个体的临床管理提供全面参考。
Functional analysis of variant effects
Para_01
- 根据变异读取计数与总读取量的比率,计算了每个时间点(D0、D5 和 D14)的复制水平变异频率。
- 使用带有目标区域特定自适应样条的复制水平广义加性模型调整了变异位置依赖效应。
- 计算了 D14 到 D0 比值的对数 2 转换倍数变化(LFC)值,并将其作为 6,959 个(99.9%)单核苷酸变异(SNVs)的原始功能评分(图 2a、扩展数据表 1 和补充表 3)。
- 一种称为 VarCall 的模型,即嵌入高斯双组分混合模型的贝叶斯层次模型,被应用于调整后的位置依赖 D14 和 D0 比值的 LFC 值。
- 每个变异都被分配了一个致病性状态指示器:已知变异为确定性分配,未知变异为概率性分配。
- 具体而言,无义变异被认为具有致病性,而沉默变异(除已知或预测会影响剪接的变异外)则被认为是良性的。
- 我们使用的方法通过包含靶向区域位置和尺度随机效应以及 t 分布误差项来校正批次效应,以允许存在离群值。
- 使用马尔可夫链蒙特卡罗(MCMC)算法获得了 6,959 个 SNVs 的调整后平均功能评分(图 2b 和补充表 3)。
- 基于 AlphaMissense 预测 BRCA2 DBD 中 22.7% 的错义变异可能具有致病性,设定了 0.2 的致病性先验概率,并计算了每个变异的致病性后验概率和贝叶斯因子。
- 根据 ClinGen 规定的 ACMG–AMP 指南的贝叶斯解释,为以下证据强度类别分配了功能性 PS3/BS3 标准的后验概率阈值:强致病性(PStrong)、中等致病性(PModerate)和支持致病性(PSupporting);强良性(BStrong)、中等良性(BModerate)和支持良性(BSupporting);以及意义不明的变异(VUS)(图 2b、扩展数据表 2、补充表 3 和补充图 2)。
- VarCall 模型分析的完整细节可在补充信息中找到。
Fig. 2: Functional annotation of BRCA2 SNVs.
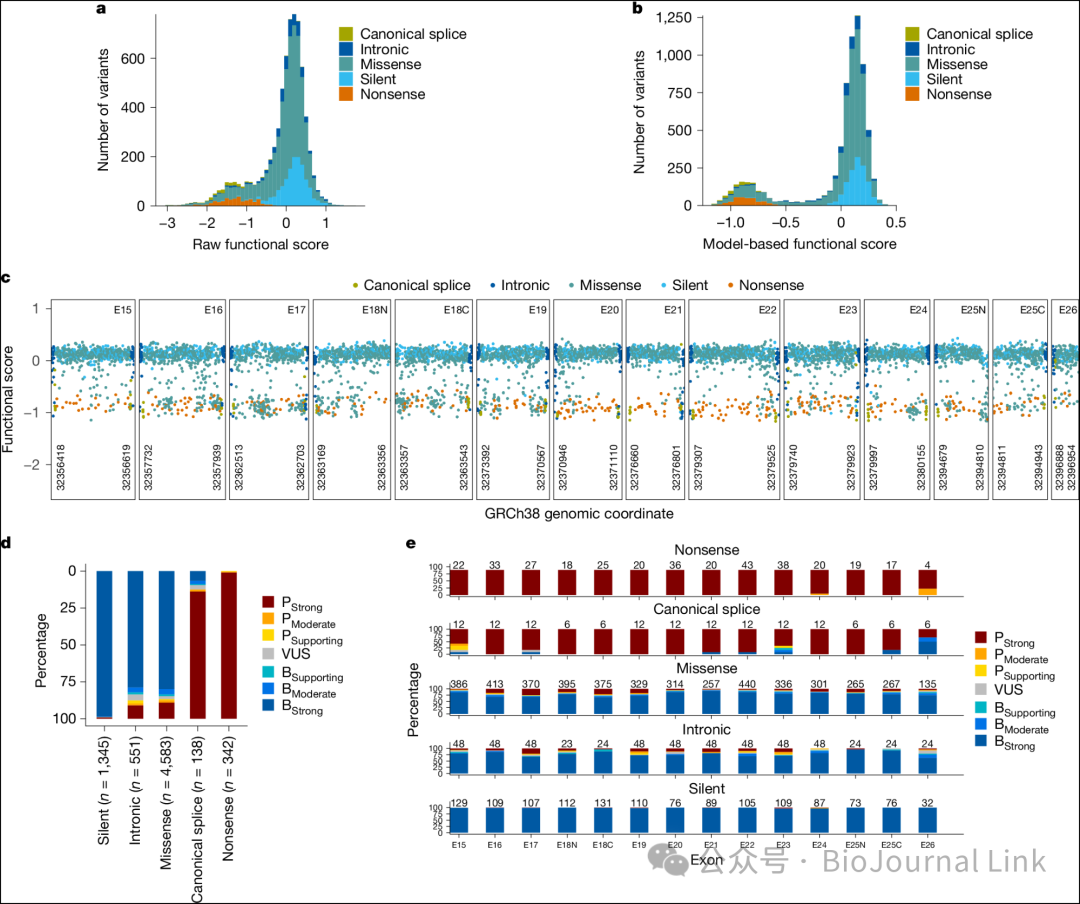
- 图片说明- a,6959个单核苷酸变异(SNVs)的原始功能评分分布,按变异类型着色。- b,VarCall模型中所有变异的调整后功能评分分布。- c,每个外显子中基于模型的功能评分分布,按变异类型分类。颜色表示变异类型。- d,柱状图展示了每个功能类别中每种变异类型的百分比。颜色表示功能类别。- e,柱状图展示了14个目标区域中按功能类别划分的SNVs的百分比。颜色表示功能类别。
Para_02
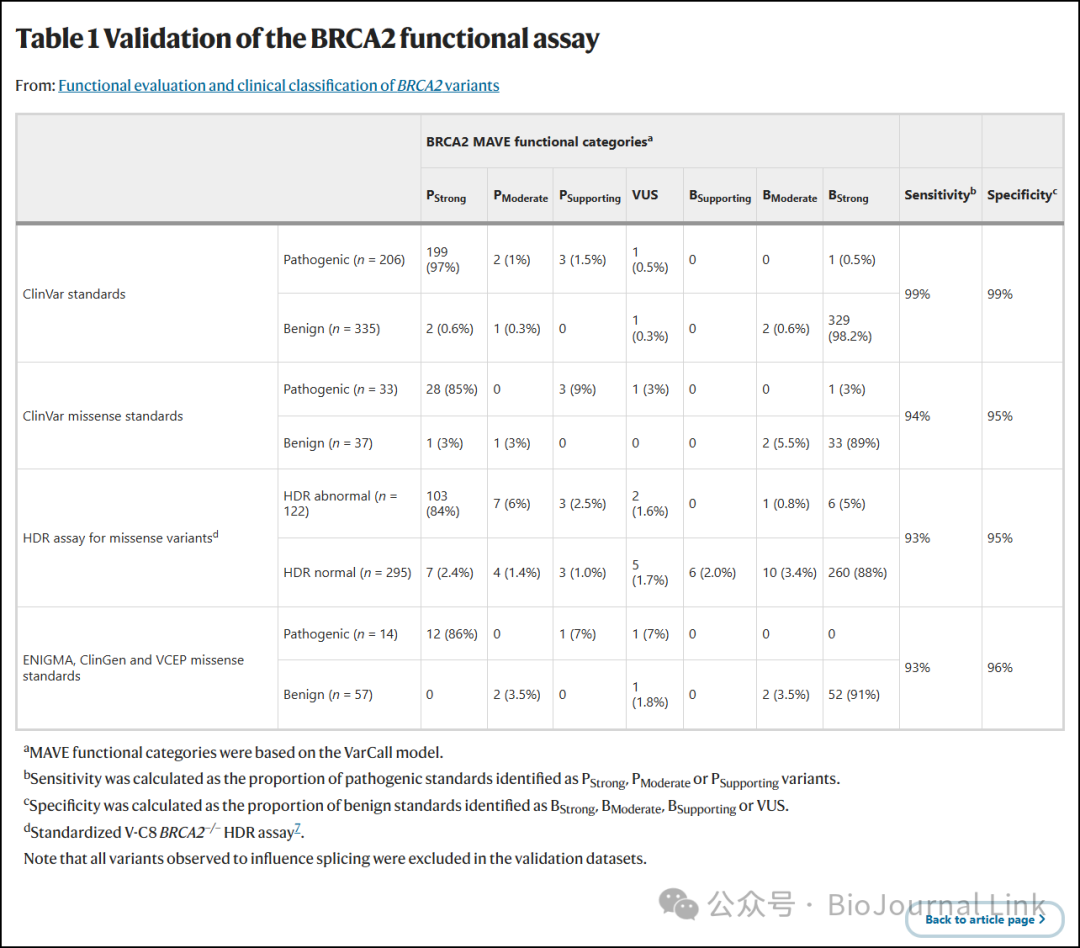
- VarCall模型使用206个已知的致病性变异和335个已知的良性变异进行了验证,其中包括来自ClinVar的70个错义变异,这些变异至少得到了两个ClinGen认证检测实验室的一致结果,或者来自BRCA1/2 VCEP的结果。
- 当包含无义变异和同义变异时,该分析显示对致病性和良性类别的敏感性和特异性均超过99%,而仅与ClinVar错义变异比较时,其敏感性为94%,特异性为95%(表1)。
- 同样地,使用417个通过良好校准的HDR功能测定评估的错义变异进行验证,达到了93%的敏感性和95%的特异性(表1)。
- 在122个HDR功能异常的错义变异中,有7个(5.8%)属于BRCA2 MAVE良性类别;而在295个HDR功能正常的错义变异中,有14个(4.8%)属于MAVE致病性类别(表1)。
- 最后,由Evidence-based Network for the Interpretation of Germline Mutant Alleles (ENIGMA)联盟和ClinGen BRCA1/2 VCEP确定的14个致病性和57个良性错义标准产生了93%的敏感性和96%的特异性。
- 此外,在57个由ENIGMA分类为良性的错义变异中,仅有2个(3.5%)属于MAVE PModerate类别(表1和补充表4a–d)。
Table 1 Validation of the BRCA2 functional assay 表1 BRCA2功能测定的验证
Para_03
- 综合良性(BStrong、BModerate 和 BSupporting)和综合病理性(PStrong、PModerate 和 PSupporting)类别分别占变异的 81.6% 和 16.6%,剩余 1.8% 为意义不明的变异(VUS)。
- 具体而言,5,430 个(78%)变异体被归类为 BStrong,其中包括 3,661 个错义变异、1,326 个同义变异、434 个内含子变异、9 个典型剪接位点单核苷酸变异(SNVs)和 0 个无义变异。
- 相比之下,1,021 个(14.7%)变异体被归类为 PStrong,其中包括 502 个错义变异、339 个无义变异、119 个典型剪接位点变异、50 个内含子 SNVs 和 11 个同义 SNVs(图 2c 和扩展数据表 2)。
- 所有编码无义变异的变异体均属于 PStrong、PModerate 和 PSupporting 类别。
- 在错义变异中,3,879 个(84.6%)属于良性类别,而 611 个(13.3%)属于病理性类别。
- 在 +1/2 和 -1/2 典型剪接位点位置的 138 个变异中,121 个(87.7%)属于病理性类别,这表明存在异常剪接效应。
- 此外,69 个(12.5%)内含子 SNVs 和 13 个(1%)同义变异属于病理性类别(图 2c-e、扩展数据表 2 和补充表 3)。
- 因此,MAVE 研究揭示了大量可能影响 RNA 剪接的变异。
- 另外,1,329 个(98.8%)同义变异属于良性类别。
Correlation with DBD architecture
Para_01
- 为了深入了解单核苷酸变异(SNVs)破坏BRCA2活性的机制,评估了由强效应错义突变引起的变化在蛋白质结构中的位置及其影响。
- 强效应错义变异在螺旋结构域和OB1结构域中富集(分别为15.3%和16.4%)。
- 这些变异在OB2和OB3结构域中较少见(分别为8.7%和10.9%,P = 5.9 × 10–6),并且在塔结构域中非常罕见(1.8%)。
- 此外,在423个强效应错义变异中,有154个(36.4%)位于螺旋结构域,125个(29.6%)位于OB1结构域,83个(19.6%)位于OB3结构域。
- 相比之下,只有45个(10.6%)位于OB2结构域,13个(3.1%)位于塔结构域。
- 这些结果与针对462个DBD错义变异的HDR检测结果一致,显示功能性异常的错义改变在螺旋、OB1和OB3结构域中富集。
- 在塔结构域中发现了13个强效应错义变异,此前该区域未发现任何致病性或非功能性错义变异,这证实了该结构域对于正常的BRCA2功能是必需的,并表明它并非是失活或潜在致癌变异的冷点。
- 在50个与DSS1相互作用的残基中,观察到26个(52%)存在强效应错义改变;而在17个与单链DNA相互作用的残基中,仅观察到2个(12%)存在此类改变。
- 这一结果表明,DSS1介导的稳定性对BRCA2同源重组修复活性至关重要。
- 在423个强效应错义改变中,有261个(62%)导致氨基酸电荷发生变化或脯氨酸残基的丢失或获得。
- BRCA2 DBD中的许多残基从河豚鱼到智人高度保守。
- 在103个(48.6%)完全保守的残基中至少观察到一种强效应错义变异,其中45个(44%)位于螺旋结构域,30个(32%)位于OB1结构域。
- 强效应变异还出现在71个(31.6%)高度保守的残基和39个(15.5%)低度保守的残基中。
- 约75%携带强效应变异的残基位于维持BRCA2必要三维折叠的α-螺旋和β-片层结构中。
Comparisons with functional predictors
Para_01
- ClinGen BRCA1/2 变异临床分类专家组使用了多种功能检测方法,以评估 BRCA2 错义变异对蛋白质功能的影响。
- BRCA2 的 MAVE 数据与基于细胞的同源定向修复 (HDR) 检测结果高度相关(P = 1.6 × 10^-52;表 1 和图 3a),并且能够有效区分 4-5 类(功能性异常)和 3 类(不确定)(P = 4.8 × 10^-7)或 1-2 类(功能性正常)(P = 2.0 × 10^-11)变异,这些数据来源于奥拉帕利 PARP 抑制剂反应检测(图 3b)。
- MAVE 数据还在针对第 15 和 17 外显子的内源性靶向精编研究中,有效区分了非功能性与功能性(P = 3.4 × 10^-8)或不确定(P = 1.3 × 10^-6)变异(图 3c),并在一个小型胚胎干细胞互补性检测中区分了非功能性与功能性错义变异(P = 1.1 × 10^-4)(图 3d)。
- 因此,MAVE 分析的数据与多种其他小规模功能检测的结果高度一致(图 3a-d 和补充表 5a-d)。
- 值得注意的是,MAVE 数据显示,这些检测中的 3 类、不确定或中间型变异主要表现为强支持、中等支持或有限支持。
Fig. 3: Comparison of BRCA2 MAVE data with data from functional assays and in silico predictors.
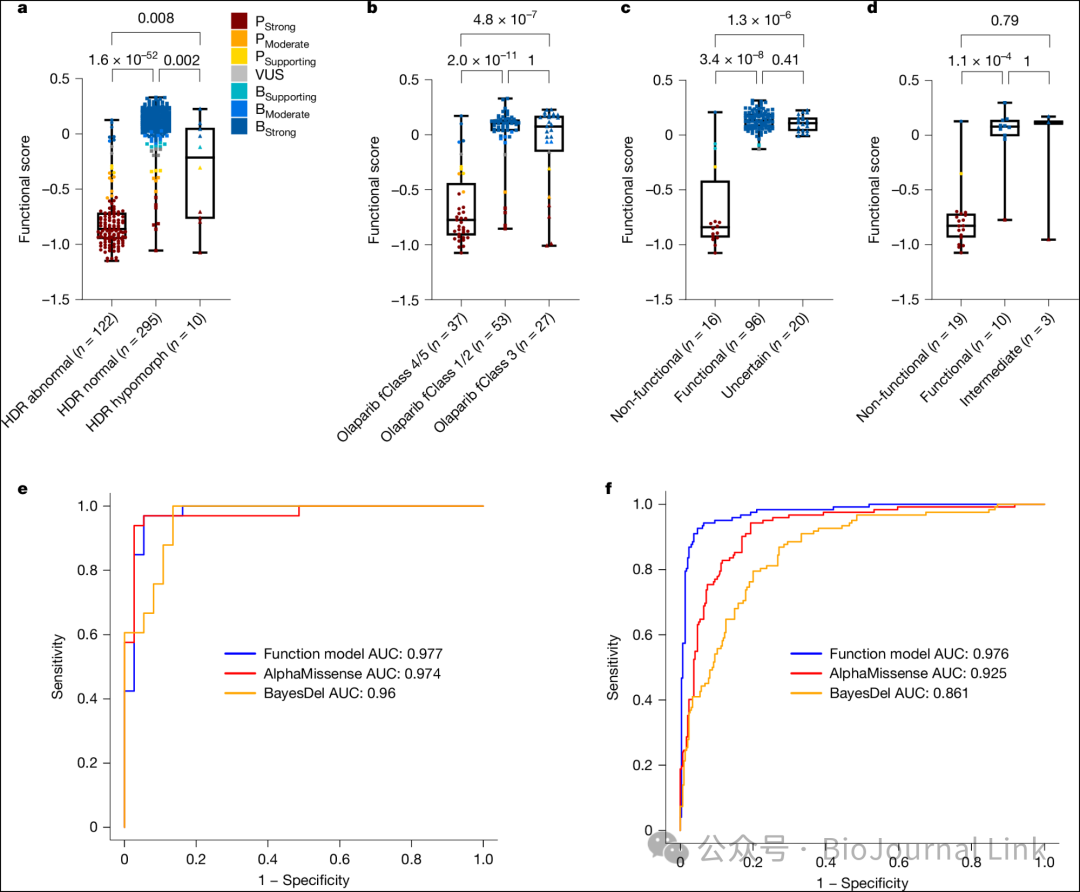
- 图片说明- 盒须图显示了与 BRCA2 缺失的 V-C8 HDR 测定(a)、DLD1 BRCA2 缺失的奥拉帕尼敏感性测定(b)、基于主编辑的单倍体细胞存活测定(c)以及小鼠 Brca2 缺失胚胎干细胞互补测定(d)相比,编码错义变异的 SNVs 的功能评分。- 各单独测定中每种类型的变异数量已显示。- 在功能上异常的变异体相较于功能正常的变异体,其功能评分显著较低,分别见于 a(P = 1.6 × 10⁻⁵²)、b(P = 2.0 × 10⁻¹¹)、c(P = 3.4 × 10⁻⁸)和 d(P = 1.1 × 10⁻⁴),使用双侧 Mann–Whitney–Wilcoxon 检验得出。- 所有比较的 P 值均已显示。- 箱线图表示四分位距,水平线为中位功能评分,须线表示最大值和最小值。- 变异体以点表示,并根据证据类别的功能性强度进行着色。- 比较了 MAVE 和两种计算预测器(AlphaMissense 和 BayesDel)在使用 ClinVar 分类的错义标准(n = 70)时的 AUC 值。- 比较了 MAVE 和两种计算预测器(AlphaMissense 和 BayesDel)在使用经过良好校准的 HDR 测定表征的错义变异(n = 417)时的 AUC 值。
Para_02
- 接下来,使用 MAVE 的 PStrong、PModerate 和 PSupporting 类别以及 BStrong、BModerate 和 BSupporting 类别,对 MAVE 结果与计算机预测方法进行了比较。
- Align-GVGD 模型的 C65 类(可能非功能性)类别表现出中等敏感性(41%)和高特异性(91%),与 MAVE 结果相比。
- AlphaMissense 深度学习模型在可能致病性评分阈值(>0.564)的情况下,也产生了中等敏感性(74%)和特异性(84%)。
- BayesDel 预测器目前被 ClinGen BRCA1/2 VCEP 用于 BRCA1/2 变异的整理,使用 ClinGen 规定的 PStrong BayesDel 预测器时,其敏感性为中等(73%),特异性为中等(83%);但使用 BRCA1/2 VCEP 致病性阈值时,其敏感性为中等(43%),特异性较高(95%)。
- 基于 70 个 ClinVar 分类的错义变异(n = 70),BRCA2 MAVE、AlphaMissense 和 BayesDel 数据的受试者工作特征曲线(AUC)下的面积值均超过 0.96。
- 然而,在与 HDR 特征变异进行比较时,BRCA2 MAVE 数据的 AUC(0.98)优于 AlphaMissense(0.93)或 BayesDel 数据(0.86)(图 3e,f)。
Cancer risks for variant categories
Para_01
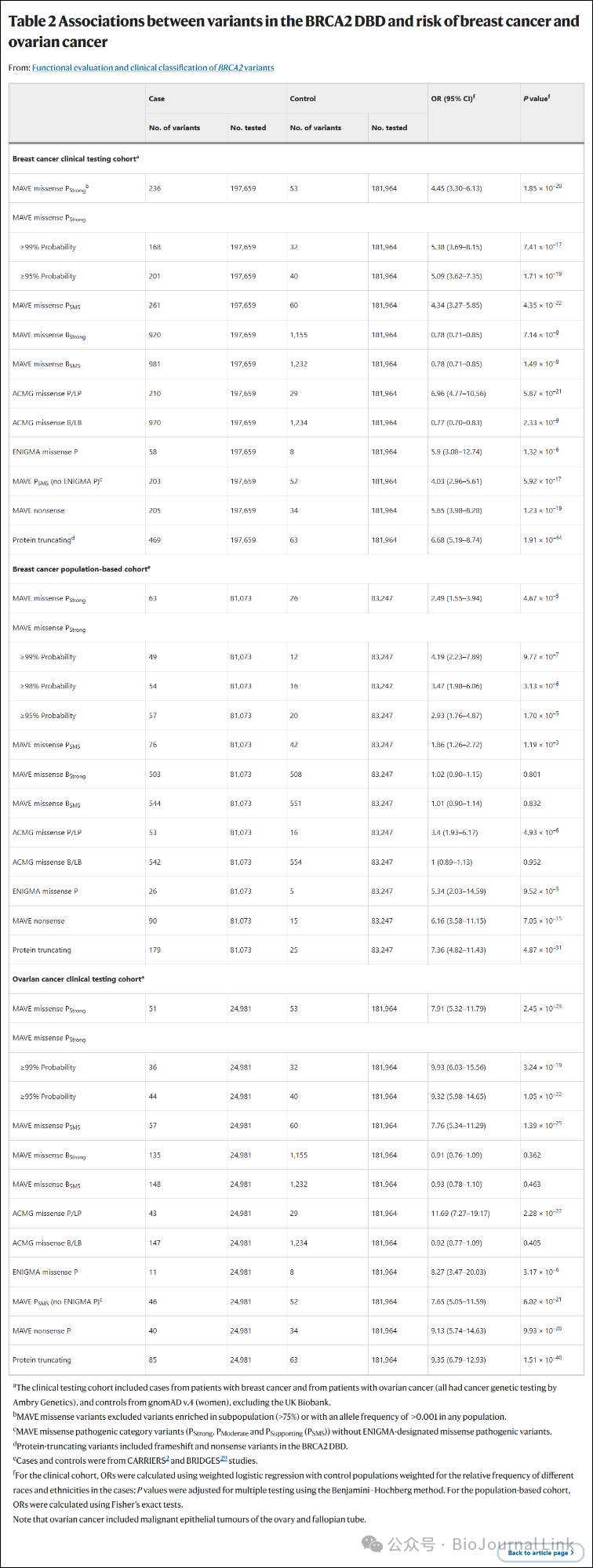
- 为了了解已确定的变异对癌症风险的贡献,评估了功能类别 PStrong、PModerate 和 PSupporting 以及 BStrong、BModerate 和 BSupporting 中组合变异与乳腺癌和卵巢癌之间的关联,这些评估基于病例对照研究。
- 具体来说,比较了2012年至2021年间接受 Ambry Genetics 遗传检测的女性乳腺癌患者中错义变异的频率,以及 gnomAD v.4 参考对照组(女性)中的频率(排除 UK Biobank 数据)。
- 仅 PStrong 类别的错义变异(比值比 (OR) = 4.45,95% 置信区间 (CI) = 3.30–6.13)以及 PStrong、PModerate 和 PSupporting 组合的错义变异(OR = 4.34,95% CI = 3.27–5.85)显示出高风险(OR > 4.0)的乳腺癌风险。
- 相比之下,BStrong、BModerate 和 BSupporting 错义变异并未表现出临床相关(OR > 2)的显著乳腺癌风险增加(OR = 0.78,95% CI = 0.71–0.85)。
- PStrong、PModerate 和 PSupporting 错义变异的 OR 相较于 PStrong、PModerate 和 PSupporting 无义变异(OR = 5.65,95% CI = 3.98–8.28)有所减弱。
- 由 ENIGMA 专家组指定的致病性错义变异(OR = 5.9,95% CI = 3.08–12.74)以及 DBD 蛋白截断变异(OR = 6.68,95% CI = 5.19–8.74)的 OR 值也高于 PStrong、PModerate 和 PSupporting 错义变异。
- 然而,在限制为 PStrong 类别中致病性后验概率 ≥95% 的变异时,76%(502 个中有 380 个)的错义变异显示出与无义变异相似的风险(OR = 5.09,95% CI = 3.62–7.35)。
- 对于致病性后验概率 ≥99% 的变异(502 个中有 60%,即 299 个),风险进一步增加(OR = 5.38,95% CI = 3.69–8.15)。
- 使用非癌症 gnomAD v.2.1 和 v.3.1 对照参考数据集代替 gnomAD v.4 数据集时,也观察到中等(OR = 2–4)至高风险的乳腺癌风险(补充表 7)。
- 在自认为是非裔美国人的女性中,PStrong、PModerate 和 PSupporting 错义变异(OR = 3.34,95% CI = 1.59–7.13)也显示出中等至高风险的乳腺癌风险(补充表 7)。
- 利用 CARRIERS 和 BRIDGES 基于人群的乳腺癌研究以及 UK Biobank(www.ukbiobank.ac.uk)的病例对照数据进行的额外分析得出了类似的结果。
- 然而,由于病例和对照组的基于人群性质,OR 值有所减弱(表 2 和补充表 7)。
- 在基于人群的研究中,值得注意的是,PStrong 类别中致病性后验概率 ≥99% 的变异(299/502;60%)与高风险的乳腺癌相关(OR = 4.19,95% CI = 2.23–7.89)。
- PStrong、PModerate 和 PSupporting 错义变异还与显著增加的卵巢癌风险相关(OR = 7.76,95% CI = 5.34–11.29),但相较于无义变异有所减弱。
- 然而,PStrong 类别中致病性后验概率 ≥95% 的变异(OR = 9.32,95% CI = 5.98-14.65)与无义变异(OR = 9.13,95% CI = 5.74–14.63)在卵巢癌风险方面表现出相似性。
- 通过当前研究中的 OR 值以及 Surveillance, Epidemiology, and End Results (SEER) 注册报告的疾病发生率,估计了乳腺癌和卵巢癌的终身风险。
- PStrong 错义变异导致的乳腺癌和卵巢癌的终身风险分别为 41% 和 11%(截至 80 岁),这与 DBD 蛋白截断变异导致的 52% 和 12% 的风险相似(扩展数据图 2)。
- 所有展示的数据均在机构审查委员会批准后,获得研究参与者的明确书面同意。
Table 2 Associations between variants in the BRCA2 DBD and risk of breast cancer and ovarian cancer 表2 BRCA2 DBD中的变异与乳腺癌和卵巢癌风险之间的关联
Clinical classification of SNVs
Para_01
- 功能性数据必须整合到分类模型中,以确定每个变异的临床相关性。
- 在这里,适应于评分系统的 ClinGen BRCA1/2 VCEP 分类框架被应用于 MAVE 的单核苷酸变异(SNVs)。
- 如上所述,基于贝叶斯解释的 ACMG–AMP 指南,确定了 PS3/BS3 代码下 PStrong、PModerate、PSupporting、BStrong、BModerate 和 BSupporting 功能类别的阈值。
- PStrong 和 BStrong 类别限制在 +4 或 -4 分以内,以避免仅凭功能证据进行分类。
- PModerate 和 BModerate 被分配 +2 或 -2 分,而 PSupporting 和 BSupporting 类别被分配 +1 或 -1 分(图 4a 和补充表 8)。
- 来自每个 VCEP 代码(包括基于功能的 PS3/BS3 代码)的每个变异得分相加后,将变异分类为致病性(P)(≥10 分)、可能致病性(LP)(6 至 9 分)、不确定/VUS(-1 至 5 分)、可能良性(LB)(-6 至 -2 分)或良性(B)(≤ -7 分)(补充表 8)。
- 总体而言,在所有 SNVs 中,5,566 个被分类为 B/LB,785 个为 P/LP,608 个为 VUS。
- 在无义 SNVs 中,3 个被分类为 LP,339 个被分类为致病性。
- 在 4,583 个错义 SNVs 中,使用 BRCA1/2 VCEP 规则时,261 个被分类为 LP/P,3,786 个为 LB/B,536 个仍为 VUS(图 4a 和扩展数据表 5)。
- 值得注意的是,分类为 LP/P 的错义变异与乳腺癌高风险相关(OR = 6.96,95% CI = 4.77–10.56),而分类为 LB/B 的错义变异未显示出增加乳腺癌风险(OR = 0.77,95% CI = 0.70–0.83)(表 2)。
- 在 138 个经典剪接位点中,23 个被分类为 LP,105 个被分类为致病性。
- 总体而言,在具有 mRNA 测序数据和 PVS1(RNA)加权分数的 48 个经典剪接位点变异中,有 43 个在 MAVE 测验中被分类为 PStrong 和 PModerate(补充表 8)。
- 在 BRCA2 功能分析中被指定为 BStrong 和 BModerate 的四个 +2 位置的经典剪接位点变异,在 BRCA1/2 VCEP 中被归类为 PVS1NA,并获得 0 分,最终被分类为 LB(补充表 8)。
Fig. 4: Clinical classification of BRCA2 SNVs.
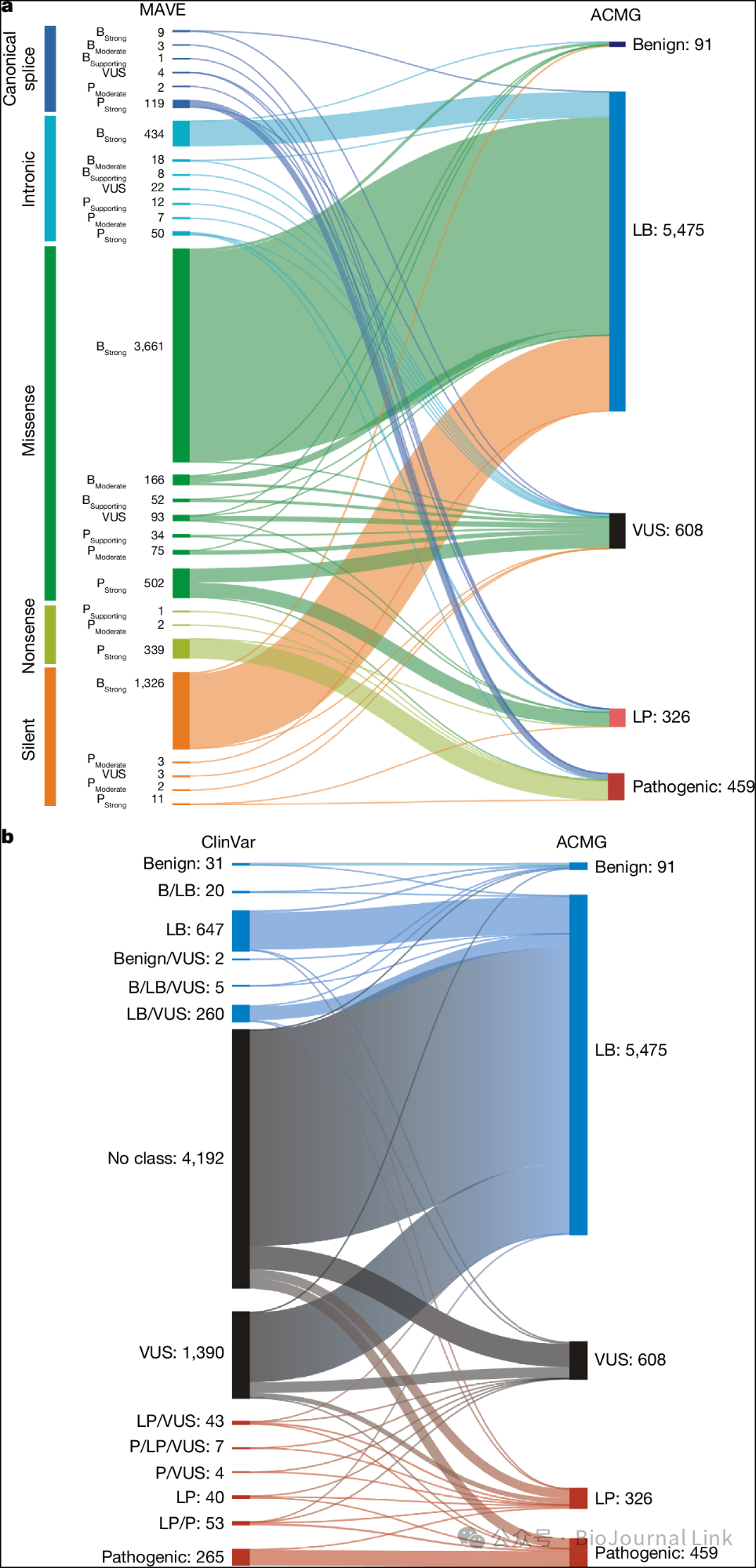
- 图片说明- 桑基图展示了在ClinGen BRCA1/2 VCEP ACMG–AMP分类框架中整合BRCA2 MAVE功能数据后,SNVs的临床分类结果。- 每种变异类型的功能类别中SNVs的数量显示在左侧的MAVE列中。- 分类类别的变异数量显示在右侧的ACMG列中。- 桑基图展示了在将BRCA2 MAVE功能结果整合到BRCA1/2 VCEP ACMG–AMP分类框架前后,ClinVar中变异分类状态的变化情况。
Para_02
- 为了评估 BRCA2 功能研究结果对变异分类的影响,将其与 ClinVar 和 ENIGMA 的分类结果进行了比较。
- 在 5,589 个被分类为 B/LB 的单核苷酸变异中,ClinVar 和 ENIGMA 占了 724 个(13.0%),而 BRCA2 功能研究单独占了 4,865 个(87.0%)。
- 在 793 个被分类为 P/LP 的变异中,ClinVar 和 ENIGMA 占了 396 个(49.9%),功能研究单独占了 397 个(50.1%)(图 4b、扩展数据表 5 和补充表 8)。
- 此外,在 ClinVar 中分类不一致的 322 个 SNVs(P/LP 对比 VUS 或 B/LB 对比 VUS)中,当将 BRCA2 功能数据纳入 BRCA1/2 VCEP 模型时,其中 290 个(90.0%)被分类为 B/LB 或 P/LP,而 32 个仍保持为 VUS。
- 为了比较当前功能研究和平行进行的小鼠胚胎干细胞存活分析的结果,将两项研究的功能数据整合到了 BRCA1/2 VCEP 分类模型中。
- 一致性达到 87%,仅有 1%(n = 60)的变异被分配到相互冲突的分类类别(扩展数据图 3)。
- 值得注意的是,ClinVar 中这 60 个冲突 SNVs 中的 5 个(c.8168A>C、c.8976A>C、c.8982A>T、c.8995C>G 和 c.9005A>G)的分类以及当前 MAVE 研究中评估的 12 个错义 SNVs 中的 10 个(c.7634T>G、c.7679T>C、c.7796A>C、c.7823C>T、c.7904A>G、c.8060T>G、c.8168A>C、c.8588A>G、c.8594T>C、c.9272T>G,但不包括 c.7967T>G 或 c.8300C>T)的 HDR 结果与当前 MAVE 研究的结果一致(补充表 3)。
Phenotypic characteristics for SNVs
Para_01
- 在基于人群的 CARRIERS 研究中,携带 PStrong 或 PModerate SNVs 的女性乳腺癌平均诊断年龄为 56 岁,明显低于携带 BStrong 或 BModerate SNVs 的女性 61 岁的平均诊断年龄(P < 0.001)。
- 在临床检测队列中也观察到类似的显著差异(P < 0.001),尽管临床队列中年轻发病的情况更为集中。
- 同样,在临床队列中,错义 SNVs(P = 0.039)以及根据 BRCA1/2 VCEP 分类模型被分类为 LP/P 和 LB/B 的 SNVs 之间也观察到了显著差异。
- 对于具有 PStrong 或 PModerate SNVs 的个体,与具有 BStrong 或 BModerate SNVs 的个体相比,或在临床检测队列中被分类为 LP/P 和 LB/B 的 SNVs,其乳腺癌家族史(定义为任何一级或二级亲属患病)存在显著差异(P < 0.001)。
- 在基于人群的研究中也观察到了类似的趋势。
Para_02
- 通过评估肿瘤中BRCA2的杂合性丢失(LOH),判断BRCA2中的PStrong和PModerate变异是否可能是肿瘤发展的驱动因素。
- 在包含超过40%肿瘤成分的50,000例乳腺肿瘤、卵巢肿瘤、前列腺肿瘤和胰腺肿瘤样本中评估了BRCA2位点的LOH,这些样本使用癌症基因面板测序技术在整合可操作癌症靶点的突变分析(IMPACT)研究中进行了测序。
- 在携带BRCA2 PStrong单核苷酸变异(SNVs)的26个肿瘤中有22个(85%)检测到LOH,而在与PStrong和PModerate SNVs相关的29个肿瘤中有23个(79%)检测到LOH(扩展数据表7和补充表10)。
- 相比之下,在携带BStrong变异的233个肿瘤中有58个(25%)观察到LOH,这与失活变异的情况显著不同(P = 3.1 × 10–9)(扩展数据表7和补充表10)。
- 因此,PStrong和PModerate变异似乎富集了野生型BRCA2等位基因的丢失以及BRCA2的失活,这一结果与这些SNVs作为肿瘤形成驱动因素的作用一致。
Discussion
Para_01
- BRCA2基因变异的功能评估一直是研究的热点领域。
- 这是因为与BRCA2失活变异相关的多种癌症(乳腺癌、卵巢癌、前列腺癌、胰腺癌和胆管癌)具有高风险,同时BRCA2中存在大量意义未明的变异(VUS),这些变异通常需要结合功能证据才能进行临床分类,并且通过这些研究可以深入了解BRCA2的功能和生物学特性。
- 然而,迄今为止,只有557个位于BRCA2 DNA结合域(DBD)的错义变异通过已建立的功能检测进行了评估。
- 由于已鉴定出的具有临床不确定性的变异数量巨大,因此需要更快速的功能表征方法。
- 在本研究中,使用单倍体细胞的饱和基因组编辑(SGE)技术对编码BRCA2 DNA结合域致病变异热点的所有BRCA2单核苷酸变异(SNVs)进行了功能评估,评估指标为细胞活力。
- 从12个编码外显子和23个相邻内含子序列中获得了6,959个SNVs的功能评分(占所有可能SNVs的99%)。
- 尽管之前已有超过600个DBD SNVs通过其他功能检测进行了评估,但本研究建立了几乎涵盖BRCA2 DNA结合域所有可能SNVs的序列-功能图谱。
- 每个变异在贝叶斯VarCall模型中被赋予了一个致病概率。
- 根据ClinGen–ACMG–AMP变异分类指南中的PS3/BS3规则(变异对蛋白质功能的影响),基于贝叶斯解释,将变异分为七个与致病性证据强度相关的类别。
- 在功能研究中直接为每个变异分配后验概率和致病性证据强度是BRCA2变异特征描述的重大进展(类似于先前对BRCA1 RING结构域错义变异的研究)。
- 以往的方法主要关注功能检测的敏感性和特异性,并将变异分为非功能性、不确定性和功能性三类,而在VarCall方法中,每个变异均独立评估。
Para_02
- 值得注意的是,功能数据并不能直接确定任何变异的临床相关性。
- 目前,只有通过将功能数据整合到 ClinGen–ACMG–AMP 分类模型中才能实现这一点。
- 为此,根据 PStrong 和 BStrong 类别,ClinGen–ACMG–AMP PS3/BS3 规则下的功能数据分别被限制在致病性得分为 +4 和良性水平得分为 -4,以避免仅凭功能证据对变异进行分类(+6 分足以达到 LP 分类)。
- 这些 PS3/BS3 分数随后与来自其他遗传和临床数据的评分相结合,按照 ClinGen–ACMG–AMP BRCA1/2 VCEP 规则对变异进行分类。
- 结果是,261 个错义单核苷酸变异(SNVs)和所有 SNVs 中的 785 个被分类为 P/LP,而 3,786 个错义 SNVs 和所有 SNVs 中的 5,566 个被分类为 B/LB。
- 尽管仍有 536 个错义变异和 608 个 SNVs 被归类为 VUS,但随着来自其他来源的数据补充到现有的功能数据中,未来许多这些变异很可能被分类为 P/LP 或 B/LB。
Para_03
- 尽管之前 ClinVar 已将 1,120 个 BRCA2 DBD 单核苷酸变异 (SNVs) 分类为致病性/可能致病性 (P/LP,n = 396) 或良性/可能良性 (B/LB,n = 724),但功能数据使分类的 SNVs 数量增加到 6,382 个。
- 因此,该功能研究涵盖了所有分类的 82%,这对比以往对意义不明变异 (VUS) 的分类有显著改进,并预计对这些种系变异的众多携带者具有重要意义。
- 携带 P/LP 变异的个体现在可能符合强化乳腺 X 光摄影和磁共振成像 (MRI) 筛查的条件,并可通过预防性乳房切除术或卵巢切除术来降低癌症发展的可能性。
- 此外,携带者可能有资格接受乳腺癌、卵巢癌以及潜在的其他癌症(如前列腺癌和胰腺癌)的 PARP 抑制剂治疗,无论是辅助治疗还是转移性治疗阶段。
- 此外,P/LP 变异携带者的家庭成员可能通过检测、预防措施和早期筛查受益,从而在癌症发生前采取行动。
- 此外,携带 B/LB 变异的人可以从这一信息中受益,因为他们所携带的变异很可能不是导致癌症易感的等位基因。
Para_04
- 通过三个独立的数据集验证了功能研究:ClinVar的致病性和良性变异;正交HDR测定的功能异常和正常变异,以及无义和同义变异。
- 总体而言,在每个验证集中,VarCall模型对标准的误分类率仅为约5%。
- 尽管这一结果可能暗示ACMG–AMP–ClinGen对BRCA2单核苷酸变异(SNVs)临床分类中存在错误,但正式分类需要多种证据来源,从而最大限度地减少了错分的可能性。
- 然而,随着其他功能研究的完成,每种变异在不同研究之间的一致性将有助于克服任何特定研究中的误差。
- 事实上,使用ClinGen–ACMG BRCA1/2 VCEP模型进行的变异分类显示,当前的BRCA2功能研究与平行进行的胚胎干细胞中细胞存活的BRCA2 DBD MAVE研究之间达到了87%的一致性。
- 为了进一步验证MAVE的结果,在另一项努力中,使用纪念斯隆凯特琳癌症中心的IMPACT肿瘤测序数据集评估功能性致病变异是否在BRCA2位点显示出杂合性缺失(LOH),这是驱动突变应表现出的现象。
- 确实,在IMPACT研究中鉴定出的PStrong变异中有85%显示BRCA2 LOH,而BStrong变异中仅25%显示BRCA2 LOH,这表明在具有功能性PStrong SNVs的肿瘤中,野生型第二个BRCA2等位基因的丢失显著富集。
Para_05
- 病例对照关联分析证实,仅 PStrong 类单核苷酸变异(SNVs)以及结合的 PStrong、PModerate 和 PSupporting 类 SNVs 与临床高危人群、基于人群的研究个体以及非裔美国人个体中乳腺癌风险增加相关。
- 这些 SNVs 还与临床高危人群中卵巢癌风险增加有关。
- 尽管在临床高危分析中使用了公开可用的参考对照,但结果的一致性确认了癌症发病风险的增加。
- 在不同人群中观察到的相似效应表明这些变异可能会在所有人群中增加风险。
- 值得注意的是,仅 PStrong 类和 PStrong、PModerate 及 PSupporting 类错义 SNVs 对于乳腺癌和卵巢癌的风险低于无义变异的风险。
- 然而,在临床队列中,502 个后验致病概率 ≥95% 的变异中有 380 个(76%),以及基于人群队列中 502 个概率 ≥95% 的变异中有 299 个(60%)与乳腺癌高风险(比值比 > 4.0)相关,类似于无义变异的风险。
- 其余 24%-40% 的错义变异与乳腺癌或卵巢癌的减弱中等风险相关。
- 这种减弱表明许多错义变异对功能的影响较小,并且癌症风险较低,或者部分是由于功能性数据的内在变异性所致。
- 未来对 BRCA2 单核苷酸变异的研究需要验证某些变异亚组的降低风险和/或存在低外显率变异的可能性,这可能需要修改风险咨询和患者管理的方法。
Para_06
- MAVE研究存在一些局限性。
- 功能评估中的小误差仍可能导致部分变异分类不准确。
- 预计通过进一步的研究以及与其他功能检测数据集的比较,可以解决一些剩余的临床意义未明变异(VUS),并验证在单倍体HAP1细胞中获得的结果。
- 尽管本研究未进行RNA研究,但已证明某些位于经典剪接位点、内含子区域或具有高SpliceAI评分的单核苷酸变异(SNVs)具有功能性致病性,这表明这些变异可能导致RNA异常剪接和蛋白质截短。
- 对于这些变异的进一步研究,虽然超出了本研究的范围,但仍需确定其是否通过异常剪接产生影响。
Para_07
- 总之,使用细胞存活测定法对BRCA2基因编码DBD突变热点的外显子中的单核苷酸变异(SNVs)进行了表征,以评估其对BRCA2活性的影响。
- 针对99%的所有SNVs生成的功能图谱使得能够区分核酸水平和蛋白质水平的功能异常,并导致对6,000多个个体变异进行临床分类。
- 未来,通过与其他数据集的整合,这些数据将有助于对所有种族和民族背景个体中该基因位点的所有变异进行特征分析和分类,并适用于所有与BRCA2相关的癌症形式。
Methods
Cell line and reagents
细胞系和试剂
Para_01
- HAP1细胞(Horizon Discovery提供)在含有10%胎牛血清和1%青霉素-链霉素的IMDM培养基中培养。
- 对于单倍体分选,1×10^7个HAP1细胞被重悬于5 mg/ml Hoechst 34580(BD公司,产品编号565877)中,并在4°C下进行分选。
- HAP1细胞使用TurboFectin 8.0(Origene公司)进行转染。
- 所有寡核苷酸和引物均由Integrated DNA Technologies合成。
Generation of site-saturation mutagenesis libraries and Cas9–sgRNA plasmids
位点饱和突变文库和Cas9–sgRNA质粒的构建
Para_01
- 选择编码 BRCA2 DNA 结合域的外显子 15-26,以及每个外显子上下游相邻的 10 个碱基对的内含子区域进行基因组编辑。
- 由于外显子 18 和 25 的尺寸较大,它们被分为氨基末端靶向区和羧基末端靶向区,最终形成了总共 14 个 SGE 靶向区域。
- 使用 Benchling 设计工具设计了多个单导向 RNA(sgRNA)。
- 通过 BbsI 酶切后,将 sgRNA 退火的寡核苷酸片段连接到 pSpCas9(BB)-2A-Puro (PX459 v.2.0) 载体上,构建每个单独 SGE 的 Cas9-sgRNA 共表达载体。
- 对于每个 SGE,从野生型 HAP1 基因组 DNA 中扩增出目标区域上下游 600-1,000 bp 的同源臂,并克隆到经 BamHI-HF 酶切的 pUC19 载体中,使用 NEBuilder HiFi DNA 组装克隆试剂盒完成操作。
- 通过反向 PCR 使用突变密码子 NNN 引物对克隆的质粒骨架进行定点饱和突变,以实现每个氨基酸位置的所有可能核苷酸变化。
- 通过定点突变引入一个编码沉默突变的原间隔序列保护编辑,修改目标区域的原间隔序列邻近基序位点或 sgRNA 识别位点,防止成功编辑后被 Cas9-sgRNA 再次切割。
- 此外,在每个同源臂的内含子中引入了一个单一的三核苷酸突变,以便于特异性重新扩增靶向 DNA。
CRISPR–Cas9 SGE
CRISPR–Cas9 单引导RNA (sgRNA)
Para_01
- 在HAP1细胞中预测具有高编辑效率的多个sgRNAs在每个目标区域的SGE实验中进行了评估,并选择了最优的sgRNAs。
- 在每个SGE实验中,500万个单倍体分选的HAP1细胞与4毫克的目标特异性变异库和16毫克的Cas9–sgRNA靶向构建物共转染。
- 细胞在嘌呤霉素(1毫克/毫升)中选择3天。
- 细胞在D0、D5(嘌呤霉素选择后24小时)和转染后D14收集,并使用Monarch基因组DNA纯化试剂盒(New England Biolabs,T3010L)提取gDNA。
- 目标区域通过PCR扩增以添加用于多重分析的条形码。
- 所有PCR反应均在50微升反应体系中进行,使用Q5高保真2×主混合液(New England Biolabs,M0492L)。
- gDNA扩增的引物见补充表2。
- 所有反应在测序前使用Ampure XP磁珠进行清理和浓缩,在Illumina MiSeq(每轮约500万次读取)或NextSeq(每轮约3000万次读取)仪器上进行150个循环的测序。
- 碱基呼叫由仪器控制软件完成,并使用定制算法进一步处理。
Sequencing data processing
测序数据处理
Para_01
- 来自 Illumina MiSeq 或 NextSeq 测序的样本的 FASTQ 文件使用 cutadapt(v.3.5)进行了接头序列修剪。
- SeqPrep(v.1.2)将成对末端读段转换为单端读段。
- 单端读段通过 bwa-mem(v.0.7.17)比对到人类参考基因组(GRCh38)。
- 比对后,使用自开发工具 CountReads 进行 DNA 测序数据分析,重点关注突变的识别和表征。
- CountReads 包括准备参考氨基酸和 DNA 序列、验证测序数据完整性和精确修剪读段至相关区域。
- 该方法还区分了变异类型,确认特定变异的存在,并汇总和报告变异数据。
- CountReads 生成了一个变异呼叫格式(VCF)文件,该文件使用 CAVA 进行注释。
- SpliceAI 工具(v.1.3.1)用于评估所有观察到的单核苷酸变异(SNVs)相关的剪接效应。
Functional read count process
功能读取计数过程
Para_01
- 使用 D14 和 D0 读取计数频率的 log2 比值来衡量每个变异体的耗尽或富集效应。
- 通过实验中 D0 和 D5 的比较,使用 Loess 转换进行位置调整。
- 在 D0 和 D5 中读取计数过低(<10)的变异体被排除在进一步分析之外。
- 在每个外显子内,根据重复实验中的中位沉默突变和中位无义突变 SNV 值,线性缩放变异体的 log2 比值。
- 对于每个变异体,计算所有重复实验中非缺失值的平均得分。
- 使用中位同义和无义值对跨外显子的得分进行线性缩放以标准化,类似于外显子内的归一化方法。
- 在完成所有数据清理和质量控制后,共有 6,959 个 SNV 获得了原始功能得分。
VarCall model for assessment of evidence of pathogenicity
用于评估致病性证据的VarCall模型
Para_01
- 在每个检测时间点(D0、D5 和 D14),通过将变异读取计数除以每个外显子的复制总数来计算复制水平的变异频率。
- 为了消除位置偏差,使用 D0 和 D5 读取计数之间的比率估计位置效应,采用具有外显子特异性自适应样条的复制水平广义加性模型。
- VarCall 模型应用于调整位置后的 D14 和 D0 读取计数的对数比值。
- VarCall 是一种贝叶斯分层模型类别,其中包含上下文特定的测量模型,并嵌入了用于变异效应的高斯双成分混合模型。
- 此处使用的公式基于之前对 BRCA2 变异的分析结果。
- 每个变异都被分配了一个二元指示符以表示其致病性状态:如果假设已知,则确定性分配;如果未知,则概率性分配。
- 沉默变异被认为无害,而无义变异被认为致病。
- 测量模型通过包含复制与外显子级别的位置和尺度随机效应来调整批次效应,并包括 t 分布误差项以允许异常值存在。
- 使用 JAGS 语言指定并拟合 VarCall 模型,采用 MCMC 算法进行计算。
- 所有相关计算均在 R 编程语言中完成。
- 基于 AlphaMissense 预测,DNA 结合区域中致病变异的频率为 0.23,因此该区域的变异致病性先验概率设为 0.2。
- 利用 MCMC 输出结果,计算每个变异支持致病性的贝叶斯因子。
- 根据致病性或良性证据强度(PStrong、PModerate 或 PSupporting,VUS,BStrong、BModerate 或 BSupporting),贝叶斯因子的阈值从 ACMG–AMP 指南的贝叶斯解释中得出。
- 分析的详细信息可在补充方法中找到。
Three-dimensional structural modelling
三维结构建模
Para_01
- 使用 PyMol 软件在 DBD 中映射了 BRCA2 功能性的强错义变异。
- 蛋白质数据库源文件(标识符 1MJE)从 NCBI 分子建模数据库下载。
- 三维结构建模基于 BRCA2-DSS1-ssDNA 复合物的晶体结构。
Multi-species amino-acid sequence conservation and in silico pathogenicity prediction
多物种氨基酸序列保守性与计算机预测致病性
Para_01
- BRCA2 氨基酸序列从 Align-GVGD (http://agvgd.hci.utah.edu/) 获取。
- 序列比对使用了十个物种:智人、黑猩猩、猕猴、挪威鼠、家犬、牛、灰短尾 opossum、鸡、非洲爪蟾和斑马鱼。
- 在包含 BRCA2 DNA 结合结构域功能致病性变异的氨基酸残基上进行了序列保守性分析。
- 使用 Align-GVGD、AlphaMissense 和 Bayes-Del 进行了计算机致病性预测。
Study populations
研究人群
Para_01
- 乳腺癌和卵巢癌病例及其相关的临床表型来自接受 Ambry Genetics 癌症基因检测的个体。
- 公开可用的参考对照组为 gnomAD(v.2.1、v.3.1 和 v.4,排除 UK Biobank)中的女性数据。
- 此外,还从基于人群的乳腺癌研究 CARRIERS 和 BRIDGES 项目中获得了匹配的乳腺癌病例-对照数据,以及来自 UK Biobank 的乳腺癌病例-对照数据(www.ukbiobank.ac.uk)。
- 等位基因频率大于 0.001 的变异被排除在分析之外。
Comparison with other BRCA2 functional assays
与其他BRCA2功能检测方法的比较
Para_01
- SGE功能结果与来自其他研究的结果进行了比较,包括基于BRCA2缺乏的细胞HDR测定、基于BRCA2缺乏的细胞系药物测定、基于先导编辑的SGE研究以及基于小鼠胚胎干细胞的功能分析。
ACMG–AMP framework for classification of BRCA2 DBD variants
ACMG-AMP框架用于分类BRCA2 DBD变异
Para_01
- ACMG-AMP基于规则的框架结合了来自人群、计算与预测、分离分析、功能以及其他数据的证据,每个证据来源分别被加权为非常强(PVS1)、强(PS1、PS2、PS3 和 PS4)、中等(PM1、PM2、PM3、PM4、PM5 和 PM6)或支持性(PP1、PP2、PP3、PP4 和 PP5)的致病证据,或者单独成立(BA1)、强(BS1、BS2、BS3 和 BS4)或支持性(BP1、BP2、BP3、BP4、BP5、BP6 和 BP7)的良性证据。
- 综合这些数据可以得出变异分类,包括良性、可能良性(LB)、致病性、可能致病性(LP)和意义不明(VUS)。
- 在本研究中,使用由ClinGen BRCA1/2 变异临床评估项目(VCEP)制定的ACMG-AMP评分规则对BRCA2 DNA结合结构域(DBD)单核苷酸变异(SNVs)进行临床分类。
- BRCA2功能数据根据PS3/BS3规则整合到ClinGen-ACMG-AMP BRCA1/2 VCEP分类模型中。
- 为了防止仅凭功能证据得出可能良性或可能致病性的分类,功能证据的值在对数尺度上被限制在+4和-4之间。
- 该研究获得了西方机构审查委员会的批准,该委员会豁免了对临床检测队列的审查,并得到了梅奥诊所机构审查委员会(编号21-008216)的批准。
- 本研究所使用的详细ACMG-AMP标准见补充方法部分。
Tumour LOH analysis
肿瘤丢失杂合性分析
Para_01
- 通过使用 IMPACT 数据集的肿瘤-正常配对测序,获取了携带生殖系 BRCA2 DBD 变异的乳腺、卵巢、胰腺和前列腺癌肿瘤的 LOH 状态。
- 使用 FACETS 算法从匹配的肿瘤-正常对中确定 LOH。
- 仅将肿瘤含量 >40% 的肿瘤样本纳入分析。
Statistical analysis
统计分析
Para_01
- 对 BRCA2 中变异分类组与乳腺癌或卵巢癌风险之间的关联性分析,是在接受 Ambry Genetics 基因检测的女性以及 gnomAD(v.2.1、v.3.1 和 v.4 版本,排除 UK Biobank 数据)中无癌症的女性中进行的,使用了加权逻辑回归方法,并根据病例中不同种族和族裔的相对频率进行了加权。
- 在基于人群的 CARRIERS 和 BRIDGES 研究中,匹配的乳腺癌病例和未受影响的女性(作为对照组),以及 UK Biobank 的乳腺癌病例和对照组的关联性分析,则采用 Fisher 精确检验完成。
- 对于功能致病性和良性变异之间的表型比较,定量变量使用 Student t 检验,定性变量使用卡方检验进行分析。
- 通过将 OR 估计值与年龄特异性的乳腺癌或卵巢癌(包括卵巢或输卵管恶性上皮肿瘤)发病率相结合,估算了不同分类组到 80 岁时的终身绝对风险,数据来源为美国国家癌症研究所的 SEER 计划(限于非西班牙裔白种人个体),同时考虑了全因死亡率。
- 为了比较其他 BRCA2 功能检测中的功能性类别得分差异,进行了单因素方差分析测试。
- 在肿瘤 LOH 分析中使用了 Fisher 精确检验。
- 所有分析均使用 R 软件(v.4.2.2)完成,所有检验均为双侧检验。
- 条形图或散点图中的 SGE 数据以重复实验的平均值形式呈现。
Ethics statement
伦理声明
Para_01
- 本文中展示的所有数据均在获得机构审查委员会批准后,得到了研究参与者的明确书面同意。
Reporting summary
报告摘要
Para_01
- 有关研究设计的更多信息可在与本文相关联的《自然组合》报告摘要中获取。
Data availability
Para_01
- 文章和/或补充方法中呈现的所有数据均可在文章中找到,或从基因表达综合数据库(标识符 GSE270424)获取。
Code availability
Para_01
- VarCall 模型及相关统计分析的所有代码均可从 GitHub 获取(https://github.com/najiemayo/Couch_SGE_BRCA2)。

