GWAS分析完,要做单倍型图,还要做单倍型的显著性分析?
GWAS分析完,要做单倍型图,还要做单倍型的显著性分析?
邓飞
发布于 2025-02-20 20:39:36
发布于 2025-02-20 20:39:36
大家好,我是邓飞。
GWAS分析完成后,进行单倍型图分析的核心目的是验证显著性位点的可靠性并深入理解其遗传背景,具体原因包括以下几点:
- 排除假阳性结果 GWAS发现的显著性位点可能因多重检验或群体结构干扰产生假阳性。单倍型图通过分析显著性位点上下游区域的连锁不平衡(LD)结构,判断这些位点是否处于高度连锁的区块(Block)中。若基因位于Block内且被高LD区域覆盖,则说明该基因的关联性更稳定,假阳性风险显著降低。
- 识别连锁区域与单倍型结构 单倍型图可直观展示SNP之间的LD关系,划分Block(如通过D’或r²阈值)。Block内的SNP组合形成少数单倍型,这些单倍型代表共同遗传的DNA片段。若目标基因或标记位于Block内,其功能或调控作用更可能因连锁效应而真实存在。
- 辅助基因定位与功能验证 当GWAS位点位于非编码区时,单倍型图可帮助锁定候选基因的调控区域。例如,若Block覆盖某基因的启动子区,可推测该基因通过调控表达水平影响表型,从而指导后续实验验证方向。
- 优化分子标记选择 在分子育种或医学遗传学中,单倍型图可筛选出Block内代表性SNP(如标签SNP),减少冗余检测成本,同时保留遗传信息完整性。
1,比较流行的图,Block + GWAS pvalue + LD result
- 最下方的热图是两两SNP之间的LD值,越高越红,比较红的区域构成一个Block(用黑线连起来)
- 如果提供gff文件,可以显示基因的上游、下游、外显子、内含子区域
- 上面是位点的曼哈顿图,是区域性的曼哈顿图
- 位点之间,也可以根据LD值进行可视化,以最显著的位点为四方形,其它位点与其LD值的大小呈现不同的颜色
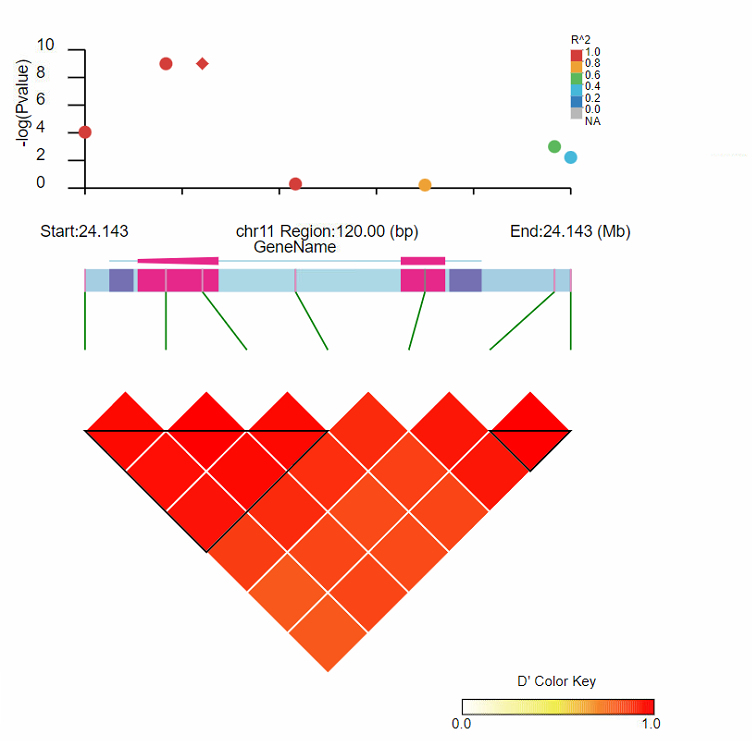
参考代码:
LDBlockShow -InVCF Test.vcf.gz -OutPut re2 -Region chr11:24100000:24200000 -InGWAS gwas.pvalue -OutPng -SeleVar 1
结果:
re2.blocks.gz re2.png re2.site.gz re2.svg re2.TriangleV.gz
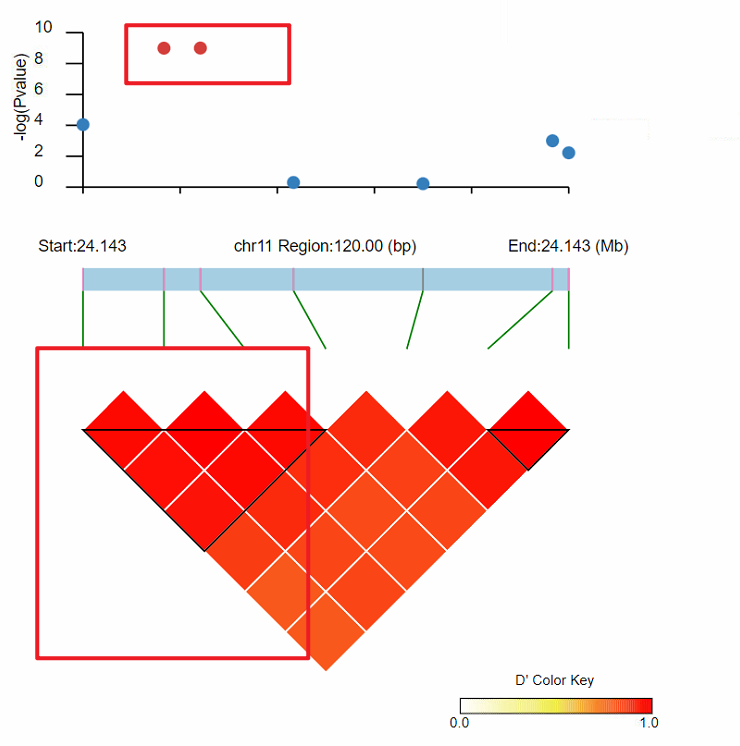
ShowLDSVG -InPreFix re2 -OutPut temp -InGWAS gwas.pvalue -Cutline 7 -ShowNum -PointSize 3
命令:
LDBlockShow -InVCF Test.vcf.gz -OutPut re3 -Region chr11:24100000:24200000 -InGWAS gwas.pvalue -OutPng -SeleVar 1 -InGFF In.gff
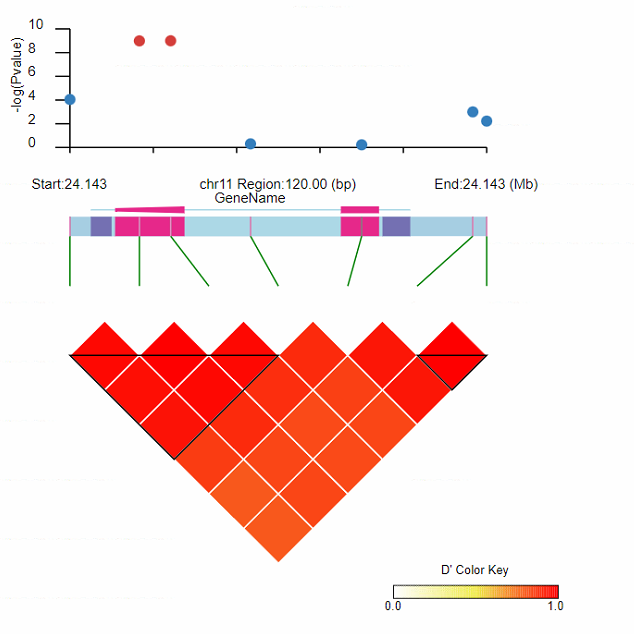
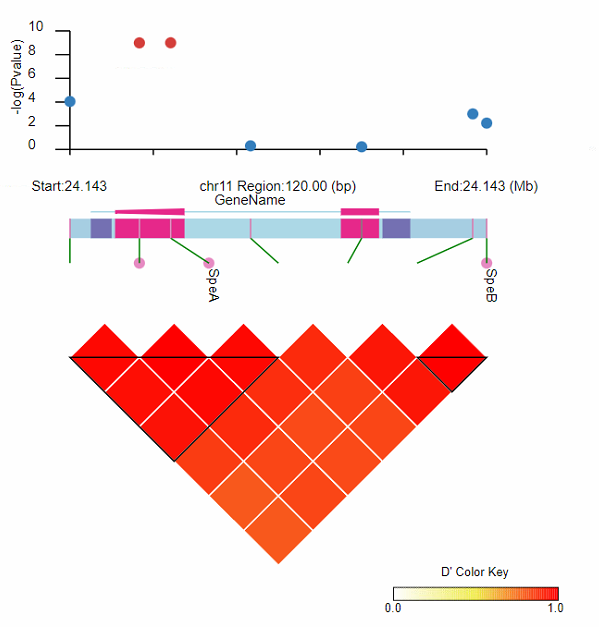
也可以增加SNP的名称:
$ cat Spe.snp
chr11 24142660
chr11 24142669 SpeA
chr11 24142760 SpeB
命令:
LDBlockShow -InVCF Test.vcf.gz -OutPut re3 -Region chr11:24100000:24200000 -InGWAS gwas.pvalue -OutPng -SeleVar 1 -InGFF In.gff -SpeSNPName Spe.snp
2,比较流行的图,Haploview + 箱线图 + 显著性分析 + 单倍型频率统计
进一步分析呢?比如将单倍型block看一下不同样本的分布,统计一下不同单倍型和表型数据的关系,做一个显著性分析不是更有说服力吗?特别是找到的单倍型应用于育种时,通过单倍型的显著性分析更有说服力:
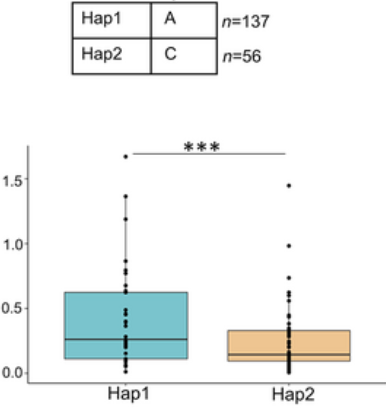
优势单倍型,优良基因,呼之欲出,一图胜千言,这样更有说服力。
如何操作呢?
1,划分单倍型,给出单倍型的类型有几类
2,统计单倍型的频率,每个单倍型有多少样本
3,对单倍型和表型数据进行显著性检验,如果是两个单倍型就用T检验,如果是3个及以上,就用方差分析
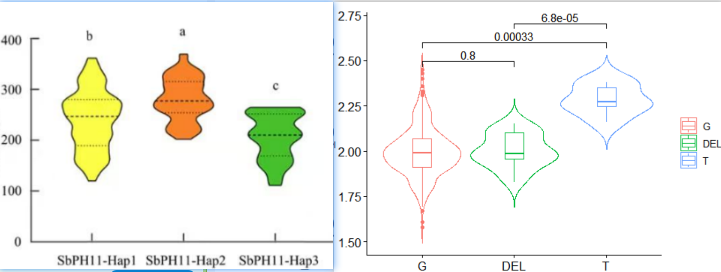
4,对结果可视化就是上面图的结果了,有时候还有小提琴图的样式:
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-02-19,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

