HOMER - motif 挖掘和分析
HOMER - motif 挖掘和分析

分析原理
1. 预处理(Preprocessing)
(1) 序列提取(Extraction of Sequences)
- 功能: 从输入的基因组区域或基因启动子区域中提取DNA序列。
- 输入类型:
- 基因组区域:提供BED格式的peak文件(如ChIP-seq的peak区域),HOMER会从参考基因组FASTA文件中提取对应序列。
- 基因启动子区域:提供基因ID(如RefSeq或Ensembl ID),HOMER会从预定义的启动子序列表中提取对应区域(默认从TSS上游2kb到下游500bp)。
- 工具:
findMotifs.pl:适用于启动子分析(基于基因ID)。findMotifsGenome.pl:适用于基因组区域分析(基于BED文件)。
说明:motif 分析简单说明就是在我们关注的序列中找规律。在 homer 分析时,一般我们只需要提供BED文件就行,它会自动提取对应的序列。
(2) 背景序列选择(Background Selection)
- 功能: 选择背景序列用于与目标序列(输入peak区域)进行比较,以识别显著富集的motif。
- 默认行为:
- 若未指定背景文件(
-bg <file>),HOMER会自动从基因组中随机选择与目标序列数量相同、GC含量匹配的区域作为背景。 - 背景序列的选择会尽量匹配目标序列的GC含量分布,以避免GC偏差对结果的影响。
- 若未指定背景文件(
- 高级控制:
说明:一个基序可能被认为是“统计显著”的,即它的出现频率与随机机会有所不同。在基序富集分析中,目标序列(如ChIP-seq峰或感兴趣的基因组区域)需要与一组背景序列进行比较,以确定哪些基序在目标序列中显著富集。背景序列代表了“随机”或“非特异性”的基因组区域,用于评估目标序列中基序的出现是否具有统计学意义。基因组的不同区域可能具有不同的序列特征(如重复序列、基因密度等),随机或匹配的背景序列可以消除这些偏差。
- HOMER2提供了更多背景序列选择的控制选项,特别是在需要控制位置偏差(positional sequence bias)时。
- 例如,可以通过
-bg <file>指定自定义背景文件(如对照样本的peak区域)。
(3) GC含量归一化(GC Normalization)
- 功能: 将目标序列和背景序列按GC含量分箱(5%间隔),并对背景序列进行加权,使其GC含量分布与目标序列一致。
- 目的: 避免因目标序列和背景序列的GC含量差异导致假阳性结果(如富集到高GC含量的motif)。
- 特殊选项:
说明:如果不进行GC含量归一化,高GC或低GC区域可能会导致假阳性或假阴性结果。
- 若分析CpG岛区域,可使用
-cpg参数进行CpG%归一化(而非GC%归一化)。
- 若分析CpG岛区域,可使用
(4) 自动归一化(Autonormalization)
- 功能: 进一步消除目标序列和背景序列之间的短寡核苷酸(short oligo)频率差异(如AA、TT等二核苷酸)。
- 背景: 目标序列中可能存在其他序列偏差(如密码子偏好性、实验偏好性等),这些偏差会导致HOMER误判为显著motif。
- 实现方法:
- HOMER通过调整背景序列的权重,使目标序列和背景序列的短寡核苷酸频率差异最小化。
- 使用“爬山算法”(hill-climbing)逐步优化背景序列权重,避免权重极端化。
- 参数控制:
- 通过
-nlen <#>指定短寡核苷酸的长度(默认2,即二核苷酸)。
- 通过
2. Motif发现流程
(5) 解析输入序列为Oligo表(Parsing input sequences into an Oligo Table)
- 功能: 将输入序列解析为指定长度的寡核苷酸(oligo),并构建Oligo表。
- Oligo表的作用:
- 记录每个唯一oligo在目标序列和背景序列中的出现次数。
- 通过Oligo表,HOMER可以高效地搜索motif(motif本质上是oligo的集合)。
- 局限性:
- Oligo表会破坏单个oligo与其来源序列的关联。
(6) Oligo自动归一化(Oligo Autonormalization,可选)
- 功能: 在Oligo表上应用自动归一化,进一步消除短寡核苷酸频率的偏差。
- 适用场景:
- 当目标序列中存在极端序列偏差时(如A-rich或T-rich区域)。
- 参数控制:
- 通过
-olen <#>指定Oligo的长度(默认与motif长度一致)。
- 通过
(7) 全局搜索阶段(Global Search phase)
- 功能: 在Oligo表中全局搜索显著富集的oligo。
- 实现方法:
- 首先筛查每个可能的oligo的富集情况。
- 为提高灵敏度,允许oligo中存在错配(通过
-mis <#>控制错配数量)。 - 跳过不显著的oligo(如背景实例多于目标实例的oligo)。
(8) 矩阵优化(Matrix Optimization)
- 功能: 将显著富集的oligo转换为位置特异性概率矩阵(Position Specific Probability Matrix, PSPM),并通过局部优化算法进一步优化。
- 实现方法:
- 对每个oligo单独优化,生成motif概率矩阵。
- 通过调整检测阈值,最大化motif在目标序列中的富集。
- 重复优化过程,直到富集值无法进一步提高。
(9) 掩蔽与重复(Mask and Repeat)
- 功能: 在发现一个motif后,掩蔽其结合位点,继续搜索下一个motif。
- HOMER2的改进:
- 不仅掩蔽motif位点,还掩蔽其相邻区域,避免重复发现相似的motif。
- 可通过
-quickMask恢复旧版掩蔽方式,或通过-homer1使用旧版HOMER。
3. 已知motif筛查
(10) 加载motif库(Load Motif Library)
- 功能: 加载已知motif库(如JASPAR、HOMER内置库),用于筛查目标序列中的已知motif。
- 自定义motif库:
- 通过
-mknown <file>指定自定义motif文件,或编辑data/knownTFs/known.motifs。
- 通过
(11) 筛查每个motif(Screen Each Motif)
- 功能: 扫描目标序列中的已知motif实例,计算其富集显著性。
- 统计方法:
- 使用超几何分布或二项分布计算motif的富集p值。
- 默认使用二项分布(适用于大规模数据集),可通过
-h强制使用超几何分布。
4. 结果输出
(12) Motif文件(Motif Files)
- 功能: 输出motif文件(
.motif),包含motif的概率矩阵和富集统计信息。 - 文件格式:
- 第一行为motif信息(如一致序列、名称、检测阈值、富集p值等)。
- 后续行为位置特异性概率矩阵(A/C/G/T的概率)。
(13) De novo motif输出(De novo motif output)
- 功能: 生成HTML报告(
homerResults.html),展示de novo发现的motif及其富集信息。 - 报告内容:
- Motif的富集p值、一致序列、位置分布图等。
- 与已知motif的比对结果(需谨慎解读)。
(14) 已知motif输出(Known motif output)
- 功能: 生成HTML报告(
knownResults.html),展示已知motif的富集信息。 - 报告内容:
- 已知motif的富集p值、结合位点数量等。
举例
假设我们有一组ChIP-seq实验的峰值区域(目标序列),希望从中发现显著富集的转录因子结合基序(motif)。以下是HOMER的motif发现流程:
1. 输入数据准备
- 目标序列:ChIP-seq实验的峰值区域(如
peaks.bed文件)。 - 背景序列:从基因组中随机选择的与目标序列长度和分布相似的区域(如
background.bed文件)。如果不指定,HOMER 会根据规则自动选择。
2. Motif发现流程
(1) 解析输入序列为Oligo表(Parsing input sequences into an Oligo Table)
- 功能: HOMER将目标序列和背景序列解析为指定长度的寡核苷酸(oligo),并构建Oligo表。
- 示例:
- 目标序列oligo:
ACGT、CGTA、GTAC、TACG。 - 背景序列oligo:
TGCA、GCAA、CAAC、AACG。 - 假设目标序列为
ACGTACGT,背景序列为TGCAACGT。 - 如果oligo长度为4,HOMER会生成以下oligo:
- Oligo表记录每个oligo在目标序列和背景序列中的出现次数。
- 目标序列oligo:
- 作用:
- Oligo表是motif发现的基础,HOMER通过分析oligo的富集情况来识别motif。
(2) Oligo自动归一化(Oligo Autonormalization,可选)
- 功能: 对Oligo表进行归一化,消除序列偏差(如A-rich或T-rich区域)。
- 示例:
- 如果目标序列中存在大量A-rich区域,HOMER会调整oligo的频率,使其与背景序列一致。
- 参数:
- 通过
-olen <#>指定oligo长度(默认与motif长度一致)。
- 通过
(3) 全局搜索阶段(Global Search phase)
- 功能: 在Oligo表中全局搜索显著富集的oligo。
- 示例:
- HOMER会计算每个oligo在目标序列和背景序列中的富集情况。
- 例如,oligo
ACGT在目标序列中出现10次,在背景序列中出现2次,HOMER会计算其富集显著性(如p值)。 - 为提高灵敏度,HOMER允许oligo中存在错配(通过
-mis <#>控制错配数量)。
(4) 矩阵优化(Matrix Optimization)
功能: 将显著富集的oligo转换为位置特异性概率矩阵(PSPM),并通过局部优化算法进一步优化。
示例:
- 假设oligo
ACGT显著富集,HOMER会将其扩展为一个4bp的motif概率矩阵: 复制A: 0.9 0.1 0.1 0.1C: 0.1 0.8 0.1 0.1G: 0.1 0.1 0.9 0.1T: 0.1 0.1 0.1 0.8 - HOMER通过调整检测阈值,最大化motif在目标序列中的富集。
(5) 掩蔽与重复(Mask and Repeat)
- 功能: 在发现一个motif后,掩蔽其结合位点,继续搜索下一个motif。
- 示例:
- 假设发现motif
ACGT,HOMER会掩蔽目标序列中所有ACGT位点及其相邻区域。 - 然后,HOMER重复上述过程,搜索下一个显著富集的motif。
- 假设发现motif
3. 已知motif筛查
(1) 加载motif库(Load Motif Library)
- 功能: 加载已知motif库(如JASPAR、HOMER内置库)。
- 示例:
- HOMER加载内置的
known.motifs文件,其中包含已知转录因子的motif信息。
- HOMER加载内置的
(2) 筛查每个motif(Screen Each Motif)
- 功能: 扫描目标序列中的已知motif实例,计算其富集显著性。
- 示例:
- 假设已知motif
ACGT在目标序列中出现20次,在背景序列中出现5次,HOMER会计算其富集p值(如p=0.001)。
- 假设已知motif
4. 结果输出
(1) Motif文件(Motif Files)
功能: 输出motif文件(.motif),包含motif的概率矩阵和富集统计信息。
示例:
- 文件
homerResults/motif1.motif内容: 复制>ACGT p-value=1e-50.9 0.1 0.1 0.10.1 0.8 0.1 0.10.1 0.1 0.9 0.10.1 0.1 0.1 0.8
(2) De novo motif输出(De novo motif output)
- 功能: 生成HTML报告(
homerResults.html),展示de novo发现的motif及其富集信息。 - 示例:
- 报告显示motif
ACGT的富集p值、一致序列、位置分布图等。
- 报告显示motif
(3) 已知motif输出(Known motif output)
- 功能: 生成HTML报告(
knownResults.html),展示已知motif的富集信息。 - 示例:
- 报告显示已知motif
ACGT的富集p值、结合位点数量等。
- 报告显示已知motif
子程序
findMotifs.pl
- 默认用途:
- 分析启动子区域的 motif。
- 默认假设输入是基因组坐标文件(如 ChIP-seq 峰值文件),并提取这些坐标附近的启动子序列(通常是转录起始位点上游 1000 bp 到下游 100 bp)进行 motif 分析。
- 输入:
- 可以是基因组坐标文件(如 BED 或 peak 文件)。
- 也可以是 FASTA 文件(直接分析序列中的 motif)。
- 默认行为:
- 如果输入是基因组坐标文件,HOMER 会提取每个坐标附近的启动子序列。
- 如果输入是 FASTA 文件,HOMER 会直接分析这些序列中的 motif。
- 输出:
- 默认输出 motif 的富集结果、序列标识图(sequence logos)等。
- 适用场景:
- 分析启动子区域中的 motif。
- 分析特定序列(如 FASTA 文件)中的 motif。
findMotifsGenome.pl
- 默认用途:
- 在全基因组范围内分析 motif。
- 默认假设输入是基因组坐标文件(如 ChIP-seq 峰值文件),并直接提取这些坐标对应的基因组序列进行 motif 分析。
- 输入:
- 必须是基因组坐标文件(如 BED 或 peak 文件)。
- 不支持直接输入 FASTA 文件。
- 默认行为:
- 从基因组中提取输入坐标对应的序列。
- 默认使用
-size 200,即从每个坐标的中心向两侧各扩展 100 bp,提取 200 bp 的序列。
- 输出:
- 默认输出 motif 的富集结果、序列标识图(sequence logos)等。
- 还可以生成基因组浏览器可视化的文件(如 BigWig 文件)。
- 适用场景:
- 分析全基因组范围内的 motif。
- 分析 ChIP-seq、ATAC-seq 等实验的峰值区域中的 motif。
使用场景
场景1: 使用基因 ID , 分析基因启动子区域是否有 Motif
findMotifs.pl 子程序
必要参数:
- 基因 ID
- 启动子集的名称
- 输出目录
必要步骤:
Step1: 准备基因 ID
HOMER 软件支持6种格式的 ID格式:
- NCBI Entrez Gene IDs
- NCBI Unigene IDs
- NCBI Refseq IDs (mRNA, protein)
- Ensembl Gene IDs
- Gene Symbols (i.e. Official Gene names, like "Nfkb1" )
- popular affymetrix probe IDs (MOE430, U133plus, U95, U75A)
我这里随机选一些 Entrez Gene IDs 演示说明
$ head EntrezID.txt
4312
8318
10874
55143
55388
991
6280
2305
9493
1062
Step2: 准备启动子集的名称
HOMER 软件有7个物种的默认的启动子集 :
- human (Homo sapiens)
- mouse (Mus musculus)
- rat (Rattus norvegicus)
- fly (Drosophila melanogaster)
- worm (Caenorhabditis elegans)
- zebrafish (Danio rerio)
- yeast (Saccharomyces cerevisiae)
不过这个基因集需要提前下载,具体下载命令在你运行命令的时候会提示你没有这个数据集,会提供基因集list和下载命令。
我这里服务器的命令是这样的
- 基因集
list
perl /home/data/zs347/micromamba/envs/bioinfo/share/homer/.//configureHomer.pl -list
会输出基因list
2025-03-05 14:14:11 (75.1 KB/s) - '/home/data/zs347/micromamba/envs/bioinfo/share/homer/.///update.txt' saved [19497/19497]
Updating Settings...
Packages with name conflicts have a trailing -o, -p, or -g
Version Installed Package Version Description
SOFTWARE
+ homer v5.1 Code/Executables, ontologies, motifs for HOMER
ORGANISMS
- zebrafish-o v7.0 Danio rerio (zebrafish) accession and ontology information
- fly-o v7.0 Drosophila melanogaster (fly) accession and ontology information
- dog v7.0 Canis lupus familiaris (dog) accession and ontology information
- ncrassa v7.0 Neurospora crassa (ncrassa) accession and ontology information
- diatom v7.0 Phaeodactylum tricornutum (diatom) accession and ontology information
- lamprey v7.0 Petromyzon marinus (lamprey) accession and ontology information
- mouse-o v7.0 Mus musculus (mouse) accession and ontology information
- arabidopsis-o v7.0 Arabidopsis thaliana (arabidopsis) accession and ontology information
- pig v7.0 Sus scrofa (pig) accession and ontology information
- laevis v7.0 Xenopus laevis (laevis) accession and ontology information
- ciona v7.0 Ciona intestinalis (ciona) accession and ontology information
软件包的末尾可能有“-p”(即“human-p”)、“-o”或“-g”。这些后缀用于在不同部分中有相同名称的软件包时帮助区分(例如,-p表示启动子)。总体而言,HOMER软件包有四种类型:
- 软件包(SOFTWARE) - 在这种情况下,只有HOMER代码。这种软件包包含所有代码以及一些通用数据文件,如基序矩阵。
- 有机体包(ORGANISMS) - 物种特定的软件包包含登录号转换数据、基因描述和特定于每个有机体的GO分析文件。大多数基于NCBI Gene数据库信息。
- 启动子包(PROMOTERS) - 启动子序列和相关文件,用于分析启动子的基序富集。大多数情况下基于RefSeq转录定义。名称中带有“-mRNA”的软件包包含用于分析RNA而非DNA的RNA序列。
- 基因组包(GENOMES) - 基因组序列和注释信息。
如果下载hg19基因组,它将自动下载“human”有机体包。每次下载启动子或基因组包时,都会检查确保您也拥有有机体包。
- 下载命令
perl /home/data/zs347/micromamba/envs/bioinfo/share/homer/.//configureHomer.pl -install human-p
HOMER也提供了自己构建启动子集的方式:http://homer.ucsd.edu/homer/introduction/update.html
这里我使用的物种是human。
Step3: 选择输出文件夹
Step4: 运行命令
findMotifs.pl 子程序的命令格式:
findMotifs.pl <输入文件.txt> <启动子集> <输出目录> [选项]
我们运行一下我们的示例
findMotifs.pl \
EntrezID.txt \
human \
LPSMotifResults/ \
-start -400 -end 100 -len 8,10 -p 4
输出结果:
-rw-rw-r-- 1 zs347 zs347 62K Mar 5 14:57 biocyc.txt
-rw-rw-r-- 1 zs347 zs347 1.9M Mar 5 14:57 biological_process.txt
-rw-rw-r-- 1 zs347 zs347 240K Mar 5 14:57 cellular_component.txt
-rw-rw-r-- 1 zs347 zs347 124K Mar 5 14:57 chromosome.txt
-rw-rw-r-- 1 zs347 zs347 572K Mar 5 14:57 cosmic.txt
-rw-rw-r-- 1 zs347 zs347 198K Mar 5 14:57 gene3d.txt
-rw-rw-r-- 1 zs347 zs347 807K Mar 5 14:57 geneOntology.html
-rw-rw-r-- 1 zs347 zs347 151 Mar 5 14:57 gwas.txt
-rw-rw-r-- 1 zs347 zs347 8.0K Mar 5 15:06 homerMotifs.all.motifs
-rw-rw-r-- 1 zs347 zs347 6.3K Mar 5 15:06 homerMotifs.motifs10
-rw-rw-r-- 1 zs347 zs347 1.8K Mar 5 15:03 homerMotifs.motifs8
drwxrwxr-x 2 zs347 zs347 4.0K Mar 5 15:07 homerResults/
-rw-rw-r-- 1 zs347 zs347 68K Mar 5 15:07 homerResults.html
-rw-rw-r-- 1 zs347 zs347 3.2M Mar 5 14:57 interactions.txt
-rw-rw-r-- 1 zs347 zs347 1.3M Mar 5 14:57 interpro.txt
-rw-rw-r-- 1 zs347 zs347 104K Mar 5 14:57 kegg.txt
drwxrwxr-x 2 zs347 zs347 4.0K Mar 5 15:03 knownResults/
-rw-rw-r-- 1 zs347 zs347 49K Mar 5 15:03 knownResults.html
-rw-rw-r-- 1 zs347 zs347 52K Mar 5 15:03 knownResults.txt
-rw-rw-r-- 1 zs347 zs347 151 Mar 5 14:57 lipidmaps.txt
-rw-rw-r-- 1 zs347 zs347 573K Mar 5 14:57 molecular_function.txt
-rw-rw-r-- 1 zs347 zs347 78 Mar 5 14:55 motifFindingParameters.txt
-rw-rw-r-- 1 zs347 zs347 151 Mar 5 14:57 msigdb.txt
-rw-rw-r-- 1 zs347 zs347 6.0K Mar 5 15:07 nonRedundant.motifs
-rw-rw-r-- 1 zs347 zs347 21K Mar 5 14:57 pathwayInteractionDB.txt
-rw-rw-r-- 1 zs347 zs347 492K Mar 5 14:57 pfam.txt
-rw-rw-r-- 1 zs347 zs347 108K Mar 5 14:57 prints.txt
-rw-rw-r-- 1 zs347 zs347 135K Mar 5 14:57 prosite.txt
-rw-rw-r-- 1 zs347 zs347 219K Mar 5 14:57 reactome.txt
-rw-rw-r-- 1 zs347 zs347 1.9K Mar 5 14:55 seq.autonorm.tsv
-rw-rw-r-- 1 zs347 zs347 73K Mar 5 14:57 smart.txt
-rw-rw-r-- 1 zs347 zs347 4.4M Mar 5 14:57 smpdb.txt
-rw-rw-r-- 1 zs347 zs347 39K Mar 5 14:57 wikipathways.txt
输出文件可分为四类:核心motif富集结果、已知motif比对结果、运行参数及中间文件,以及其他注释文件。
文件名 | 文件说明 | 来源/分析工具关联性 |
|---|---|---|
homerResults.html | HOMER核心结果文件,展示从头预测的motif富集分析结果,包含统计显著性(P值、q值)、序列占比、富集倍数等信息 | HOMER工具输出(核心文件) |
homerResults/ | 目录,存储每个motif的详细结果文件(如motif序列、可视化图、反向互补序列等) | HOMER工具输出 |
homerMotifs.all.motifs | 包含所有从头预测的motif信息,标准motif格式文件,可直接用于下游分析或与其他数据库比对 | HOMER工具输出 |
homerMotifs.motifs8/10 | 特定长度(如8bp、10bp)的motif子集文件,用于不同参数下的分析结果 | HOMER工具输出 |
knownResults.html | HOMER已知motif富集结果,展示与公共数据库(如JASPAR、HOCOMOCO)匹配的motif信息 | HOMER工具输出(已知motif库比对) |
geneOntology.html | 基因本体(GO)富集分析结果,通常包含生物过程(BP)、分子功能(MF)、细胞组分(CC)的注释信息 | 其他工具(如DAVID、clusterProfiler) |
biological_process.txt | 基因本体-生物过程(GO-BP)的文本格式富集结果 | GO分析输出 |
kegg.txt | KEGG通路富集分析结果,包含通路名称、富集P值、基因列表等信息 | KEGG数据库分析工具(如KOBAS) |
reactome.txt | Reactome通路数据库的富集分析结果 | ReactomePA等工具 |
interpro.txt | InterPro蛋白结构域注释结果,包含蛋白质家族、结构域等信息 | InterProScan工具 |
chromosome.txt | 染色体位置相关注释信息,可能包含基因/peak的染色体分布统计 | 基因组注释工具 |
cosmic.txt | COSMIC(癌症体细胞突变数据库)相关注释,可能关联癌症突变位点信息 | COSMIC数据库 |
interactions.txt | 基因/蛋白相互作用网络数据,可能包含相互作用类型、置信度等信息 | STRING数据库或Cytoscape导出 |
motifFindingParameters.txt | HOMER或其他motif分析工具的运行参数记录文件 | 分析日志文件 |
注:
- 部分文件(如gwas.txt、msigdb.txt、smpdb.txt)可能分别对应GWAS关联数据、MSigDB基因集、SMPDB代谢通路数据库的注释结果。
- 目录(如homerResults/、knownResults/)内的具体文件需结合HOMER文档进一步查看(例如motif序列文件、HTML可视化文件)。
- 若需完整分析解读,建议优先查看HOMER核心文件(homerResults.html、knownResults.html)及GO/KEGG通路富集结果,其他文件多为辅助注释。
主要说明几种文件
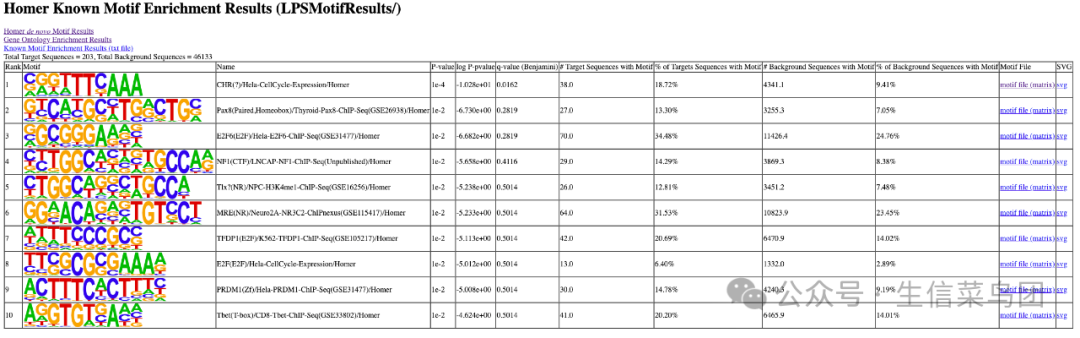
knownResults.html
表头 | 说明 |
|---|---|
Rank | 基序(Motif)在富集分析中的排名,数值越小表示富集程度越高。 |
Motif | 基序的 DNA 序列模式,可能包含简写符号(如 S 表示可变碱基)。 |
Name | 基序的名称,通常与已知的转录因子或调控元件相关,可能包含数据库来源。 |
P-value | 基序富集的统计学显著性,值越小表示富集越显著。 |
log P-value | P 值的对数形式(以 10 为底),用于更直观展示显著性差异。负值绝对值越大,富集越显著。 |
# Target Sequences with Motif | 目标序列中含有该基序的序列数量,数值越大表示该基序在目标中出现的次数越多。 |
% of Target Sequences with Motif | 目标序列中含有该基序的序列百分比,百分比越高表示该基序在目标中的富集程度越高。 |
% of Background Sequences with Motif | 背景序列中含有该基序的序列百分比,用于与目标序列对比,评估富集特异性。 |
MotifFile (matrix/svg) | 基序文件的存储格式: - matrix:权重矩阵,描述基序碱基偏好性; - svg:可视化图形。 |
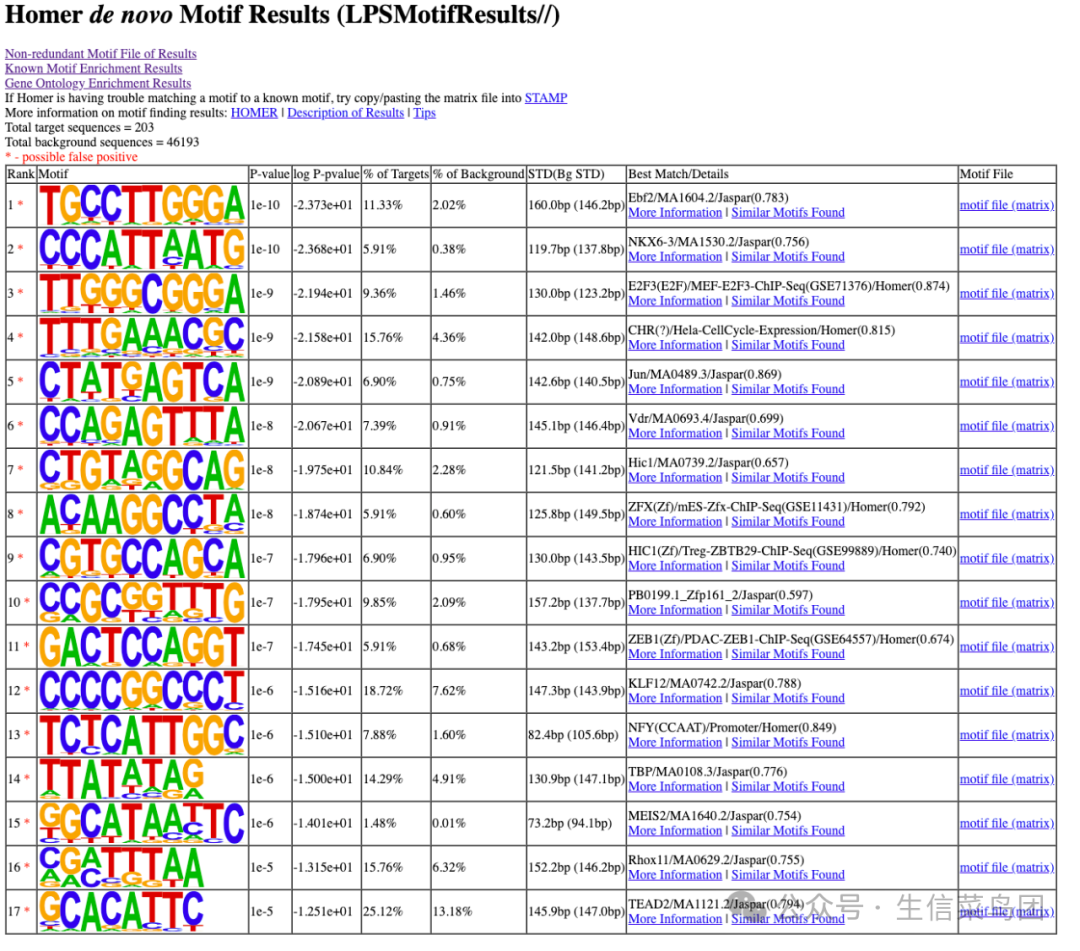
homerResults.html
表头 | 说明 |
|---|---|
Rank/Motif | - Rank: 基序(Motif)在富集分析中的排名,数值越小表示富集程度越高。 - Motif: 基序的 DNA 序列。 |
P-value | 基序富集的统计学显著性,值越小表示富集越显著。 |
% of Targets | 目标序列(如 ChIP-seq 峰区)中含有该基序的百分比,百分比越高表示基序在目标中富集程度越高。 |
% of Background | 背景序列(如随机基因组区域)中含有该基序的百分比,用于对比富集特异性。 |
STD(Bg STD) | 背景序列中基序频率的标准差(Standard Deviation),反映背景数据的离散程度。 |
Best Match/Details | 基序的最佳匹配信息: - 已知转录因子名称或数据库条目(如 JASPAR); - 匹配得分或相似度。 |
Motif File | 基序文件的存储格式: - matrix:权重矩阵文件,描述基序碱基偏好性; - svg:可视化文件。 |
其中每个基序都会有一个基序权重矩阵文件
>TGCCTTGGGA 1-TGCCTTGGGA 8.577033 -23.732495 0 T:23.0(11.33%),B:933.1(2.02%),P:1e-10 Tpos:253.5,Tstd:160.0,Bpos:247.2,Bstd:146.2,StrandBias:-0.2,Multiplicity:1.04
0.001 0.001 0.001 0.997
0.078 0.001 0.920 0.001
0.055 0.808 0.046 0.091
0.001 0.906 0.001 0.092
0.063 0.001 0.001 0.935
0.001 0.001 0.071 0.927
0.082 0.001 0.916 0.001
0.082 0.001 0.841 0.076
0.001 0.061 0.937 0.001
0.856 0.036 0.107 0.001
字段拆分及意义:
字段 | 说明 |
|---|---|
TGCCTTGGGA | Motif 的 DNA 序列(核心识别序列)。 |
1-TGCCTTGGGA | Motif 的编号和序列(1 表示排名第1)。 |
8.577033 | Motif 的富集得分(得分越高,富集越显著)。 |
-23.732495 | 对数似然比(Log Likelihood Ratio),负值绝对值越大表示与背景差异越大。 |
0 | 未明确标注,可能为保留字段或统计参数。 |
T:23.0(11.33%) | 目标序列中匹配该 Motif 的数量(23)及百分比(11.33%)。 |
B:933.1(2.02%) | 背景序列中匹配该 Motif 的数量(933.1,可能为均值)及百分比(2.02%)。 |
P:1e-10 | 富集显著性 P 值,此处为极显著(10⁻¹⁰)。 |
Tpos:253.5 | Motif 在目标序列中的平均位置(距峰顶 253.5 bp)。 |
Tstd:160.0 | 目标序列中 Motif 位置的标准差(160 bp)。 |
Bpos:247.2 | Motif 在背景序列中的平均位置(247.2 bp)。 |
Bstd:146.2 | 背景序列中 Motif 位置的标准差(146.2 bp)。 |
StrandBias:-0.2 | 链偏好性(-0.2 表示轻微偏向负链)。范围 [-1, 1],0 表示无偏好。 |
Multiplicity:1.04 | 每个序列中该 Motif 的平均出现次数(1.04 表示每条序列约出现1次)。 |
后续行表示 Motif 每个位置的碱基偏好性(概率或权重值),每行对应一个位置,四列依次为 A, T, C, G 的概率(范围 0~1)。
比如第一行的解读方法:
- 每行:代表 Motif 的一个位置(从5'到3'方向)。
- 每列:对应碱基的概率(A, T, C, G)。
示例(位置1)
- A: 0.001(0.1%)
- T: 0.001(0.1%)
- C: 0.001(0.1%)
- G: 0.997(99.7%)
- 结论:位置1高度偏好 G。
场景2: 查找 Motif 位置
默认情况下,HOMER 不会返回在基序发现过程中发现的每个基序的位置。要恢复基序位置,您必须首先通过获取 HOMER 输出的“基序文件”来选择您感兴趣的基序。
方法1: findMotifs.pl 子程序
-find 选项
- 主要用途:在已发现的 motif 或自定义的 motif 文件中,扫描目标区域(如 ChIP-seq 峰或差异基因的启动子区域)并输出每个 motif 实例的 精确位置、序列、匹配得分 等信息。
命令格式:
findMotifs.pl <目标区域文件> <物种> <输出目录> -find <motif文件> [其他参数] > 输出结果.txt
必要步骤:
step1: 准备目标区域文件
那我们还是使用基因ID,然后制定启动区域 -start -400 -end 100
step2: 选择物种
step3: 指定输出目录
step4: 输入motif矩阵文件
选择这个吧,也可一下指定多行
>TGCCTTGGGA 1-TGCCTTGGGA 8.577033 -23.732495 0 T:23.0(11.33%),B:933.1(2.02%),P:1e-10 Tpos:253.5,Tstd:160.0,Bpos:247.2,Bstd:146.2,StrandBias:-0.2,Multiplicity:1.04
0.001 0.001 0.001 0.997
0.078 0.001 0.920 0.001
0.055 0.808 0.046 0.091
0.001 0.906 0.001 0.092
0.063 0.001 0.001 0.935
0.001 0.001 0.071 0.927
0.082 0.001 0.916 0.001
0.082 0.001 0.841 0.076
0.001 0.061 0.937 0.001
0.856 0.036 0.107 0.001
step4: 运行命令
findMotifs.pl \
EntrezID.txt \
human \
FindMotifFindResults/ \
-find motif1 \
-start -400 -end 100 -p 4 > FindMotifFindResults.txt
注:如果不是使用基因 ID,然后指定在其启动子区域找基序的位置,可以直接指定 BED 区域文件。
findMotifs.pl \
file.bed \
human \
FindMotifFindResults/ \
-find motif1 \
-p 4 > FindMotifFindResults.txt
输出结果:
$ head FindMotifFindResults.txt
GeneID PromoterID Offset Sequence Motif Name Strand MotifScore Unigene Refseq Ensembl Name Alias Orf Chr Description Type
6790 NM_003600 7 TCACAAGGCA 1-TGCCTTGGGA - 10.436320 NM_003600 ENSG00000087586 AURKA AIK|ARK1|AURA|BTAK|PPP1R47|STK15|STK6|STK7 - 20q13.2 aurora kinase A protein-coding
1556 NR_001278 92 TCCCATGGCA 1-TGCCTTGGGA - 10.142767 NR_001278 ENSG00000291083 CYP2B7P CYP2B|CYP2B7|CYP2B7P1 - 19q13.2 cytochrome P450 family 2 subfamily B member 7, pseudogene pseudo
1311 NM_000095 -41 TGCCTTGGGG 1-TGCCTTGGGA + 10.760737 NM_000095 ENSG00000105664 COMP CTS2|EDM1|EPD1|MED|PSACH|THBS5|TSP-5|TSP5 - 19p13.11 cartilage oligomeric matrix protein protein-coding
2922 NM_002091 -323 TCTCAAGGCA 1-TGCCTTGGGA - 10.512306 NM_002091 ENSG00000134443 GRP BN|GRP-10|preproGRP|proGRP - 18q21.32 gastrin releasing peptide protein-coding
3868 NM_005557 -269 TGCCTGGGGA 1-TGCCTTGGGA + 10.270905 NM_005557 ENSG00000186832 KRT16 CK16|FNEPPK|K16|K1CP|KRT16A|NEPPK|PC1 - 17q21.2 keratin 16 protein-coding
3868 NM_005557 -351 CCCCAAGACA 1-TGCCTTGGGA - 8.577034 NM_005557 ENSG00000186832 KRT16 CK16|FNEPPK|K16|K1CP|KRT16A|NEPPK|PC1 - 17q21.2 keratin 16 protein-coding
9055 NM_003981 -144 TGGCTTGGGA 1-TGCCTTGGGA + 9.974258 NM_003981 ENSG00000198901 PRC1 ASE1|MAP65 - 15q26.1 protein regulator of cytokinesis 1 protein-coding
5346 NM_002666 -94 TGCCTGGGGA 1-TGCCTTGGGA + 10.270905 NM_002666 ENSG00000166819 PLIN1 FPLD4|PERI|PLIN - 15q26.1 perilipin 1 protein-coding
9493 NM_004856 -287 TGACTTGGGA 1-TGCCTTGGGA + 10.152950 NM_004856 ENSG00000137807 KIF23 CDA3|CDAIII|CDAN3|CDAN3A|CHO1|KNSL5|MKLP-1|MKLP1 - 15q23 kinesin family member 23 protein-coding
表头 | 定义 | 示例 | 备注 |
|---|---|---|---|
GeneID | 基因的官方符号或名称(如 HGNC 或 NCBI Gene Symbol)。 | 6790(对应 AURKA) | 若输入是坐标文件,此列为最近邻基因名。 |
PromoterID | 关联的转录本或启动子区域标识符(通常为 RefSeq 转录本 ID)。 | NM_003600(AURKA 的转录本) | HOMER 默认使用 RefSeq 转录本定义启动子区域(TSS ±300bp)。 |
Offset | Motif 中心相对于 转录起始位点(TSS) 的位置。 | -41(上游 41bp)、+92(下游 92bp) | 负值表示 TSS 上游,正值表示下游。 |
Sequence | 匹配到 motif 的 实际 DNA 序列。 | TGCCTTGGGG | 序列方向与参考基因组一致(由 Strand 列决定正负链)。 |
Motif Name | 匹配的 motif 名称或标识符(来自输入 -find 的 motif 数据库或自定义文件)。 | 1-TGCCTTGGGA | 1- 可能表示 motif 编号,TGCCTTGGGA 是 motif 的核心序列。 |
Strand | Motif 在基因组上的方向。 | +(正链)、-(负链) | 负链上的序列是参考基因组的反向互补。 |
MotifScore | Motif 匹配的得分(分值越高,匹配越显著)。 | 10.436320 | 一般 >8 表示高置信度匹配(基于对数似然比)。 |
Unigene | UniGene 数据库的基因聚类标识符。 | 空白 | 现代分析中此列通常为空(UniGene 已逐步被淘汰)。 |
Refseq | RefSeq 转录本 ID。 | NM_003600 | 与 PromoterID 列重复。 |
Ensembl | Ensembl 基因 ID。 | ENSG00000087586 | |
Name | 基因的官方名称。 | AURKA(极光激酶 A) | |
Alias | 基因的其他别名(用 ` | ` 分隔)。 | `AIK |
Orf | 开放阅读框(Open Reading Frame)标识符。 | - | 通常为空。 |
Chr | 基因所在的染色体位置。 | 20q13.2 | |
Description | 基因的功能描述。 | aurora kinase A | |
Type | 基因的类型。 | protein-coding(编码蛋白)、pseudo(假基因) | 需注意假基因(如 CYP2B7P)可能无功能。 |
方法2: annotatePeaks.pl子程序
tss 模式下使用“ -m <motif file> ”选项
- 主要用途:该命令用于 注释启动子区,并检测其中是否包含特定 motif(如转录因子结合位点),最终输出包含 motif 实例信息的文件。
命令格式:
annotatePeaks.pl tss <基因组> -size -500,250 -hist <目标基因ID> -m <motif文件> > out.txt
必要步骤:
step1: 准备想要查看的基因ID
如果不输入关注基因ID,会在整个基因组上查找。
step2: 准备 motif 文件
step3: 运行命令
annotatePeaks.pl \
tss hg38 \
-size -500,250 \
-hist EntrezID.txt \
-m motif1 \
-p 4 > annotatePeaks.txt
输出结果:
输出文件将包含以下列:
- 峰/区域 ID
- 染色体
- TSS 区域开始
- TSS 区域结束
- 山峰海岸
6-18:注释信息
- CpG%
- GC%
- 基序实例
...
基序实例具有以下格式: <与 TSS 的距离>(<序列>,<链>,<保守性>) ,即 -29(TAAATCAACA,+,0.00)
场景3: 直接分析基因组上的特定区域(如ChIP-seq峰、ATAC-seq峰等)
方法1:findMotifsGenome.pl 子程序
命令格式:
findMotifsGenome.pl <peak/BED 文件> <基因组> <输出目录> -size # [选项]
必要步骤:
Step1: 选择关注 peaks 峰 的BED 文件
Step2: 选择基因组
Step3: 选择输出文件夹
Step4: 选择分析每个 peak 中心左右多少长度的序列 (size)
举例:
findMotifsGenome.pl ERpeaks.txt hg18 ER_MotifOutput/ -size 200 -mask
输出结果:
- homerMotifs.motifs<#>:这些是从头基序发现的输出文件,按基序长度分开,代表算法的单独运行。
- homerMotifs.all.motifs:简单地说,这是由所有homerMotifs.motifs<#>文件组成的合并文件。
- motifFindingParameters.txt:用于执行findMotifsGenome.pl的命令。
- knownResults.txt:包含已知基序富集统计信息的文本文件(可以在EXCEL中打开)。
- seq.autonorm.tsv:用于低阶寡核苷酸归一化的自动归一化统计信息。
- homerResults.html:从头基序发现的格式化输出。
- homerResults/目录:包含用于homerResults.html网页的文件,包括用于查找每个基序特定实例的motif<#>.motif文件。
- knownResults.html:已知基序发现的格式化输出。
- knownResults/目录:包含用于knownResults.html网页的文件,包括用于查找每个基序特定实例的known<#>.motif文件。
场景4: 在 RNA 区域找 Motif
HOMER最初并不是专门为RNA设计的,但它可以成功用于分析RNA基序数据。我们所说的RNA基序是指RNA序列中的短序列元素,类似于DNA基序,而不是像发夹结构这样的结构元素。例如,HOMER可以成功地确定共同调控mRNA集合中的miRNA种子,或者CLIP-Seq数据中的RNA结合元素。
可以在findMotifs.pl和findMotifsGenome.pl中使用“-rna”选项,这将导致仅进行正链的基序搜索,并使用“U”而不是“T”进行基序显示/匹配。HOMER目前还没有包含已知“RNA基序”的列表,因此不会进行“已知基序”分析。如果使用FASTA文件,请在输入文件中使用“T”(正常的DNA编码)。
方法一:提供基因 ID
举例:
findMotifs.pl mir1-downregulated.genes.txt human-mRNA MotifOutput/ -rna -len 8
方法二: 提供位置信息
举例:
findMotifsGenome.pl fox2.clip.bed hg38 MotifOutput -rna

