Nat. Commun. | 准确理解RNA翻译,RiboTIE直接处理核糖体信号
Nat. Commun. | 准确理解RNA翻译,RiboTIE直接处理核糖体信号

DrugOne
发布于 2025-04-18 16:54:10
发布于 2025-04-18 16:54:10
DRUGAI
今天为大家介绍的是来自美国密歇根大学的John R. Prensner与Jim Clauwaert团队以及比利时根特大学的Gerben Menschaert联合的一篇论文。RNA翻译是细胞生命活动中的一个基础过程,对人类疾病的发生和发展有着深远影响。然而,由于这个过程极其复杂,加上现有技术的限制,准确研究RNA翻译的变化一直是科学界面临的重大挑战。为了解决这个问题,研究团队开发了一个名为RiboTIE的新工具。这个工具基于人工智能中的Transformer模型,专门用来分析核糖体测序(RiboSeq)数据。与其他现有方法相比,RiboTIE的独特之处在于它能直接处理原始的核糖体信号数据,准确识别出基因组中可能被翻译的DNA片段(ORFs)。通过在多个不同数据集上的测试,这个工具展现出了极高的准确性和灵敏度。研究发现,RiboTIE不仅能够验证已知的科学发现,还能在正常脑组织和脑癌(髓母细胞瘤)样本中发现RNA翻译调控的新特征。这些结果表明,RiboTIE是一个功能强大的分析工具,能够显著提高核糖体测序数据分析的准确度和深度,有助于我们更好地理解蛋白质的合成过程及其在疾病中的作用。

RNA翻译是一个复杂的生物过程。在这个过程中,核糖体的两个组成部分(40S和60S亚基)会依次与RNA结合,同时还需要多种起始因子和辅助因子的参与,最终形成能够进行蛋白质合成的活性核糖体。这个过程直接决定了细胞内蛋白质的数量,在包括癌症在内的多种疾病中扮演着关键角色。在疾病状态下,多种遗传和非遗传因素都可能改变核糖体的组成、翻译效率和准确性。为了全面了解核糖体的活动,科研人员开发了核糖体测序(Ribo-Seq)技术。这项技术可以测定信使RNA的翻译效率,还能发现传统分析方法难以识别的非常规基因片段(ORFs)和新型蛋白质形式。因此,Ribo-Seq数据分析已成为多个研究领域的重要工具,包括新基因发现、RNA调控研究、蛋白质组学、微型蛋白质研究,以及针对RNA翻译的药物开发研究。
然而,Ribo-Seq数据分析面临着多重挑战。比如,在检测小型基因片段时缺乏统计可靠性,以及核糖体读数模式与实际翻译情况之间存在差异。这些差异可能来自生物因素(如组织类型的不同)或技术因素(如实验方法的差异)。现有的分析工具(如ORFquant、RpBp等)往往依赖人工筛选的特征来判断基因片段是否被翻译,这导致不同工具之间的结果差异较大,且常出现错误预测。
模型框架
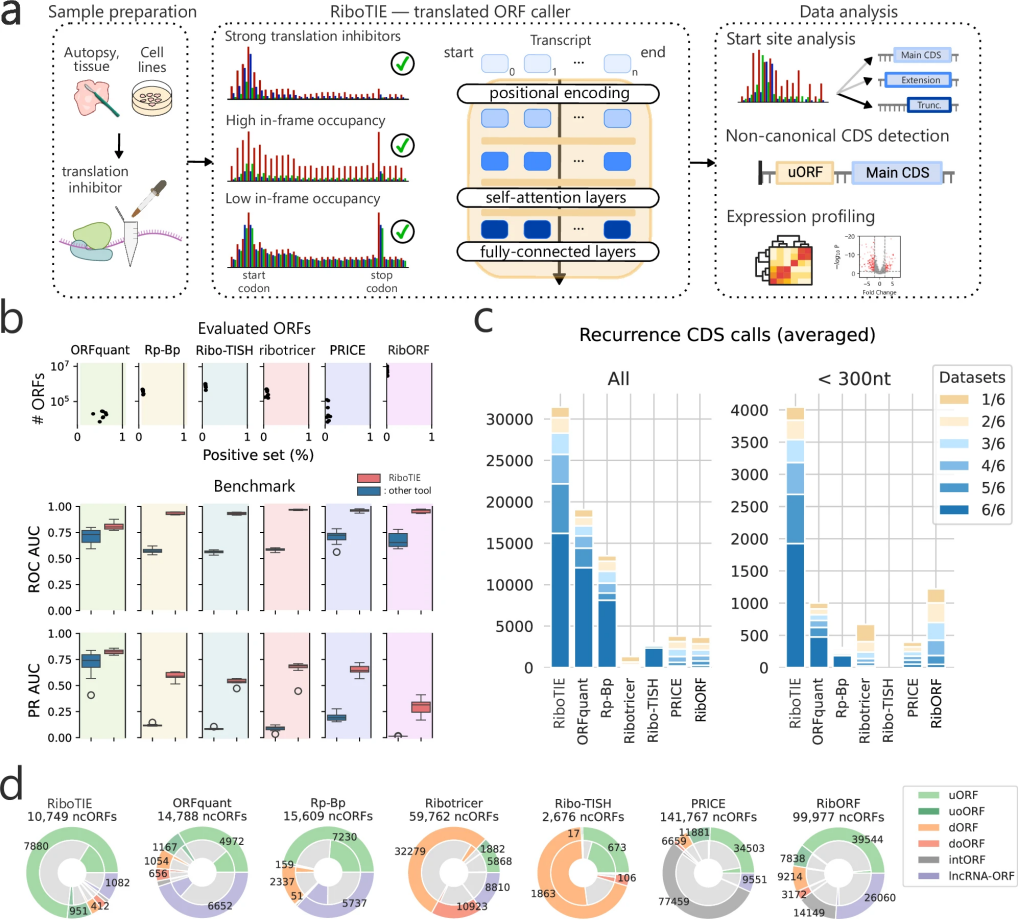
图 1
针对这些问题,研究团队开发了RiboTIE工具(如图1a所示)。这个工具采用了最新的机器学习技术,专门用于处理大规模生物数据。它可以利用预先训练好的模型,在识别小型翻译基因片段时表现出色。更重要的是,RiboTIE能够根据每个数据集的特点,准确识别核糖体保护的RNA片段与实际翻译基因之间的关系,这使得它能在不同类型的数据中都保持稳定的表现。在对脑癌(髓母细胞瘤)患者的样本和细胞系的分析中,RiboTIE不仅验证了已知的研究发现,还展示了其在不同样本质量条件下的稳定性。因此,RiboTIE为生物学家和生物信息学家提供了一个强大的工具,有助于深入了解人类细胞功能和疾病状态。
RiboTIE工具具有四个突出特点:首先,它简化了数据预处理步骤,直接处理所有位置和长度的读数,提高了整体性能。其次,它采用创新的方法,先预测后构建基因片段,这使得工具能够评估转录组中所有可能的翻译起始位点。第三,工具只处理核糖体保护片段的计数数据,避免了其他因素带来的偏差。第四,采用了先进的机器学习技术(Transformer网络),能够自动提取特征并处理不同长度的输入数据。
实验结果
研究团队在八个不同实验数据集上评估了RiboTIE的性能。如图1b所示,与其他工具相比,RiboTIE在准确性和灵敏度方面都表现出色。在胰腺前体细胞的测试中(图1c),RiboTIE识别出的编码序列(CDS)数量比第二名多出64.9%,在识别小于300bp的编码序列时,其效果更是其他工具的3倍。图1d显示,RiboTIE在识别非常规基因片段方面也具有独特优势。
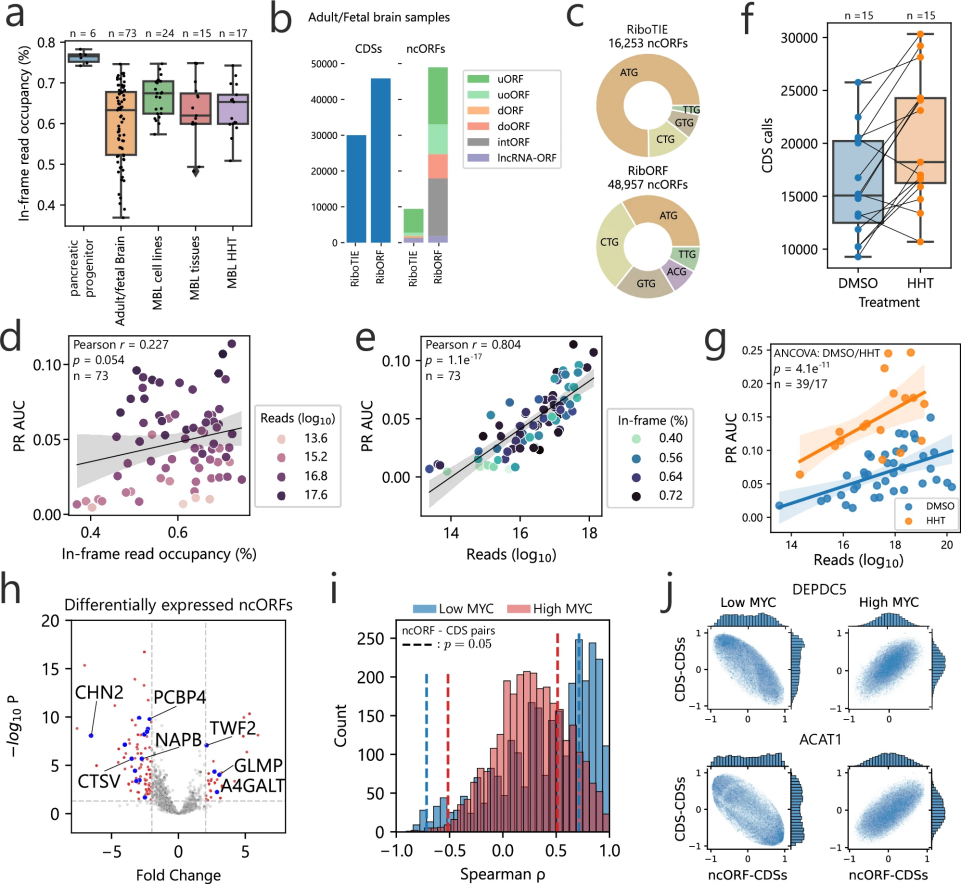
图 2
为了验证工具在实际医学研究中的应用价值,研究人员分析了73个人类脑组织样本(包括30个胎儿样本和43个成年样本)以及15个髓母细胞瘤患者的组织样本(图2a)。即使在部分样本质量不佳的情况下,RiboTIE仍然表现稳定。如图2b-e所示,该工具在识别编码序列和非编码ORFs方面都优于现有方法。
在髓母细胞瘤研究中(图2h-j),RiboTIE帮助研究人员发现了201个与疾病相关的非编码ORFs。这些发现与已知的髓母细胞瘤生物学特征相符,特别是在神经系统发育和分化相关基因中的表现。通过与另一个工具(TIS Transformer)的结果对比,研究人员确定了22个高置信度的候选非编码ORFs,这些基因片段的翻译水平能够准确区分不同类型的髓母细胞瘤。这些发现为理解疾病机制和开发潜在治疗方法提供了新的视角。
为了进一步验证RiboTIE的预测准确性,研究团队对三对高MYC表达和低MYC表达的髓母细胞瘤细胞系进行了深度蛋白质组学分析。通过严格的数据筛选标准,研究人员发现了44个能够证实非编码ORFs或蛋白质新形式存在的肽段,这个数量与其他工具(如ORFquant)的预测结果相当。
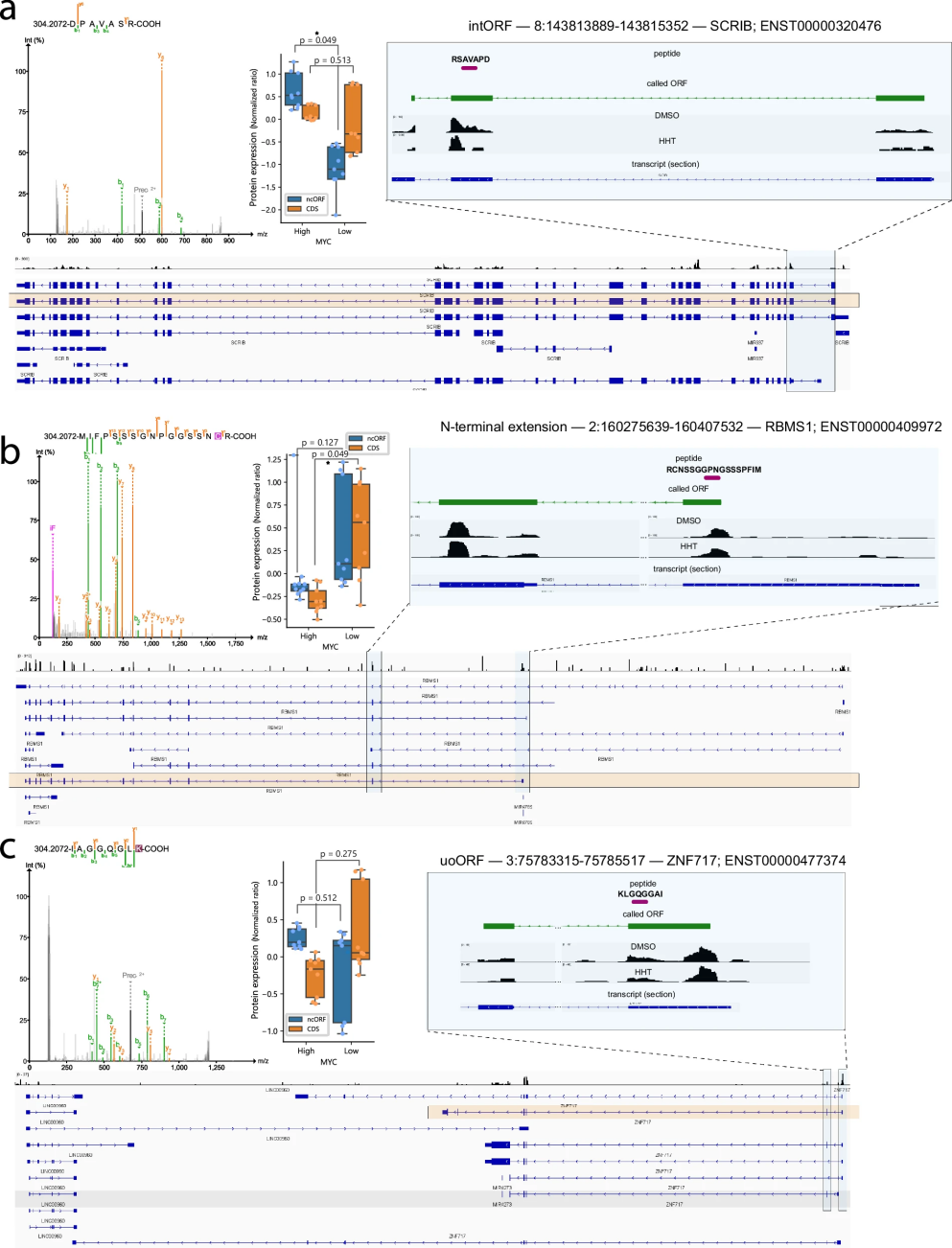
图 3
特别值得注意的是,研究发现了几个在高MYC和低MYC表达细胞中表现不同的新型蛋白质(如图3所示):
- 在SCRIB基因中发现了一个特殊的内嵌型基因片段,虽然SCRIB基因本身的表达没有明显变化,但这个新发现的片段在不同类型的髓母细胞瘤中表现不同(图3a)。
- 在RBMS1基因中,研究者发现了一个延长的蛋白质形式。这个新形式与原有的基因产物一样,在高MYC表达的样本中都显示出降低的表达水平(图3b)。
- 在ZNF717基因中,发现了一个位于基因上游的新片段。有趣的是,虽然ZNF717基因本身在不同类型的髓母细胞瘤中表达存在差异,但这个新发现的片段的表达却保持稳定(图3c)。
这些发现表明,非编码基因片段在疾病中可能发挥着独特的作用,它们的表现模式可能与传统认知的基因产物完全不同。这为理解髓母细胞瘤的发病机制提供了新的视角,也为未来的治疗策略开发提供了潜在的新靶点。
讨论
RiboTIE是一款创新的人工智能工具,专门用于分析细胞中的蛋白质合成过程。它不仅能准确识别基因组中的功能片段,还能在数据质量不理想的情况下保持稳定表现。研究人员已在166个不同类型的数据集上验证了它的可靠性,特别是在研究儿童脑癌等疾病时,这个工具帮助发现了许多新的生物学特征,为疾病研究开辟了新途径。
编译|于洲
审稿|王梓旭
参考资料
Clauwaert J, McVey Z, Gupta R, et al. Deep learning to decode sites of RNA translation in normal and cancerous tissues[J]. Nature Communications, 2025, 16(1): 1275.
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-04-17,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

