高分杂志中的Cox回归模型构建以及筛选生物标志物分析(Q1/IF: 58.7)
高分杂志中的Cox回归模型构建以及筛选生物标志物分析(Q1/IF: 58.7)
生信技能树
发布于 2025-10-11 13:54:53
发布于 2025-10-11 13:54:53
前面我们给大家分享了一个将文章分析的数据和代码整理为在线书籍形式的文献,见:分享一篇将代码整理成在线book形式的顶刊文献(Q1/IF: 58.7),今天学习其中的Figure4,使用Cox回归模型挑选生物标志物并绘制森林图。(「这个系列到这里就结束啦,我挑选出来学习的部分个人觉得是比较有代表性的,重复性的图就没有展示出来,以及对作者提供的代码进行了一些修改」(主要是给出来的代码跟文献里面出来的图的结果不一样)。文献中还有一些其他的小细节大家可以再看看,挑选标志物的技巧用到自己的文章中去。)
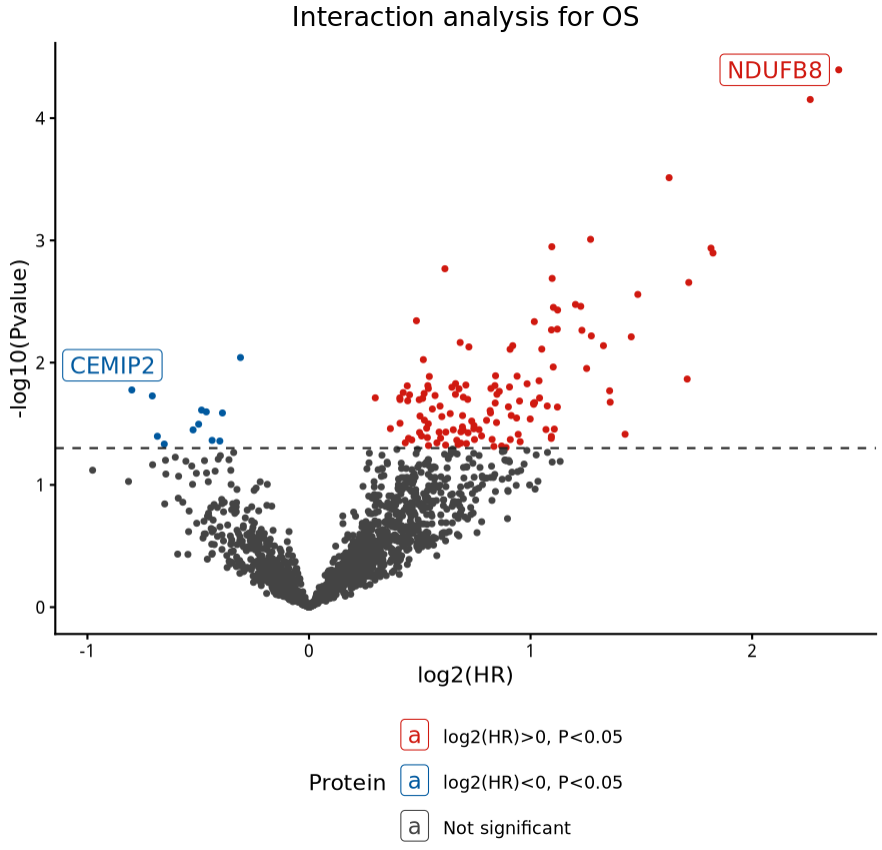
图四:鉴定两种用于预测PDAC患者辅助化疗结果的蛋白质biomarkers
a,散点图显示化疗与生物标志物交互项的log2(HR)与- log10(交互项P值)的关系。蓝色和红色点分别代表PDAC辅助化疗的有利和不利蛋白质组学生物标志物(交互项P < 0.05)。排名前两位的蛋白质用黑色圆圈标记。
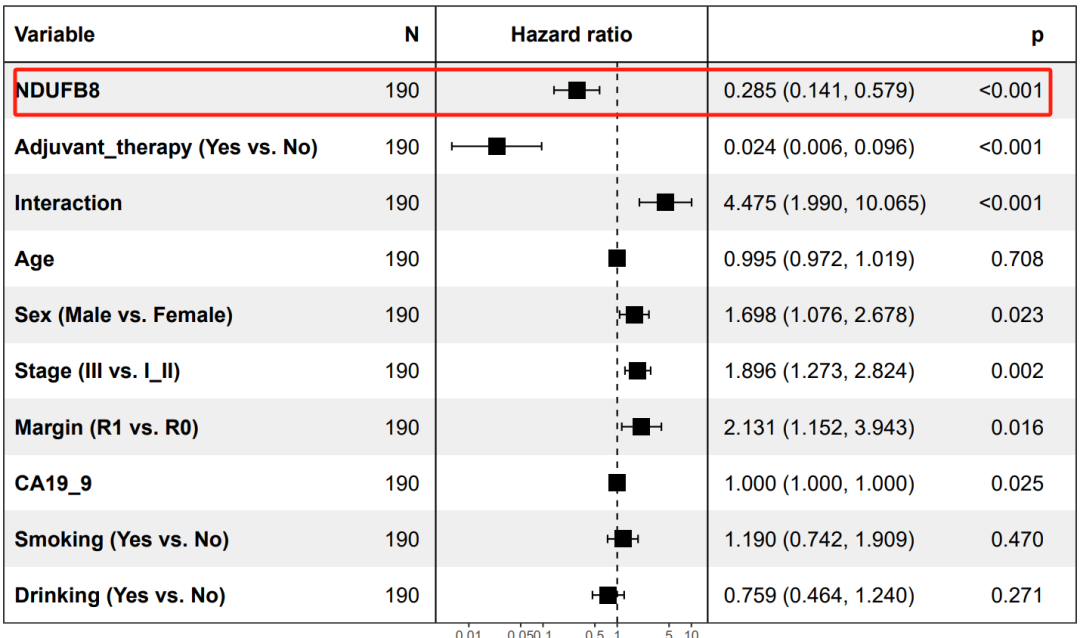
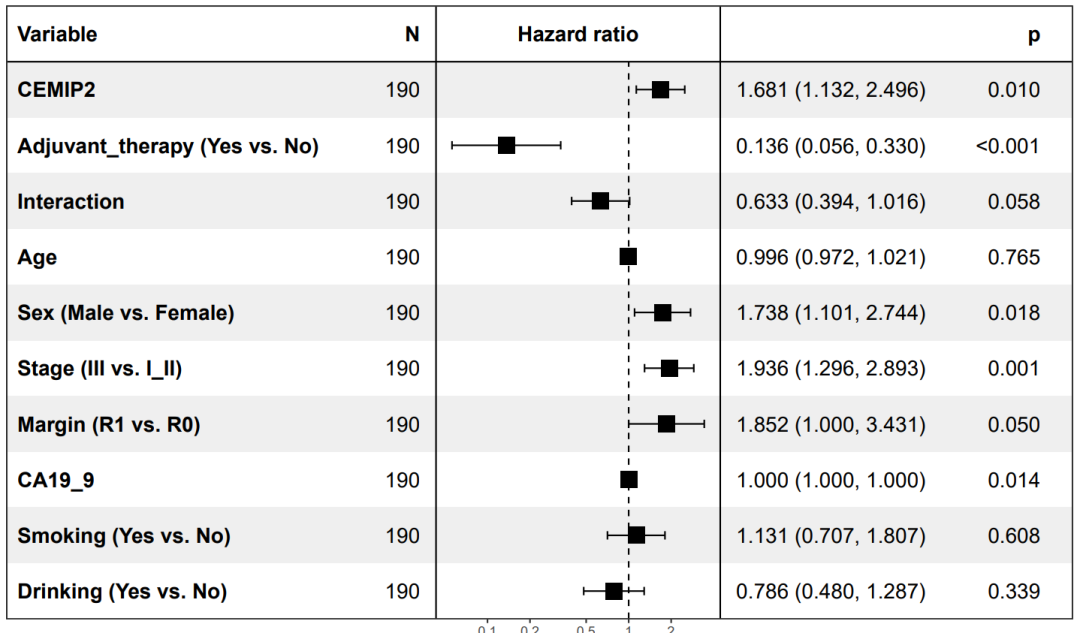
b,分别为NDUFB8和CEMIP2的多变量Cox比例风险回归的森林图。
c,d,Kaplan-Meier生存曲线比较接受辅助化疗和根据 NDUFB8(c)和 CEMIP2(d)的高/低丰度(中位数截断)分层的患者亚组之间的总生存期(OS)。
「代码和数据」
数据和代码分享:http://www.genetictargets.com/PDAC2BOOKLET/index.html。「好多人说找不到数据,这个链接点击进去」:

化疗biomarkers鉴定
化疗与生物标志物交互项的对数危险比(log2)与负对数P值(-log10)的散点图,该交互项通过Cox比例风险模型计算得出,以总生存期(OS)为依据。蓝色和红色的点分别代表对胰腺导管腺癌(PDAC)辅助化疗有利和不利的蛋白质生物标志物(交互P值 < 0.05)。排名前两位的蛋白质用黑色圆圈标记。
Step 1: 加载相关数据
分别加载前面的差异分析结果deg,蛋白表达矩阵pro,以及样本临床信息带有生存时间数据cli:
rm(list=ls())
library(openxlsx)
library(tidyverse)
library(stringr)
library(survival)
library(survminer)
library(pbapply)
library(ggthemes)
dir.create("./results/Figure4")
# Step 1: Load the Data
# 差异蛋白以及差异模块信息
deg <- readRDS("./PDAC2_data_results/data/proteomics/wgcna/2D-node.data.rds") # DEG & module genes
head(deg)
table(deg$Module)
table(deg$phosSig)
# down up zz
# 594 314 1151
# 表达矩阵
pro <- readRDS("./PDAC2_data_results/data/proteomics/20230412_PDAC_PRO_exp.rds")
dim(pro)
pro[1:5,1:5]
# 样本临床信息
cli <- readRDS("./PDAC2_data_results/data/clinical/20230412_PDAC_191_patients_clinical.rds")
dim(cli)
cli[1:10,1:10]
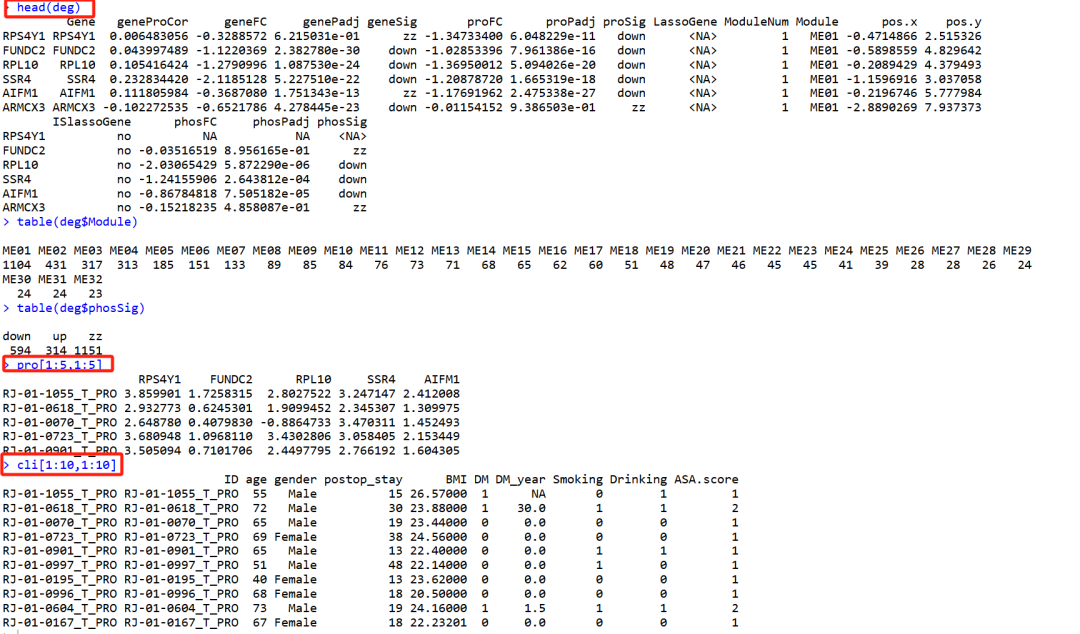
Step 2: 准备Coxph分析数据
提取差异蛋白以及相关的临床信息:
# Step 2: Coxph data
# clinical data + DEG genes' expression data
# 得到显著差异蛋白
deg <- deg %>% filter(proSig !="zz") %>% pull(Gene) # 1860 DEG genes
head(deg)
length(deg)
# 表达矩阵和临床信息取交集
rownames(cli) <- cli$ID
id.inter <- intersect(rownames(cli),colnames(pro)) # 191 patients
pro <- pro[deg,id.inter] %>% t()
identical(rownames(cli),rownames(pro))
# 差异蛋白与临床信息合并在一起
biomarker_data <- cbind(cli %>% dplyr::select(OS_Time,OS_Status,Censoring_chemo),pro)
biomarker_data[1:10,1:10]
# category attribute: chemotherapy treatment
biomarker_data <- biomarker_data %>% mutate(chem=factor(Censoring_chemo,levels = c("0","1")))
table(biomarker_data$chem)
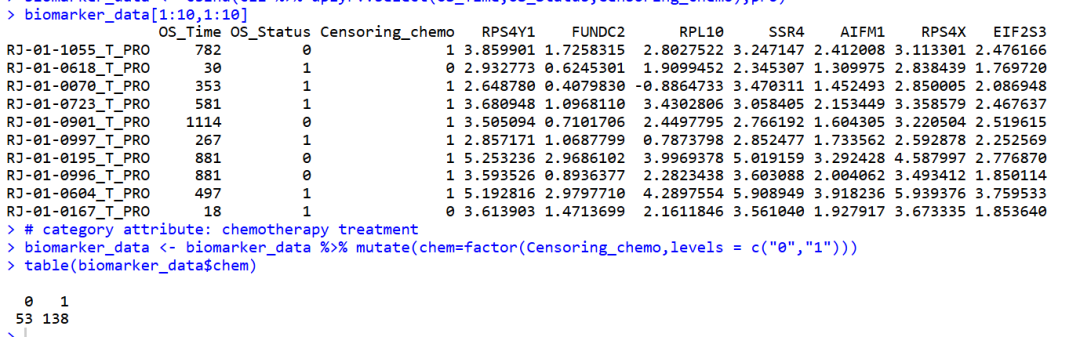
Step 3: Coxph 分析
使用survival包中的coxph函数来拟合一个Cox比例风险模型,用于分析基因表达与化疗效果之间的交互作用对患者总生存期(OS)的影响。
Gene * chem:表示基因表达与化疗状态的交互作用。这个交互项用于评估基因表达在不同化疗状态下的效应是否不同。具体来说,它会拟合以下模型:
- 主效应:
Gene和chem的单独效应。 - 交互效应:
Gene和chem的交互效应。
# Step 3: Coxph
# Coxph : OS ~ chemotherapy*gene
# 所有的基因做cox回归分析
ls_re <- pbapply::pblapply(colnames(pro),FUN=function(g){
data <- biomarker_data %>%
select(OS_Time,OS_Status,chem) %>%
mutate(Gene=biomarker_data%>%pull(g))
fit <- coxph( Surv(OS_Time,OS_Status) ~ Gene*chem ,data=data) ## interaction term
broom::tidy(fit) %>% mutate(HR=exp(estimate)) %>% select(p.value,HR) %>% t() %>% as.vector()
})
# result
res <- do.call(rbind,ls_re)
colnames(res) <- c("biomarker_p","biomarker_HR","chemotherapy_p",
"chemotherapy_HR","interaction_p","interaction_HR")
## predictive biomarker
res <- res %>% as_tibble() %>% mutate(Gene=colnames(pro)) %>%
arrange(interaction_p) %>%
dplyr::select(Gene,everything())
# cutoff
res$group <- "Not significant"
res[which(res$interaction_HR>1 & res$interaction_p <0.05),"group"] = "log2(HR)>0, P<0.05"
res[which(res$interaction_HR<1 & res$interaction_p <0.05),"group"] = "log2(HR)<0, P<0.05"
table(res$group)
head(res)
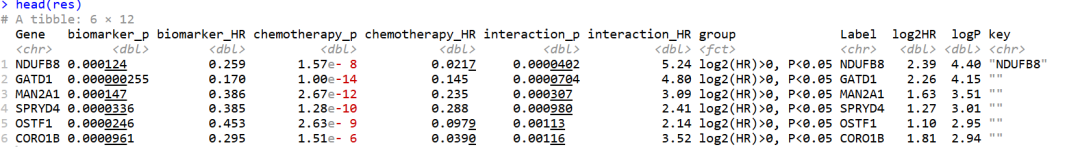
Step 4: 结果可视化
这里的代码作者出来的效果跟文献中的也不一样,我进行了修改。
# Step 4: Visualization
color.tri <- c("#D01910","#00599F","#444444") # up ,down ,not
## volcano
res <- res %>% mutate(group=factor(group,levels = c("log2(HR)>0, P<0.05","log2(HR)<0, P<0.05","Not significant")),
Label=Gene,
Label=replace(Label,is.na(group),""))
head(res)
table(res$group)
res <- res %>% mutate(log2HR=log2(interaction_HR)) %>%
mutate(logP= -log10(interaction_p))
head(res)
res$key <- ""
res$key[match(c("CEMIP2","NDUFB8"),res$Gene)] <- c("CEMIP2","NDUFB8")
p <- ggscatter(res,
x = "log2HR", y = "logP",
color = "group", size = 1,
main = paste0("Interaction analysis for OS"),
xlab = "log2(HR)", ylab = "-log10(Pvalue)",
palette = color.tri,
label = res$key,font.label = 12, label.rectangle = T, repel = T)+
theme_classic() +
geom_hline(yintercept = -log10(0.05), linetype="dashed", color = "#222222") +
labs(color = "Protein") + # 修改颜色图例的标题
guides(color = guide_legend( nrow = 3)) +
theme(plot.background = element_blank(),
plot.title = element_text(hjust = 0.5),
legend.position = "bottom"
)
p
ggsave("./results/Figure4/4A-Predictive-biomarker.pdf", p, width = 5, height = 5.2)
write.xlsx(as.data.frame(res),"./results/Figure4/4A-Predictive-biomarker.xlsx",colNames=T,rowNames=F,overwrite = T)
蓝色和红色的点分别代表对胰腺导管腺癌(PDAC)辅助化疗有利和不利的蛋白质生物标志物(交互P值 < 0.05)。排名前两位的蛋白质进行了标记。
多变量Cox回归的森林图
分别为 NDUFB8 和 CEMIP2 绘制多变量Cox比例风险回归森林图。
Step 1: 加载数据
使用的数据为独立队列 the RJ-cohort 1,数据在作者提供的
library(forestmodel)
library(RColorBrewer)
# Step 1: Load the Data
# Sample information and clinical characteristics of the 191 PDAC patients (the RJ-cohort 1)
rj1 <- read.xlsx("./PDAC2_data_results/data/Extended Data Table 2.xlsx", startRow = 2)
dim(rj1)
rj1[1:10,1:10]
# 处理一下相关临床信息为0,1数字化
rj1$`LassoLevel (High vs. Low)` <- ifelse(rj1$LassoLevel == 'High', 1, 0)
rj1$`Adjuvant_therapy (Yes vs. No)` <- ifelse(rj1$Censoring_chemo == 'Chem', 1, 0)
rj1$`Stage (III vs. I_II)` <- ifelse(rj1$Stage == 'III', 1, 0)
rj1$`NDUFB8 (High vs. Low)` <- ifelse(rj1$NDUFB8_l == 'High', 1, 0)
rj1$`CEMIP2 (High vs. Low)` <- ifelse(rj1$CEMIP2_l == 'High', 1, 0)
rj1$`Sex (Male vs. Female)` <- ifelse(rj1$Gender == 'Male', 1, 0)
rj1$`Margin (R1 vs. R0)` <- ifelse(rj1$Margin == 'R1', 1, 0)
rj1$`Smoking (Yes vs. No)` <- rj1$Smoking
rj1$`Drinking (Yes vs. No)` <- rj1$Drinking
rj1[1:10,1:15]
数据如下:
Step 2: plot for NDUFB8 * Adjuvant_therapy
绘制NDUFB8基因的多变量Cox比例风险回归的森林图:
# Step 2: plot for NDUFB8 * Adjuvant_therapy
# custom panels for column: Adjust the font style, thickness, and column width of variables in the graph
forest.panels <- list(
list(width = 0.01),
list(width = 0.1, display = ~variable,
fontface = "bold", heading = "Variable"),
list(width = 0.1, display = ~level),
list(width = 0.05, display = ~n, hjust = 1, heading = "N"),
list(width = 0.03, item = "vline", hjust = 0.5),
list(width = 0.55, item = "forest", hjust = 0.5,
heading = "Hazard ratio", linetype = "dashed",line_x = 0),
list(width = 0.03, item = "vline", hjust = 0.5),
list(width = 0.12, display = ~ ifelse(reference, "Reference", sprintf("%0.3f (%0.3f, %0.3f)",
trans(estimate), trans(conf.low), trans(conf.high))), display_na = NA),
list(width = 0.03,
display = ~ ifelse(reference, "", format.pval(p.value, digits = 1, eps = 0.001) ),
display_na = NA, hjust = 1, heading = "p"),
list(width = 0.03))
Interaction <- rj1[["NDUFB8"]] * rj1[['Adjuvant_therapy (Yes vs. No)']]
pdf('./results/Figure4/Figure4b-1.pdf', width = 10, height = 6)
print(forest_model(coxph(as.formula(paste0('Surv(OS_month, OS_status) ~', '`', "NDUFB8", '`', '+ `Adjuvant_therapy (Yes vs. No)` + Interaction + Age + `Sex (Male vs. Female)` + `Stage (III vs. I_II)` + `Margin (R1 vs. R0)` + CA19_9 + `Smoking (Yes vs. No)` + `Drinking (Yes vs. No)`')), data = rj1),
panels = forest.panels, factor_separate_line = F))
dev.off()
Step 3: plot for CEMIP2 * Adjuvant_therapy
绘制 CEMIP2 基因的多变量Cox比例风险回归的森林图:
# Step 3: plot for CEMIP2 * Adjuvant_therapy
Interaction <- rj1[["CEMIP2"]] * rj1[['Adjuvant_therapy (Yes vs. No)']]
Interaction
pdf('./results/Figure4/Figure4b-2.pdf', width = 10, height = 6)
print(forest_model(coxph(as.formula(paste0('Surv(OS_month, OS_status) ~', '`', "CEMIP2", '`', '+ `Adjuvant_therapy (Yes vs. No)` + Interaction + Age + `Sex (Male vs. Female)` + `Stage (III vs. I_II)` + `Margin (R1 vs. R0)` + CA19_9 + `Smoking (Yes vs. No)` + `Drinking (Yes vs. No)`')), data = rj1),
panels = forest.panels, factor_separate_line = F))
dev.off()
今天分享到这~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-10,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

