Nature | 面向超大规模生物序列数据库的高效精准检索框架
Nature | 面向超大规模生物序列数据库的高效精准检索框架

DrugOne
发布于 2025-11-17 20:41:04
发布于 2025-11-17 20:41:04
DRUGONE
随着生物测序技术的快速发展,全球已积累超过拍字节级规模的基因序列数据。这些数据蕴含丰富的生物多样性、进化与病原体传播信息,但如何在如此庞大的序列库中快速、准确地搜索与比对,仍是生物信息学的一大挑战。
研究人员开发了一个名为 MetaGraph 的新型搜索框架,能够在拍字节级别的核酸序列数据库中实现高效比对。该系统基于压缩的可扩展图索引结构,并结合高效的字符串匹配算法,实现了对短读段(reads)与长片段(contigs)的全局搜索。MetaGraph不仅能够在海量数据库中快速定位相关序列,还能保持接近参考比对的精度。研究人员在多个公共基因组与宏基因组数据集上验证了该方法,包括人类基因组重测序、病毒追踪以及环境微生物分析任务。MetaGraph实现了比传统工具高数百倍的搜索速度,并在灵敏度与精度上保持一致甚至更优。

随着测序成本大幅下降,生物学进入了“数据爆炸”时代。到目前为止,公共序列数据库如ENA、SRA与NCBI已累计存储超过数十亿样本与数千万亿条reads,数据量突破拍字节级别。这些数据库为研究生命多样性、病原体追踪与个体基因变异提供了前所未有的资源,但同时带来了极大计算挑战。
传统的序列搜索算法(如BLAST、BWA或Bowtie)在单一参考基因组上表现良好,但在面对全球范围的异源数据集合时会失效,主要瓶颈包括:
- 内存与索引规模过大:传统哈希索引需占用上百TB内存;
- 搜索效率低下:无法实时处理海量样本比对;
- 缺乏灵活的多样本整合:现有系统难以同时覆盖宏基因组、病毒组与宿主序列。
研究人员指出,实现可在全球数据库中搜索的“基因组级搜索引擎”,必须在存储压缩、索引结构与搜索算法上实现突破。因此,他们提出了MetaGraph——一种面向大规模序列数据的高压缩、高通量搜索系统。
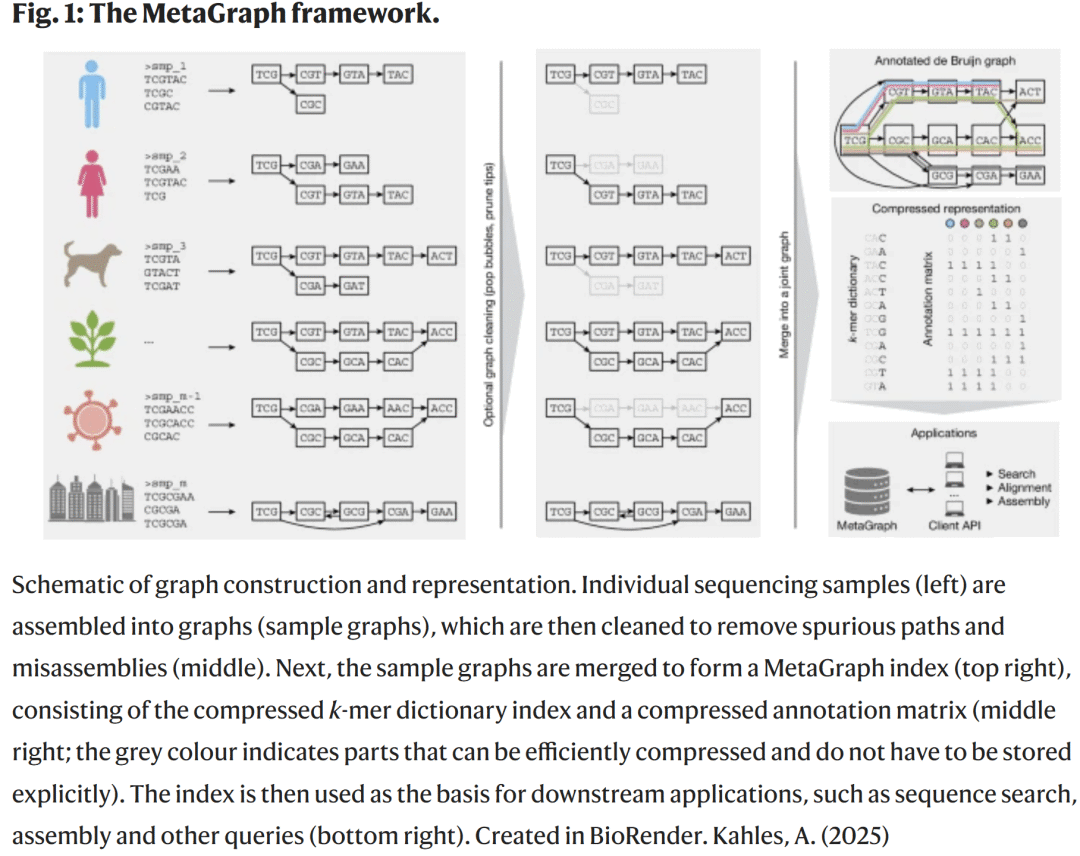
方法
序列图索引的构建
MetaGraph采用压缩的 de Bruijn图 表示大规模序列集合。每个节点代表一个固定长度的k-mer,边表示相邻序列关系。为了应对超大规模数据,研究人员使用分布式哈希表和压缩位向量存储图结构,从而在保持可搜索性的同时大幅降低存储需求。
此外,MetaGraph通过“分层索引策略”组织不同来源的数据(例如人类、病毒、环境样本),并利用增量更新机制实现快速扩展,无需重建整个索引。
查询与比对机制
搜索过程基于k-mer匹配原理。MetaGraph将输入序列分解为k-mer集合,通过快速查找定位到候选路径,然后进行局部比对。为提高精度,系统引入动态路径合并算法,可重建完整的序列上下文并消除噪声匹配。此外,研究人员开发了批量搜索接口,允许同时查询数十万条reads,并使用多线程并行化加速执行。
可扩展性与分布式计算
为实现拍字节级搜索,MetaGraph采用了基于云的分布式图分片(graph sharding)策略。每个节点独立维护局部索引,通过哈希分配查询任务并行处理。系统支持GPU加速与远程I/O调度,使得单次搜索可在数分钟内完成。
准确性与效率评估
研究人员在人工基准数据与真实宏基因组数据上对MetaGraph进行系统评估。比较对象包括传统比对工具(如BWA、MMseqs2)及其他压缩图索引系统(如BIGSI与COBS)。
性能指标涵盖:
- 搜索精度(查准率、查全率);
- 单查询与批量搜索耗时;
- 内存与存储占用;
- 样本间共享k-mer比例。
结果
MetaGraph实现拍字节级可扩展索引
MetaGraph成功构建了一个包含超过1 拍字节原始序列数据的索引系统。其核心索引结构仅占用约60 TB存储,比传统哈希方法压缩约20倍。
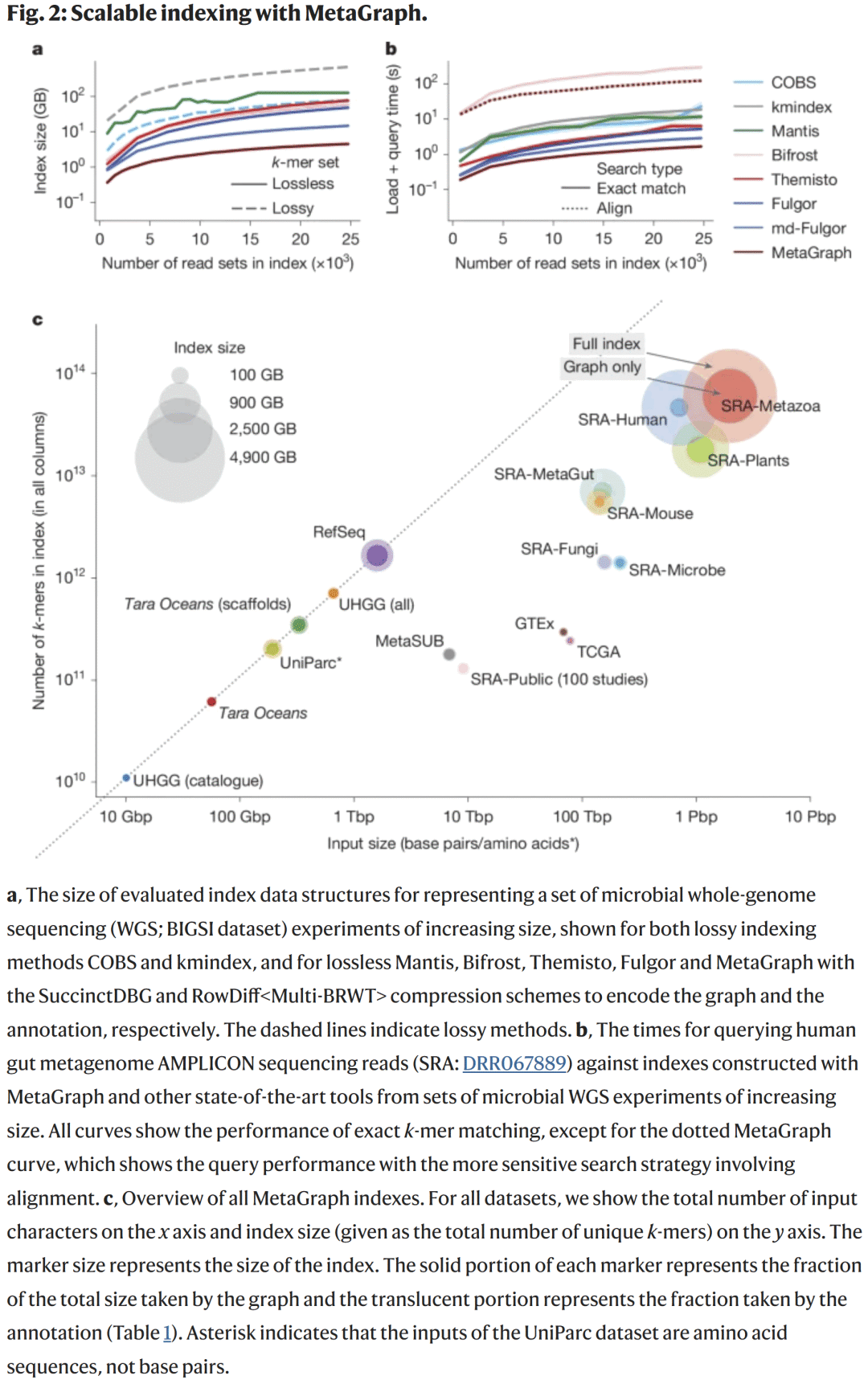
搜索速度提升数百倍
在100万个样本的查询任务中,MetaGraph平均每条read的查询时间低于0.1毫秒,相比BWA-MEM提高约800倍。

病原体追踪与宏基因组应用
MetaGraph被应用于多个公共健康项目中,包括COVID-19病毒变异追踪与细菌群落分析。
在病毒追踪任务中,系统能够在全球宏基因组样本中快速识别出与新突变株相关的序列痕迹,从而提前发现传播趋势。
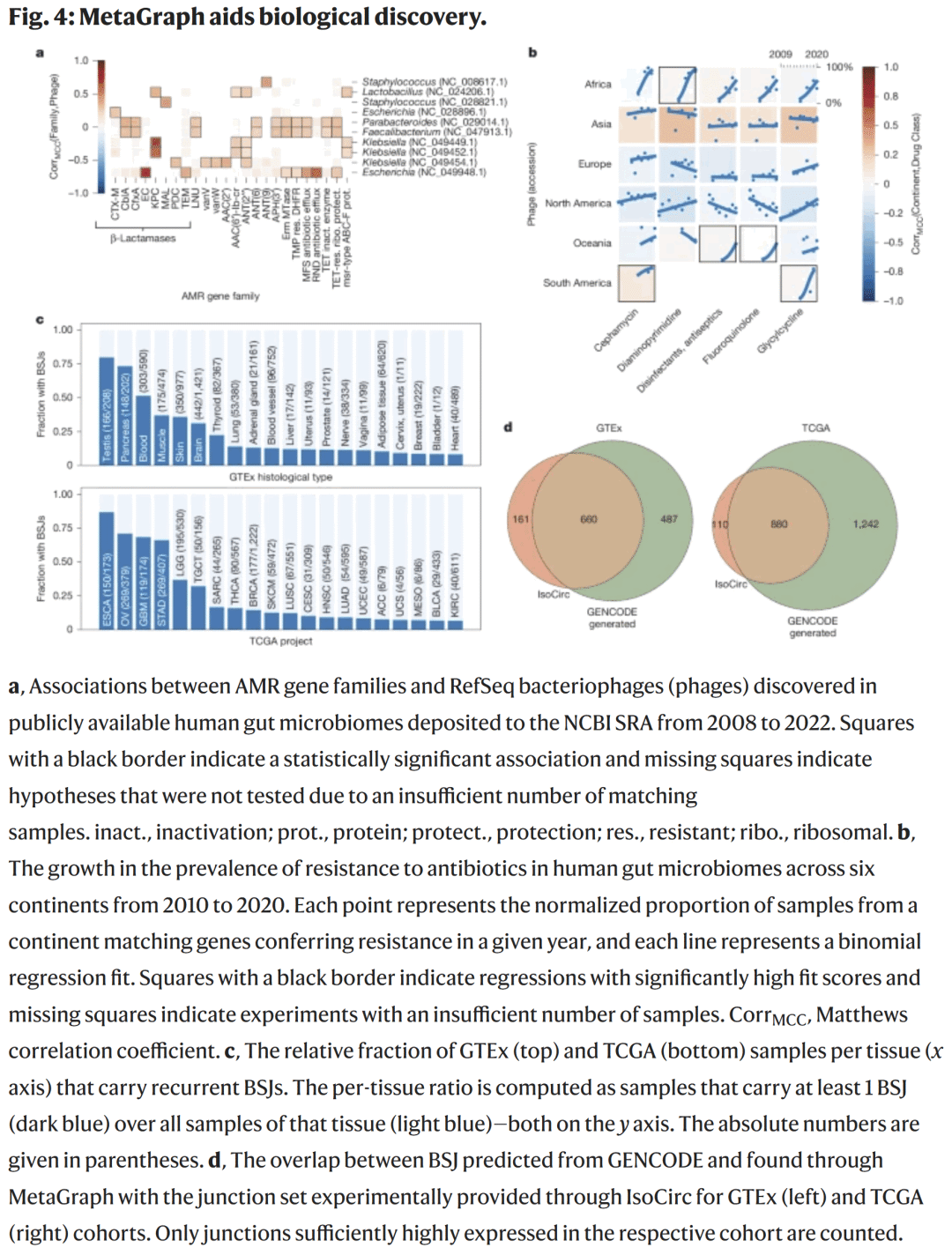
与参考比对方法的准确性对比
研究人员验证了MetaGraph的搜索精度与传统参考比对方法一致。对于人类基因组reads,MetaGraph的召回率超过99.5%,误检率低于0.2%,几乎达到BWA-MEM精度。
实时跨数据库搜索与动态更新
MetaGraph支持动态索引扩展,可实时整合新样本,无需全量重建。研究人员在实际运行中实现了每小时更新一次索引,并对新数据进行即时搜索。
讨论
研究人员提出的MetaGraph框架在大规模序列数据搜索领域实现了重大突破:
- 高压缩可扩展索引:首次实现拍字节级数据库的全局可搜索性;
- 高效并行搜索机制:在保证精度的同时实现百倍至千倍速度提升;
- 灵活的多源整合:可同时处理人类、病毒与环境样本数据。
然而,研究人员也指出当前系统仍存在两方面挑战:
- 对高重复性区域的索引可能引入存储冗余;
- 对非标准化测序数据的噪声鲁棒性需进一步优化。
未来方向包括:
- 引入深度学习驱动的序列相似性嵌入模型以提升搜索语义能力;
- 开发分层索引压缩算法以进一步降低存储需求;
- 构建跨物种基因组搜索引擎以支持生态学与进化研究。
研究人员认为,随着全球序列数据库的持续扩张,MetaGraph代表了下一代生物信息检索系统的核心方向,可推动从“数据堆积”到“信息挖掘”的重大转变。
整理 | DrugOne团队
参考资料
Karasikov, M., Mustafa, H., Danciu, D. et al. Efficient and accurate search in petabase-scale sequence repositories. Nature (2025).
https://doi.org/10.1038/s41586-025-09603-w
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-16,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

