OpenFold3发布!用于蛋白质、核酸和药物结构预测的开源基础模型
OpenFold3发布!用于蛋白质、核酸和药物结构预测的开源基础模型

DrugOne
发布于 2025-11-17 20:51:20
发布于 2025-11-17 20:51:20
OpenFold3终于发布了!在深入探讨这对蛋白质结构预测未来意味着什么之前,先看看此次发布当下会产生什么影响。OpenFold3 是一个完全开源的 AlphaFold3 替代品,由生物技术和制药公司组成的 OpenFold 联盟(Tamarind Bio 是其成员之一)支持开发。让我们来看看基准测试结果。

现状如何?
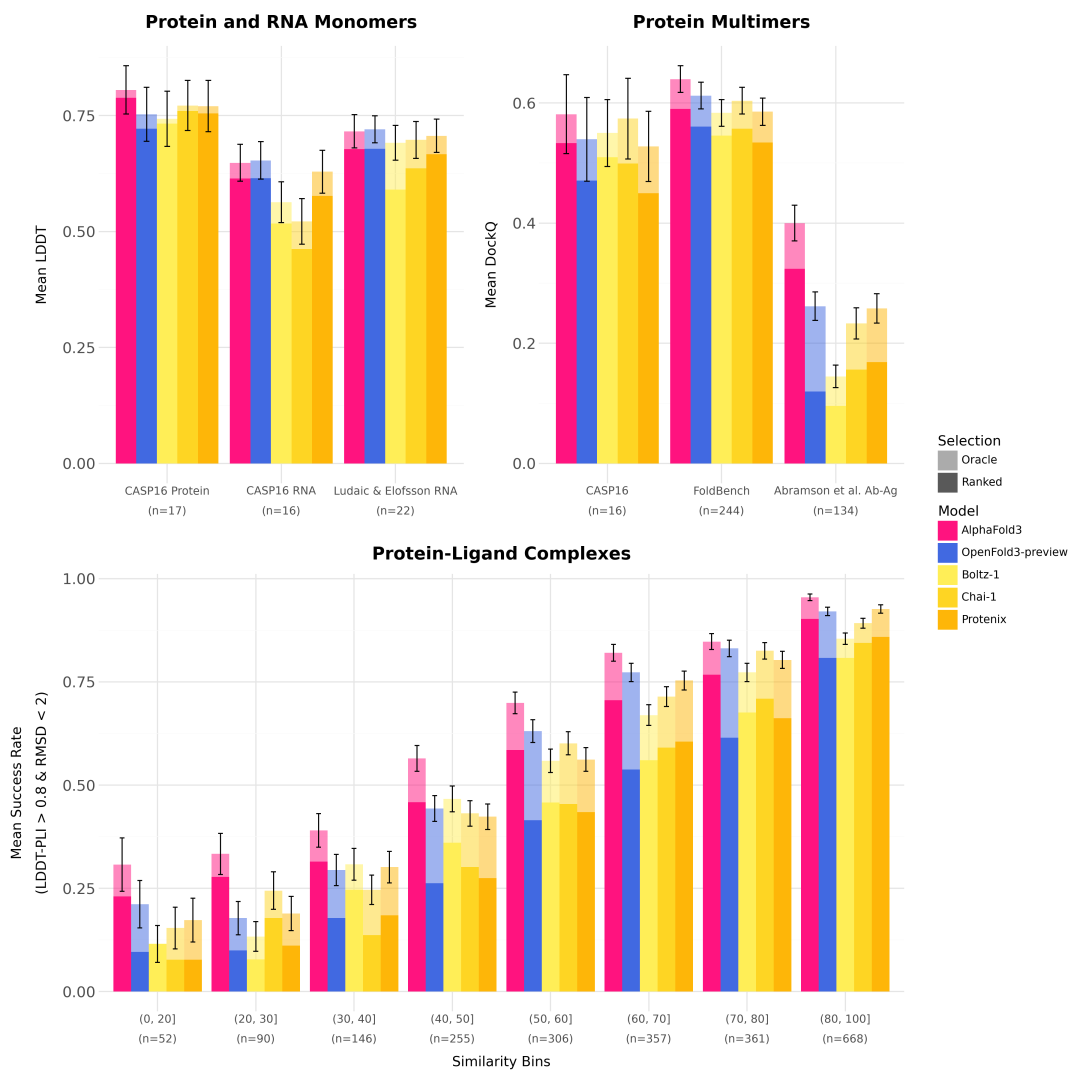
与其他可商用的 AF3 复现模型一样,OpenFold3 在某些模式/应用上确实落后,而在其他方面则达到或超越了 AlphaFold3。
小分子(对接/构象预测)
对于与训练数据稍显相似的配体,OF3 的表现大致与 AF3 相当;但在最具挑战性的新型配体类别(例如 GUNS & Poses)上落后于 AF3。 为了公平比较,训练数据截止日期与 AF3 一致;一些竞争对手使用了更新的数据,使得图表无法直接比较。
抗体-抗原
存在较大差距:根据作者的基准测试,在针对 Boltz、Chai 和 OpenFold3 的抗体-抗原对接测试中,AF2 仍然是表现最佳的模型,所有这些模型都大幅落后于 AF3。 相较于其他替代方案,AF3 得益于其更大量的采样,OpenFold3 也不例外。
单体与多聚体
对于单链蛋白质和非抗体复合物,所有 AlphaFold 风格的结构预测器在准确性上基本趋同,AF3 略占优势。
RNA
OF3 表现最佳的模式:在所示指标上达到或超过 AF3。 优势可能源于数据准备:在裁剪过程中,仅将聚合物计入 20 条链的预算(不计入离子/小配体),从而保留了 RNA 的上下文信息而无需过度采样。
未来何去何从?
除了在 RNA 结构预测方面达到的最高精度结果外,此次发布的 OpenFold3 预览版基本符合预期结果,但它为下一代生物分子结构预测器可能的样子提供了一个模板。一些具体目标包括实现与 AF3 的性能对等,这将需要使用整个 PDB(蛋白质数据库)进行完整的重新训练。其他应用可能包括类似 Boltz-2 风格的蛋白质-配体结合亲和力预测、构象集合预测、用于虚拟筛选的快速推理等。 一个不太明显但需要克服的障碍(尤其是对于抗体而言)是匹配 AF3 通过额外采样提升的效能。DeepMind 团队发现,对相同的输入生成大量结构(例如 100 到 1000 个样本)并挑选最佳的一个,能显著提高对接构象预测的准确性。OpenFold3(以及 Chai 或 Boltz)目前尚未展现出这种收益。
作者对下一代结构预测器做一些推测:
物理原理可能在推理中扮演更重要的角色。机器学习模型擅长捕捉进化线索,但当这些信息未在训练数据中体现时,它就无能为力了。刚性、约束良好的区域在很大程度上已被解决;难点在于灵活、功能性的区段。我们必将看到一些扩散模型组件,用于引导在结构空间中的探索;受分子动力学(MD)启发的目标函数;以及用于结构优化的局部能量最小化方法。
多序列比对(MSA)似乎会持续存在,尽管出现了基于语言模型的方法,它们并非消失,而是在进化。虽然大型单序列模型开始内化类似 MSA 的统计信息,但近期的理想方案是较小的模型结合检索机制,在推理时引入 MSA,而非依赖庞大的单体记忆模型。
模型校准也将得到改进,方法是使用明确的负样本(非相互作用的蛋白质对和无序区域)进行训练,以抑制假阳性蛋白质相互作用(PPI)以及在预期无序区域出现过度自信的螺旋结构预测,从而使置信度反映生化现实,而非假设输入是一个蛋白质/稳定复合物。
代码链接
https://github.com/aqlaboratory/openfold-3
部署文档
https://openfold-3.readthedocs.io/en/latest/index.html
英伟达部署链接
https://build.nvidia.com/openfold/openfold3
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-29,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

