Nat. Methods | 利用DNA基础模型实现单核苷酸分辨率的基因组注释
Nat. Methods | 利用DNA基础模型实现单核苷酸分辨率的基因组注释

DrugOne
发布于 2025-11-17 20:52:21
发布于 2025-11-17 20:52:21
DRUGONE
基因组注释模型能够直接分析DNA序列,对现代生命科学研究至关重要。研究人员将基因组注释问题重新定义为多标签语义分割任务,并提出了一种基于预训练DNA基础模型的细粒度注释方法。该方法通过微调 Nucleotide Transformer (NT) 模型,开发出 SegmentNT,能够在单核苷酸分辨率下对14种基因与调控元件进行预测。SegmentNT 可以处理最长达 50 kb 的DNA序列,在基因注释、剪接位点检测及调控元件识别中均实现了当前最优性能。研究人员还集成了 Enformer 和 Borzoi 两种基础模型,将序列上下文扩展至 500 kb,进一步增强了调控元件检测能力。此外,基于人类基因组训练的 SegmentNT 模型可直接迁移到其他物种,并通过多物种联合训练显著提升了跨物种泛化能力。该框架为大规模基因组注释提供了一种可扩展的通用方法。

现代生物学的核心任务之一是准确注释DNA序列中的功能区域,包括基因结构(外显子、内含子、UTR)和调控元件(启动子、增强子等)。传统的注释工具(如 HMM 模型 Genscan、AUGUSTUS)虽然具备单核苷酸级分辨率,但在预测复杂的可变剪接、基因异构体及跨染色体特征时性能有限,且依赖外部实验数据辅助修正。同时,针对特定功能元素(如启动子、增强子或poly(A)信号)的模型通常仅在小样本数据集上训练,导致在真实基因组场景中泛化性不足。
近年来,大规模 DNA 基础模型的出现为解决这些问题提供了新途径。这类模型通过自监督学习在数十亿碱基序列上预训练,能够捕获序列的语法和调控规律。借鉴计算机视觉中的图像分割思想,研究人员将DNA序列分割任务视为“像素级标注”,以SegmentNT 模型为核心,实现了对基因组功能元件的自动注释。
方法
研究人员基于 Nucleotide Transformer (NT) 的预训练嵌入构建 SegmentNT 框架,并在人工标注的人类基因组数据集上微调。
数据来源包括 GENCODE 和 ENCODE,覆盖 14类功能元素:蛋白编码基因、长链非编码RNA、外显子、内含子、5′UTR、3′UTR、剪接供体/受体位点、poly(A)信号、组织特异及非特异启动子、增强子以及CTCF结合位点。
SegmentNT 的神经网络采用 一维U-Net结构 作为分割头,对每个碱基计算其属于不同元素的概率分布。模型在3 kb、10 kb、20 kb、30 kb及50 kb 序列长度下训练与测试,并通过旋转位置编码插值(RoPE)扩展上下文建模范围。
结果
SegmentNT模型架构与性能评估
SegmentNT 结合了 DNA 编码器(NT)与一维U-Net分割头,用于同时预测14类基因组元素的碱基掩码。模型在3 kb与10 kb输入上训练,平均 MCC 达0.37–0.42。在外显子、UTR及启动子识别中表现最佳。
研究人员在 NOP56 与 IDH3B 基因区域的测试中验证模型预测,SegmentNT 能准确标出蛋白编码区、UTR、剪接位点及增强子区域。
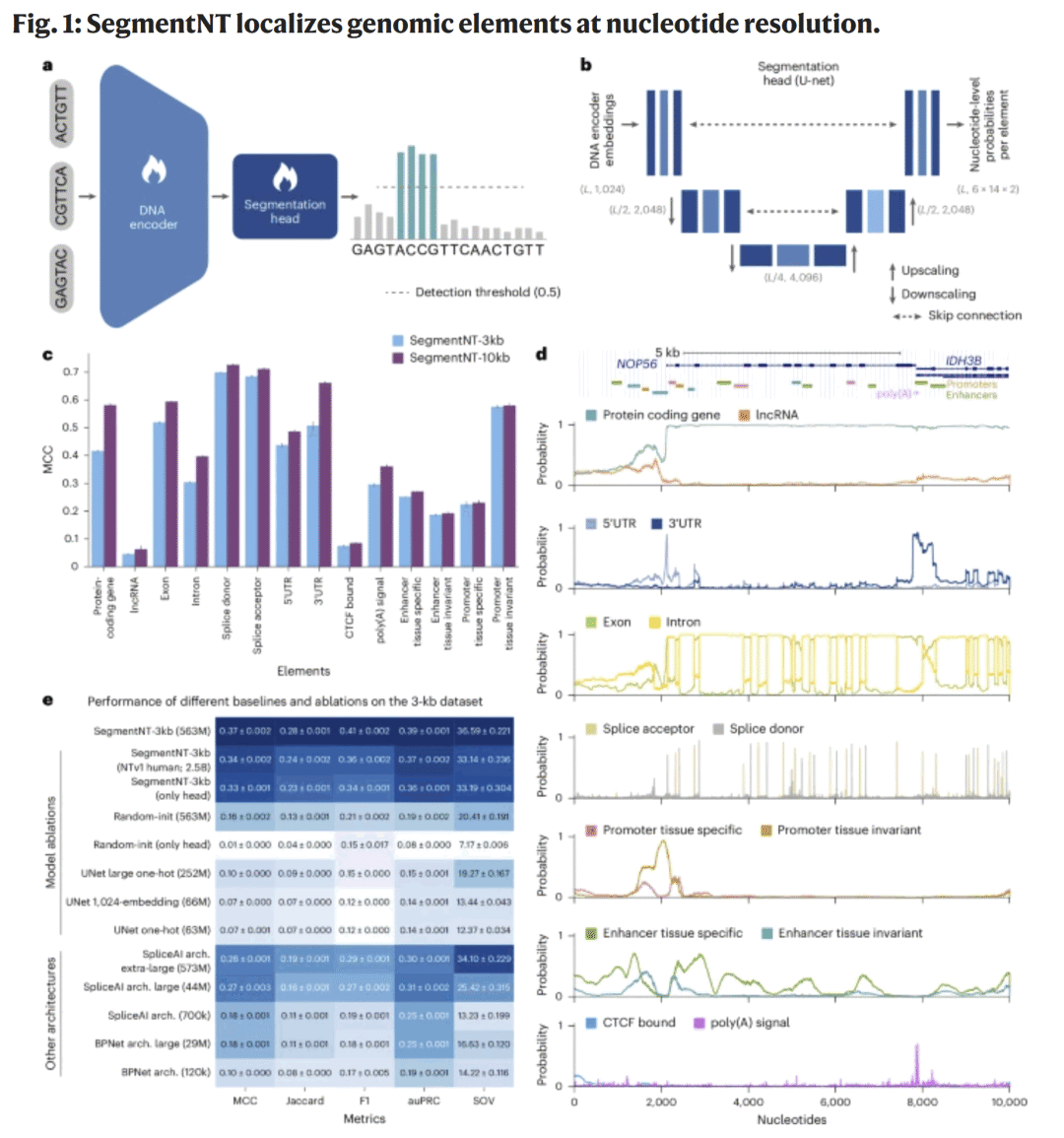
图1|SegmentNT 在单核苷酸分辨率下定位基因组元素
上下文扩展与长序列建模
为提升模型对长距离依赖的捕获能力,研究人员引入 RoPE频率重标定插值,将输入序列扩展至30 kb及50 kb。
结果显示,SegmentNT-30kb 在各元素上的平均 MCC 达0.45,对外显子与UTR预测尤为显著。利用上下文重标定后,模型在100 kb序列上仍能保持 MCC 0.45。
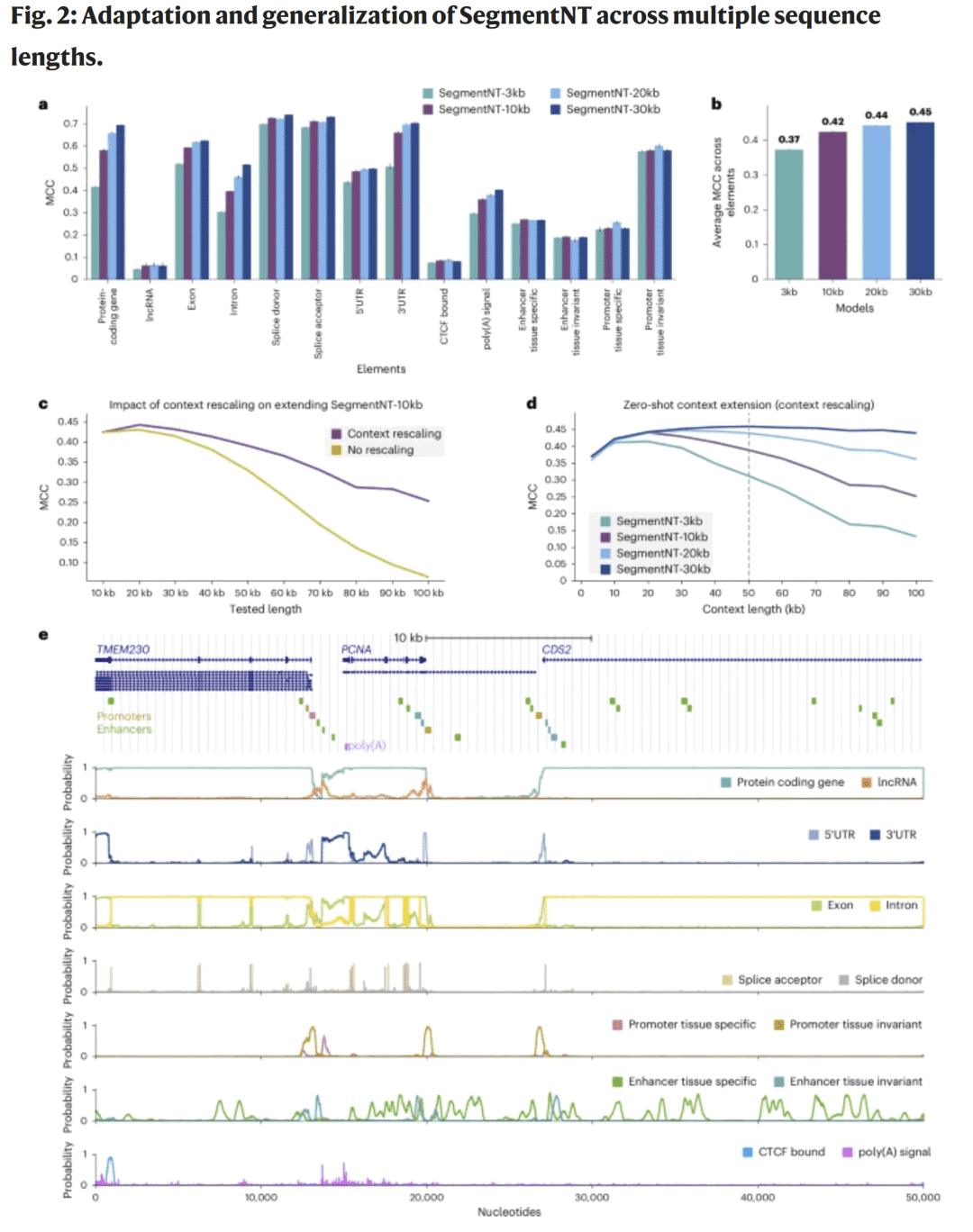
图2|SegmentNT 在不同序列长度下的泛化与扩展能力
整合不同基础模型以实现500 kb注释
SegmentNT 框架可无缝兼容其他DNA基础模型。研究人员引入 Enformer 与 Borzoi 作为DNA编码器,分别将输入上下文扩展至196 kb与524 kb。
结果表明:
- SegmentNT 在基因结构识别上表现最佳;
- SegmentEnformer 与 SegmentBorzoi 在增强子与CTCF结合位点预测中更具优势。
这种差异源于不同预训练数据类型(转录组 vs 表观组学)。
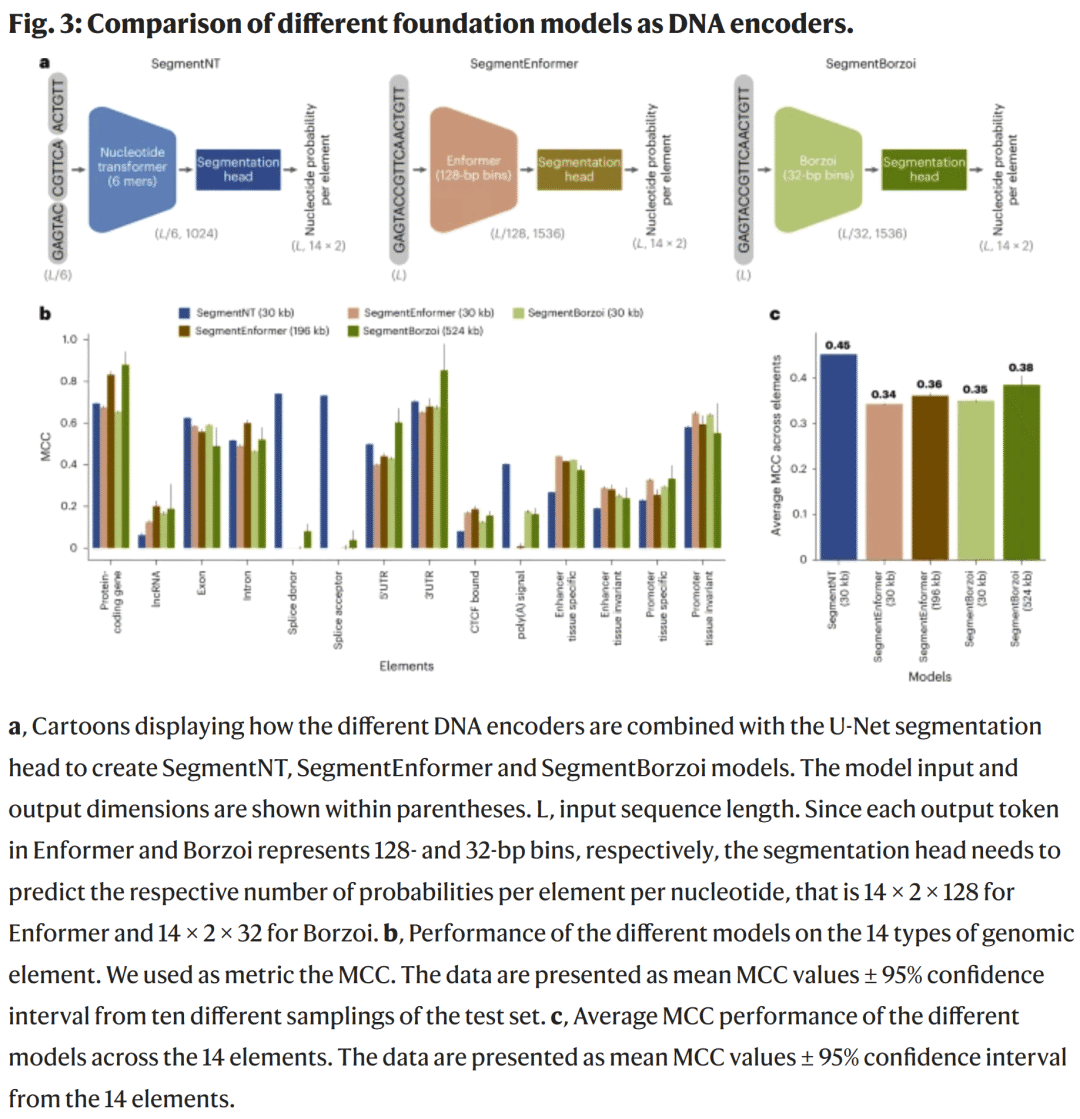
图3|不同DNA基础模型在SegmentNT框架中的对比
与AUGUSTUS基因注释工具比较
研究人员将 SegmentNT-30kb 与传统HMM模型 AUGUSTUS 在基因注释任务上对比。在单异构体场景下,两者性能相当;而在多异构体及整条染色体级别测试中,SegmentNT-30kb 在所有指标上均超越AUGUSTUS。尤其在外显子、内含子与剪接位点,SegmentNT 的召回率与精确率均更高。
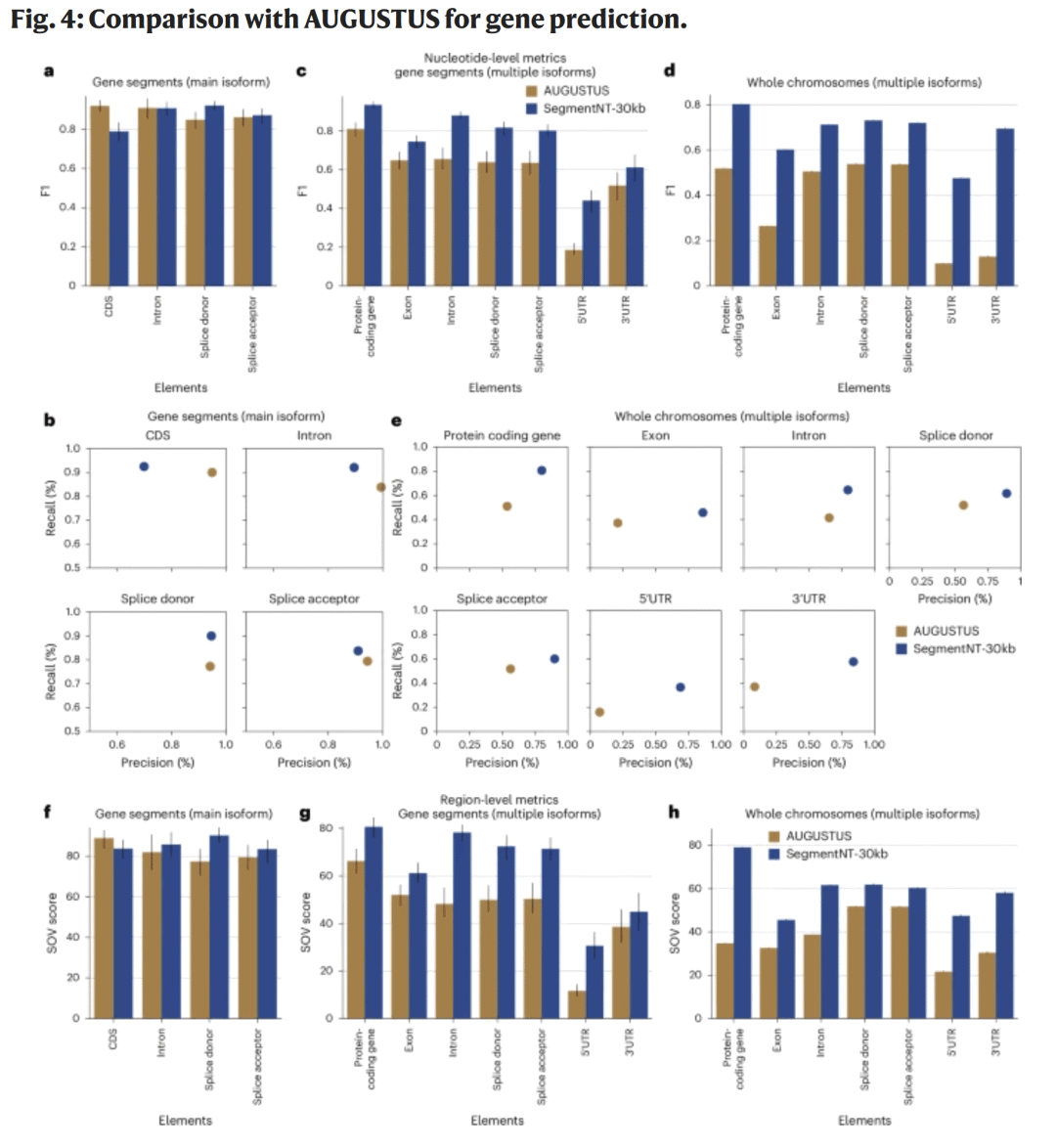
图4|SegmentNT 与 AUGUSTUS 在基因注释中的对比
剪接位点预测性能
SegmentNT-30kb 在剪接供体与受体检测中与 SpliceAI、Pangolin 等模型比较。结果表明,SegmentNT 能正确定位所有真实剪接点,且假阳性显著减少。其在全基因组层面的 auPRC 达0.93–0.94,与专业模型持平甚至略优。
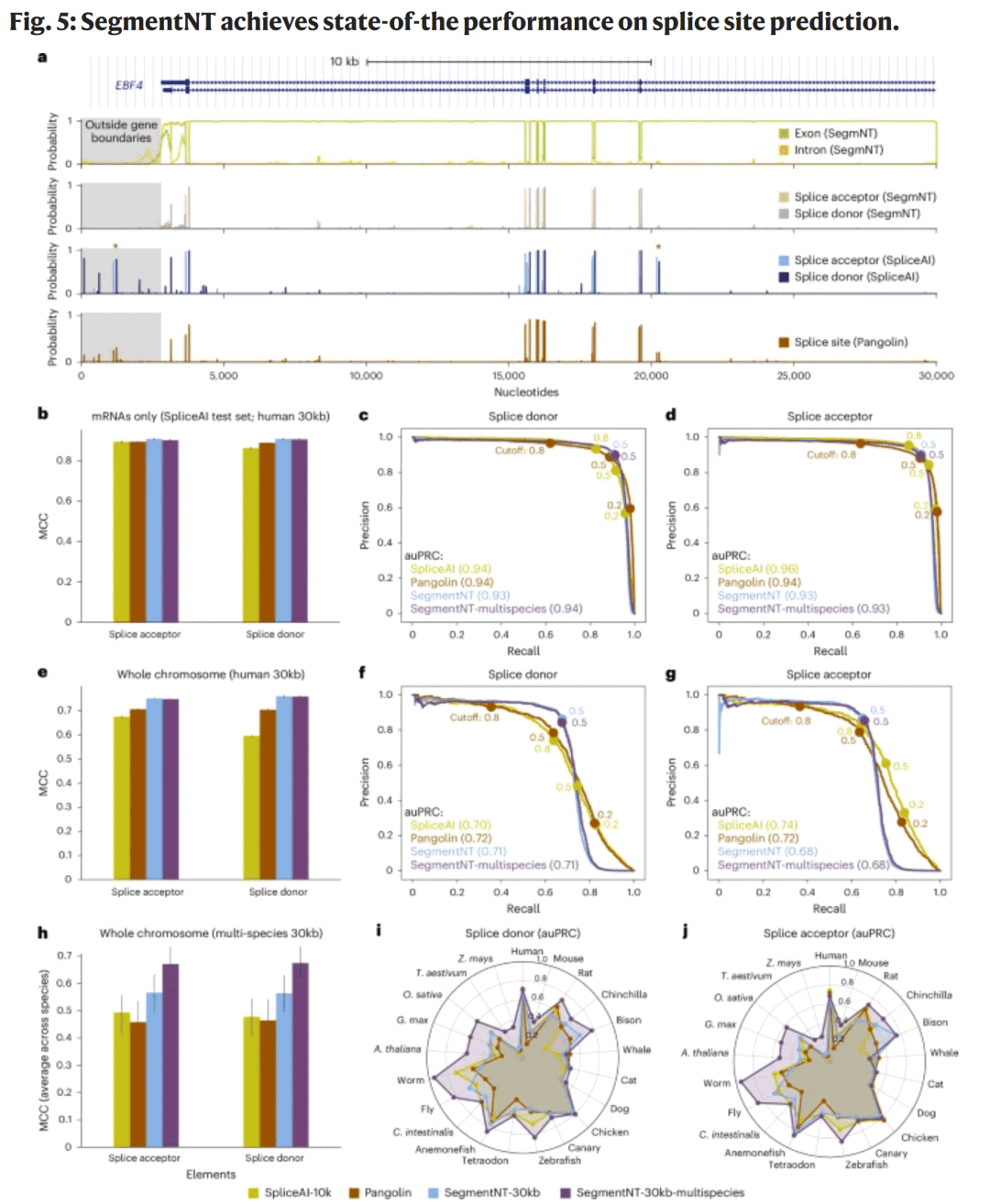
图5|SegmentNT 在剪接位点检测中的表现
跨物种泛化能力
研究人员评估了人类训练的 SegmentNT 模型在 15种动物和5种植物基因组 上的泛化性能。
结果显示:
- 在灵长类与哺乳动物中,模型对外显子与剪接位点预测保持高精度(MCC>0.6);
- 在远缘物种与植物中,性能下降但仍能识别主要基因结构。
多物种联合训练(SegmentNT-MS)进一步提升了跨物种表现。
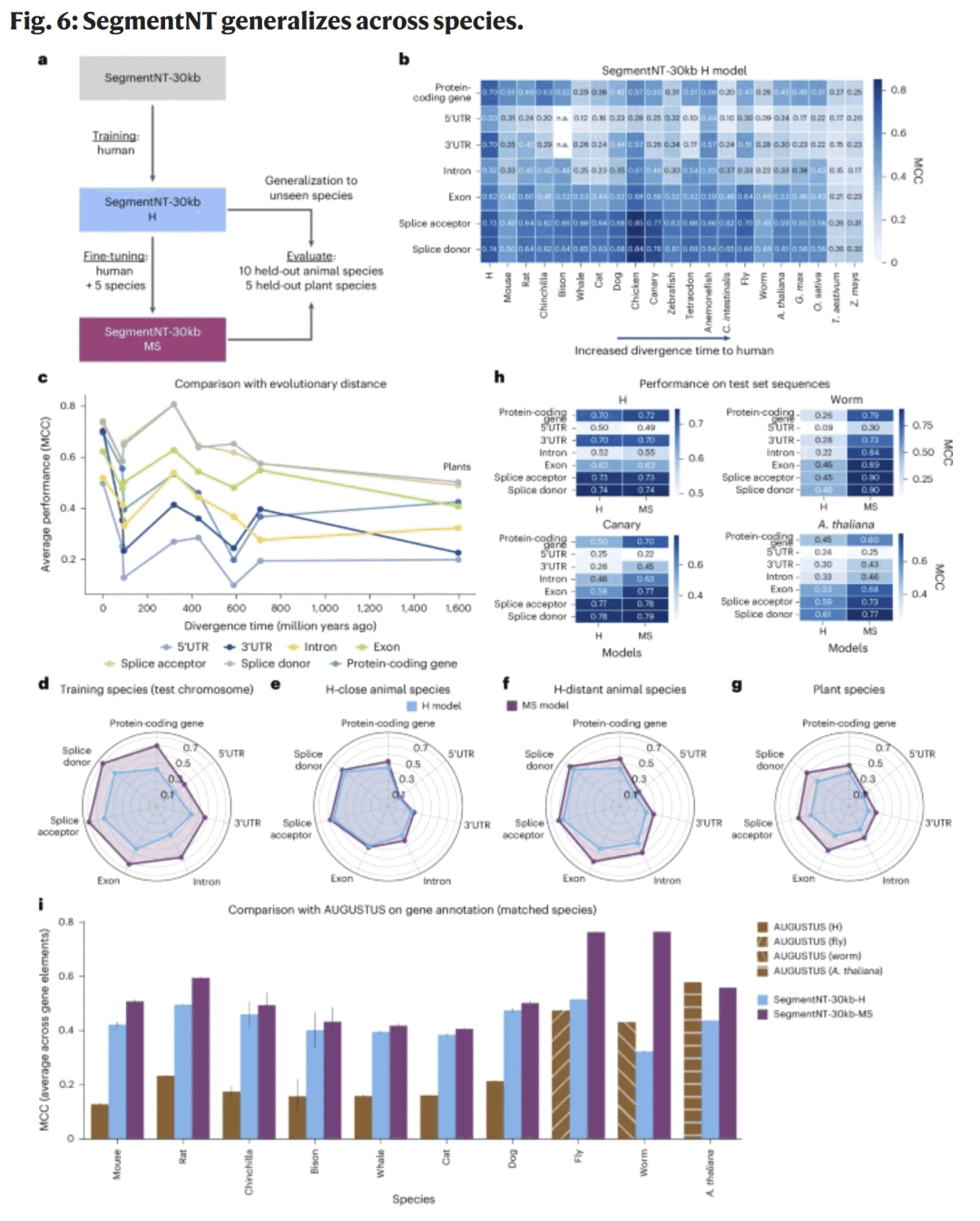
图6|SegmentNT 在多物种基因组上的泛化表现
讨论
SegmentNT 的提出标志着基因组注释进入“单核苷酸精度”的新阶段。研究人员表明,预训练DNA基础模型可显著提升多任务性能,尤其在多类型元素同时预测时展现优异的泛化与稳定性。该模型的可扩展框架允许与更长上下文DNA编码器(如Enformer、Borzoi)结合,实现从局部基因结构到远程调控的统一建模。
此外,SegmentNT 的跨物种适应性揭示了DNA语言模型中蕴含的“进化一致性”。这为未注释物种的基因功能推断及调控元件预测提供了强大工具。
未来方向包括:
- 结合表观组学和转录组信息,建立多模态联合注释模型;
- 利用模型生成能力设计人工调控元件;
- 构建统一的“基因组语言空间”,支持不同生物体系的共享注释。
SegmentNT 证明了 DNA 基础模型能够跨越实验与计算的边界,为精准基因组学与合成生物学奠定了新基础。
整理 | DrugOne团队
参考资料
de Almeida, B.P., Dalla-Torre, H., Richard, G. et al. Annotating the genome at single-nucleotide resolution with DNA foundation models. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02881-2
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-31,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

