Nat. Commun. | 用重构的蛋白生成模型预测蛋白稳定性
Nat. Commun. | 用重构的蛋白生成模型预测蛋白稳定性

DrugOne
发布于 2025-12-25 15:05:20
发布于 2025-12-25 15:05:20
DRUGONE
蛋白稳定性变化预测对疾病机制解析与蛋白工程至关重要,但受限于数据偏倚与模型泛化能力,长期难以规模化应用。研究人员提出 SPURS,一种通过模型重构(rewiring)整合蛋白语言模型与逆折叠模型的深度学习框架,并在超大规模稳定性数据上进行高效微调。SPURS 在多个独立数据集上实现了更高准确性、更强泛化性与更优计算效率,不仅可预测单点与多点突变的稳定性变化,还可拓展至功能位点识别、低样本蛋白适应度预测以及人类疾病变异机制解析。

蛋白稳定性通常以突变引起的热力学变化来衡量,是决定蛋白折叠、功能与疾病相关性的核心因素。尽管实验手段如定向进化有效,但成本高昂、覆盖有限。现有机器学习方法受制于小规模训练数据、结构或序列信息利用不充分,在未知蛋白或稀有突变上的泛化能力不足。近年来,大规模蛋白生成模型在无监督任务中展现出潜力,但直接微调用于稳定性预测常导致过拟合或性能退化,亟需新的整合范式。
方法
SPURS 通过重构策略将序列驱动的蛋白语言模型与结构驱动的逆折叠模型进行深度融合。研究人员在语言模型内部引入轻量级适配模块,使其能够吸收结构模型学习到的几何稳定性特征,同时保留进化先验。该设计仅需更新极少量参数即可完成微调,并支持在一次前向传播中同时预测所有可能单点突变的稳定性变化,显著提升可扩展性。
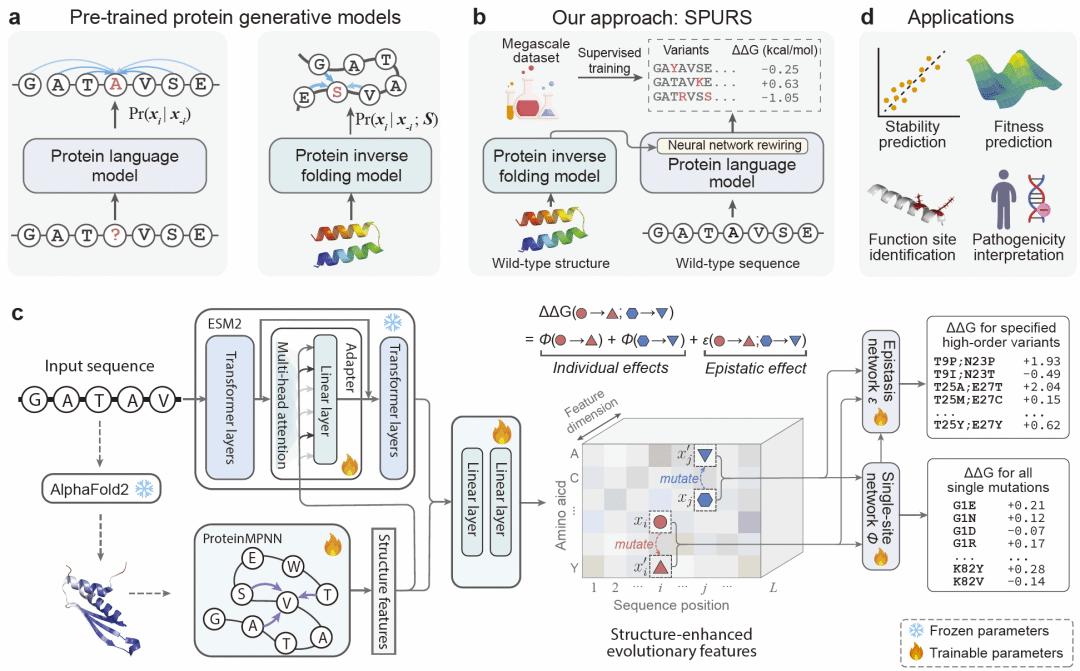
图1| SPURS 模型结构。
结果
蛋白稳定性预测的准确性与泛化性
在 Megascale 及多个独立稳定性数据集上,SPURS 在相关性与误差指标上系统性优于现有主流方法,尤其在未见蛋白与未见突变场景中保持稳健性能。
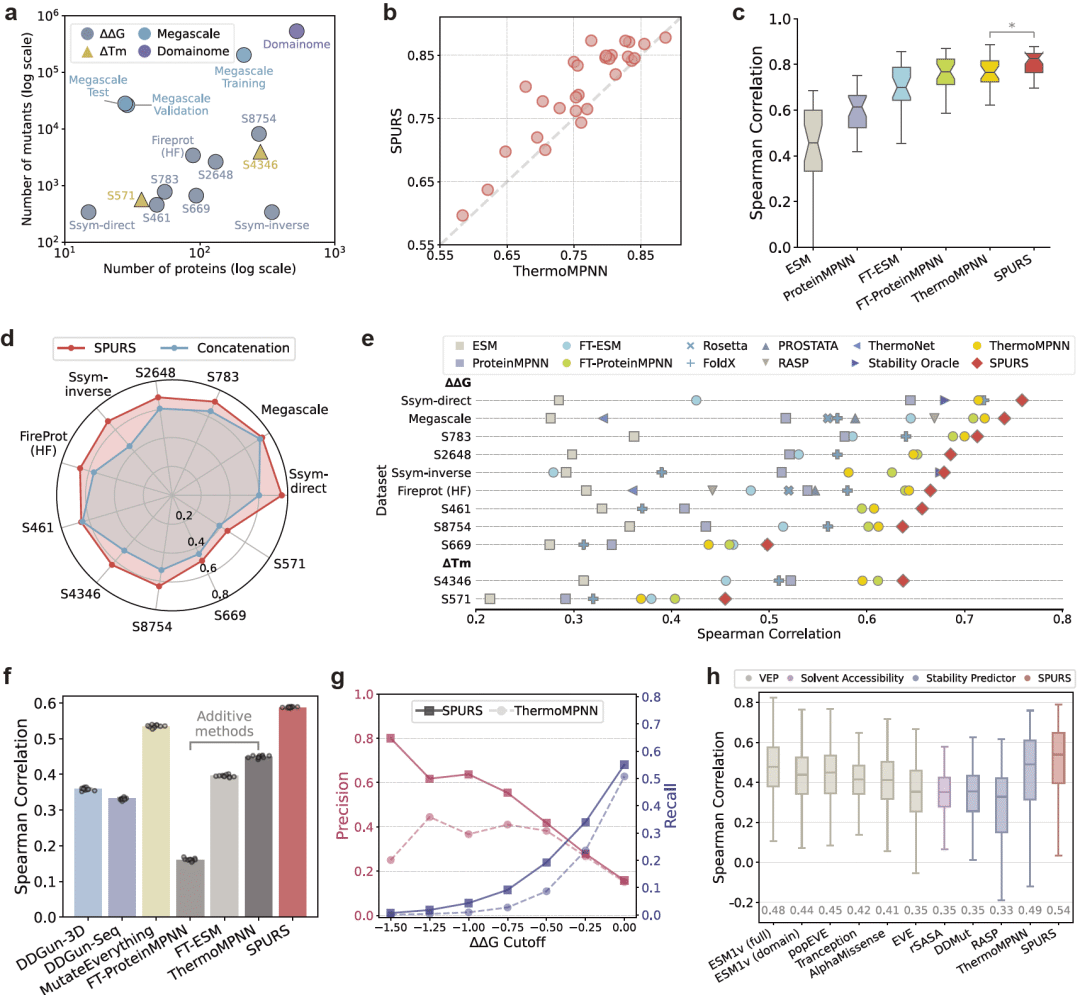
图 2|SPURS 实现高精度的蛋白稳定性预测。
稳定突变识别与多突变建模
SPURS 能更准确地区分稳定化与不稳定化突变,并在多点突变预测中显著优于简单叠加模型,显示其对非加性突变效应的有效刻画能力。
蛋白功能位点的无监督识别
结合稳定性预测与进化约束信号,SPURS 能在无需功能标注的情况下,准确定位结合界面与关键功能残基,并在多个蛋白结构实例中得到验证。
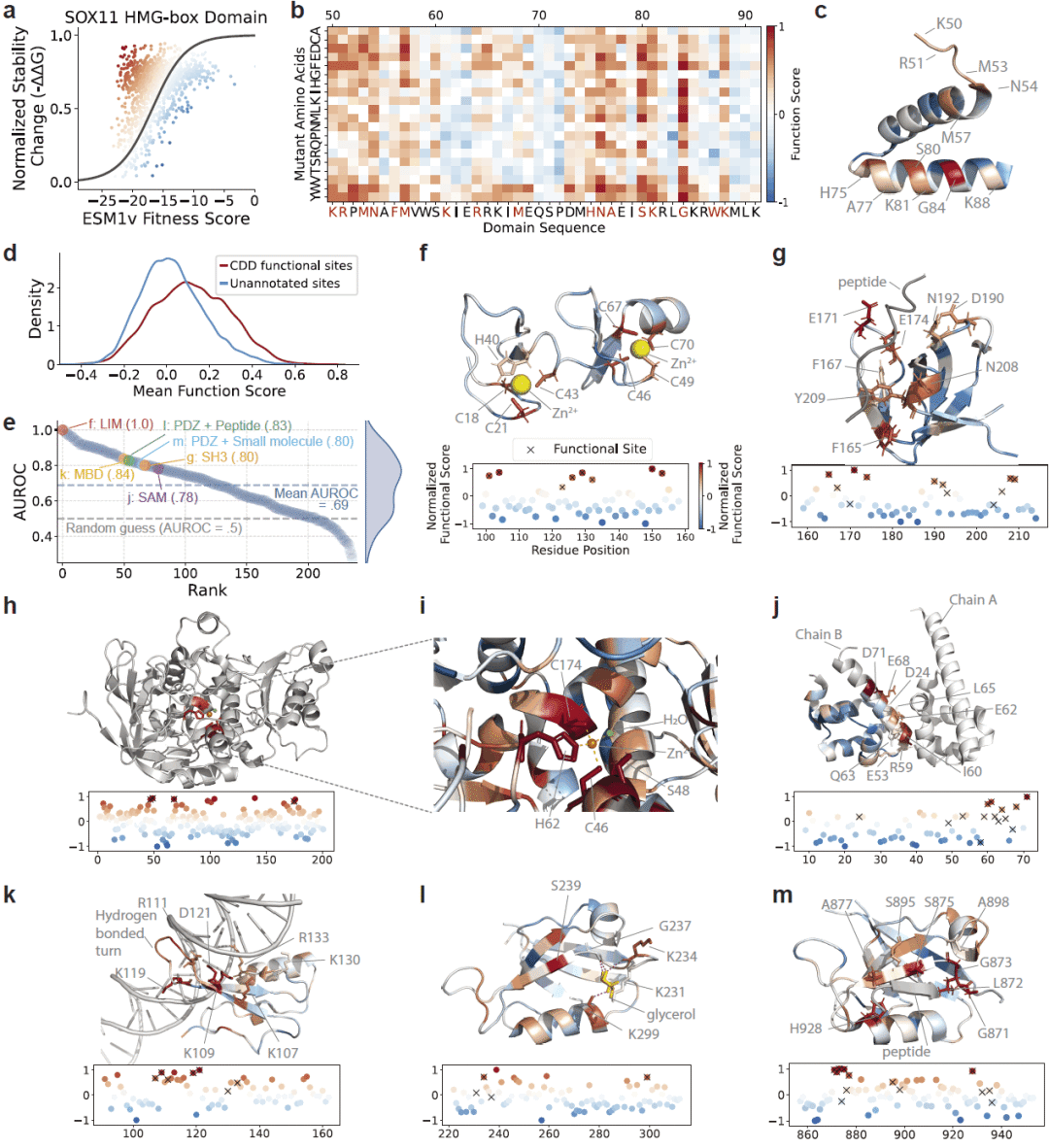
图 3|SPURS 准确指导蛋白功能位点注释。
低样本蛋白适应度预测增强
将 SPURS 的稳定性预测作为先验信息引入低样本适应度预测模型后,在多种深度突变扫描数据集中均显著提升预测性能,尤其在表达量与整体适应度任务中效果突出。
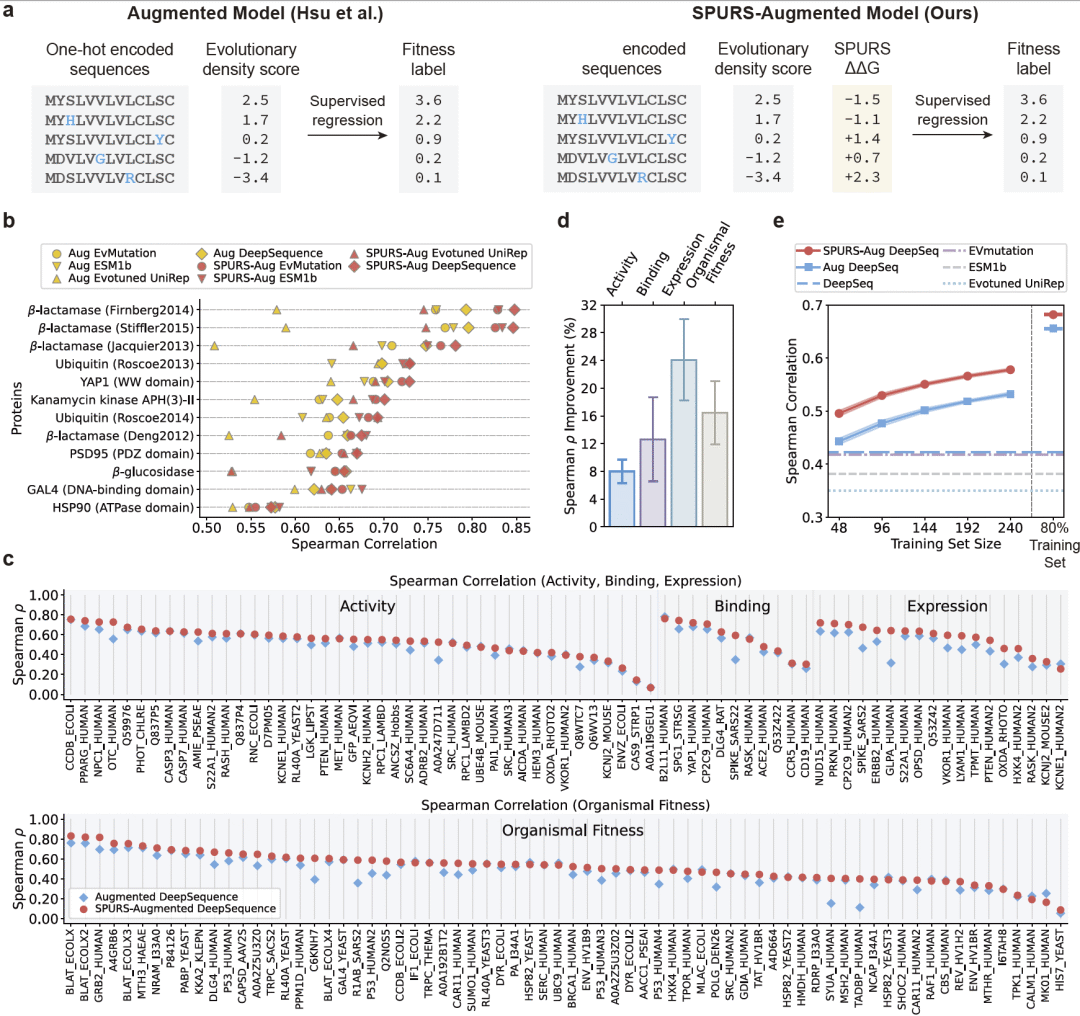
图 4|SPURS 增强模型提升低样本(low-N)蛋白适应度预测性能。
稳定性在疾病致病机制中的作用
对人类蛋白组的系统分析表明,致病突变整体上更具破坏性稳定性效应,且这种效应与残基埋藏程度和遗传模式密切相关。SPURS 揭示了不同疾病基因中稳定性损失与功能扰动的差异机制。

图 5|SPURS 揭示蛋白去稳定化在变异致病性中的作用。
讨论
SPURS 展示了一种将蛋白生成模型从“生成”转向“预测”的通用范式。通过高效融合序列进化信息与结构几何约束,该方法在稳定性预测任务中实现了前所未有的泛化性与可扩展性。其一次前向传播覆盖全突变空间的能力,使蛋白组尺度分析成为现实。未来,SPURS 有望作为稳定性预测的通用基础模型,与蛋白设计、变异致病性评估及生成式模型协同,进一步推动蛋白工程与精准医学的发展。
整理 | DrugOne团队
参考资料
Li, Z., Luo, Y. Generalizable and scalable protein stability prediction with rewired protein generative models. Nat Commun (2025).
https://doi.org/10.1038/s41467-025-67609-4
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-22,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

