Proc. Natl. Acad. Sci. | 基于 AlphaFold3 的条件扩散,实现精准位点特异性蛋白折叠
Proc. Natl. Acad. Sci. | 基于 AlphaFold3 的条件扩散,实现精准位点特异性蛋白折叠

DrugAI
发布于 2026-01-06 13:46:46
发布于 2026-01-06 13:46:46
DRUGONE
精准预测生物大分子复合物结构是理解生命过程与药物发现的关键。尽管 AlphaFold3(AF3)在复合物预测方面取得重大进展,但在特定位点(如结合口袋或变构位点)的预测精度仍存在不足。研究人员提出 SiteAF3,一种基于 AlphaFold3 框架的条件扩散方法,用于实现高精度的位点特异性折叠。SiteAF3 通过固定受体结构并可选性地整合结合口袋及热点残基信息,优化了扩散过程。在蛋白–小分子、蛋白–肽段及蛋白–核酸等多类数据集上,SiteAF3 的预测精度均显著优于 AlphaFold3,尤其在孤儿蛋白及变构配体的结构预测中表现突出,同时减少计算开销。该模型以插件形式兼容 AlphaFold3,为高精度生物分子互作建模提供了实用工具。

准确预测生物分子复合物结构是解析生命机理与理性药物设计的核心环节。AlphaFold2 在单蛋白结构预测上实现了突破性成功,而 AlphaFold3 进一步扩展至蛋白–蛋白、蛋白–核酸、蛋白–配体等多种类型复合物,为计算生物学带来了新范式。然而,AF3 仍存在若干挑战。其性能高度依赖于多序列比对(MSA),对序列同源性低的孤儿蛋白或错误MSA可能出现显著偏差。此外,AF3 的共折叠模型在预测变构配体时精度不足;其构象采样仍耗时,GPU 内存需求高;并且 AF3 的口袋引导模块未开放,限制了其在特定靶点下的可定制性。
为此,研究人员开发了 SiteAF3,通过在 AlphaFold3 框架中引入 条件扩散(conditional diffusion)机制 与 口袋/热点信息引导,实现受体固定条件下的高精度位点特异性折叠。这一方法显著提升了复合物结构预测的准确性与可控性。
方法概述
SiteAF3 基于 AF3 框架改进了两个核心模块:
- 条件扩散模块:在扩散过程中固定受体结构,仅对配体原子坐标进行噪声采样与更新,使采样集中于目标结合位点附近。
- 口袋与热点信息引导:在 MSA 模块中引入结合口袋与关键残基信息,作为条件输入,增强局部位点的结构约束与定位精度。
噪声初始化以口袋中心为高斯分布核心,同时引入随机旋转与平移以增加构象多样性。配体的更新通过掩码机制限制在可变部分,从而降低显存消耗并增强对大体系的适用性。模型训练与推理均保持与 AF3 兼容,用户可将其作为轻量化插件直接调用。
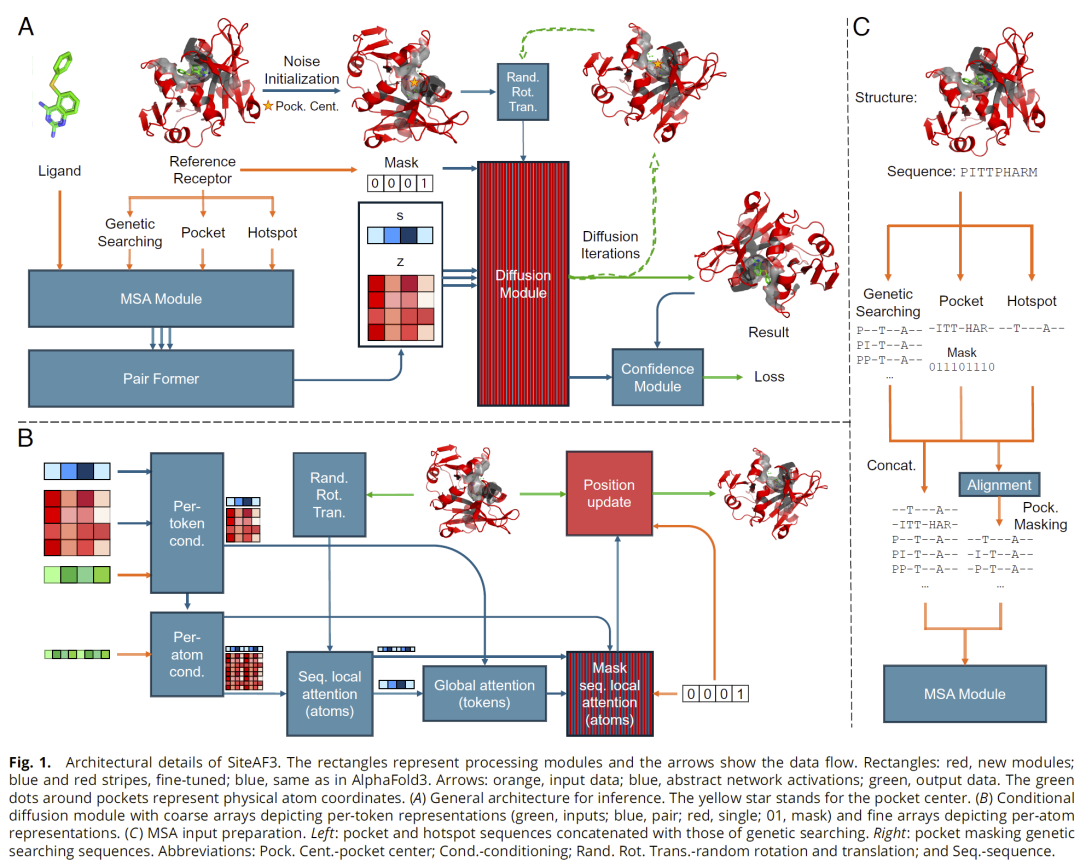
结果
SiteAF3 网络架构与关键改进
SiteAF3 的总体架构继承 AF3 的推理流程,包括输入准备、表示学习、结构预测与置信度评估。主要改进体现在:
- 条件扩散机制:仅更新配体坐标,受体固定;
- 局部注意力掩码机制:优化显存占用并加速训练;
- 位置更新模块:将固定受体与预测配体无缝结合形成完整复合物。
这些改动确保了模型能在已知受体结构条件下高效搜索配体构象空间,特别适用于特定靶点的结合预测。
蛋白–小分子复合物预测
在 FoldBench 与 PoseBustersV2 数据集上,SiteAF3 的成功率分别为 71.6% 与 81.2%,均显著高于 AF3(62.0% 和 73.1%)。模型在五种配置模式下进行比较,其中“pocket-masked AF3 MSA”模式表现最佳,平均 LRMSD 降低 30%以上。在变构配体(allosteric ligands)预测中,SiteAF3 准确识别了 CK2α–LVF 等变构结合位点,而 AF3 将其错误定位至正构口袋。此外,SiteAF3 对孤儿蛋白和含辅因子体系同样表现出更高鲁棒性与泛化性。
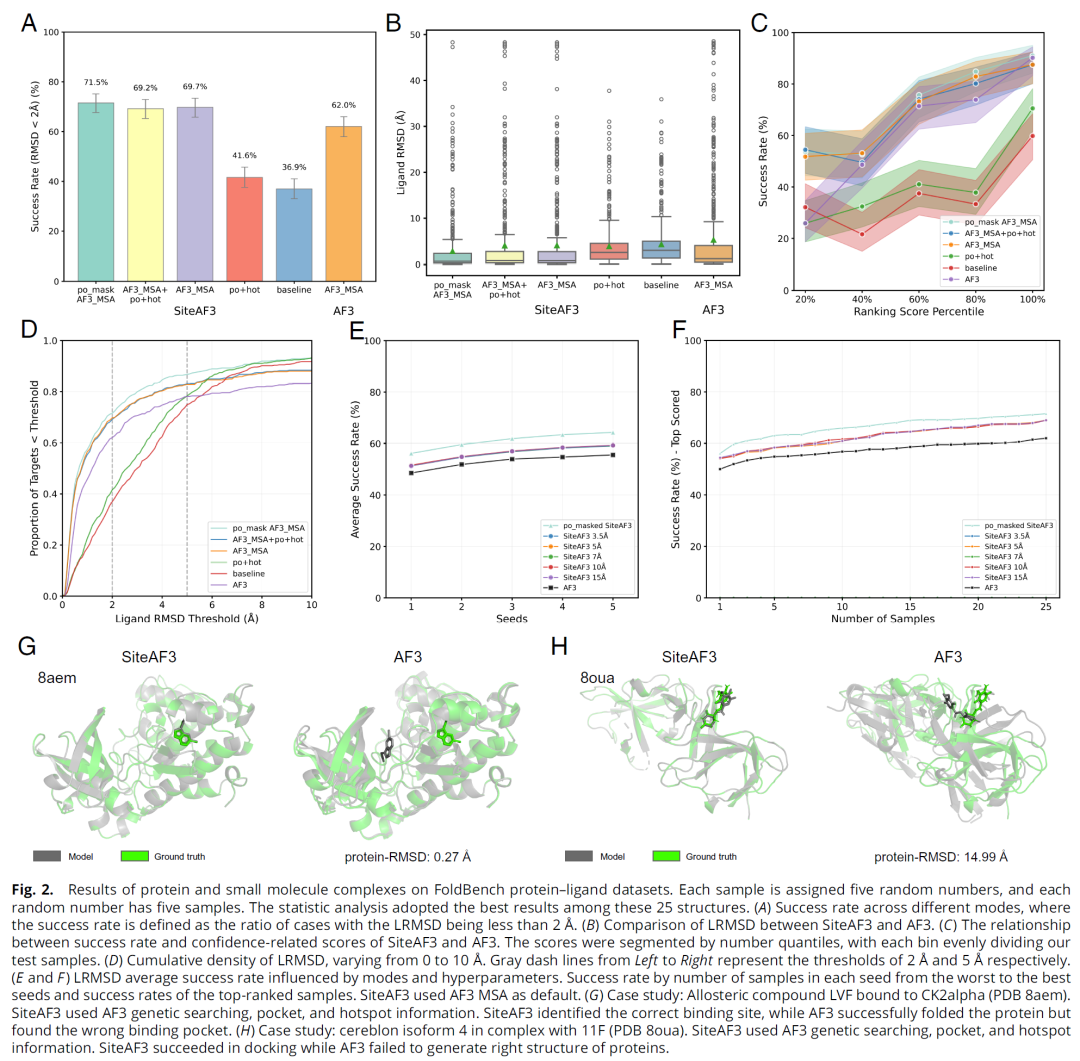
蛋白–肽段复合物预测
在 PepPCBench 数据集上,SiteAF3 的平均 DockQ 得分比 AF3 提高 11.2%,中位数提高 7.0%。研究人员进一步分析了不同肽段长度的影响:
- 短肽(5–9 aa)与中肽(10–15 aa)提升最显著;
- 长肽(16–30 aa)提升较小。
这与模型以口袋中心为噪声核心的初始化策略相一致。
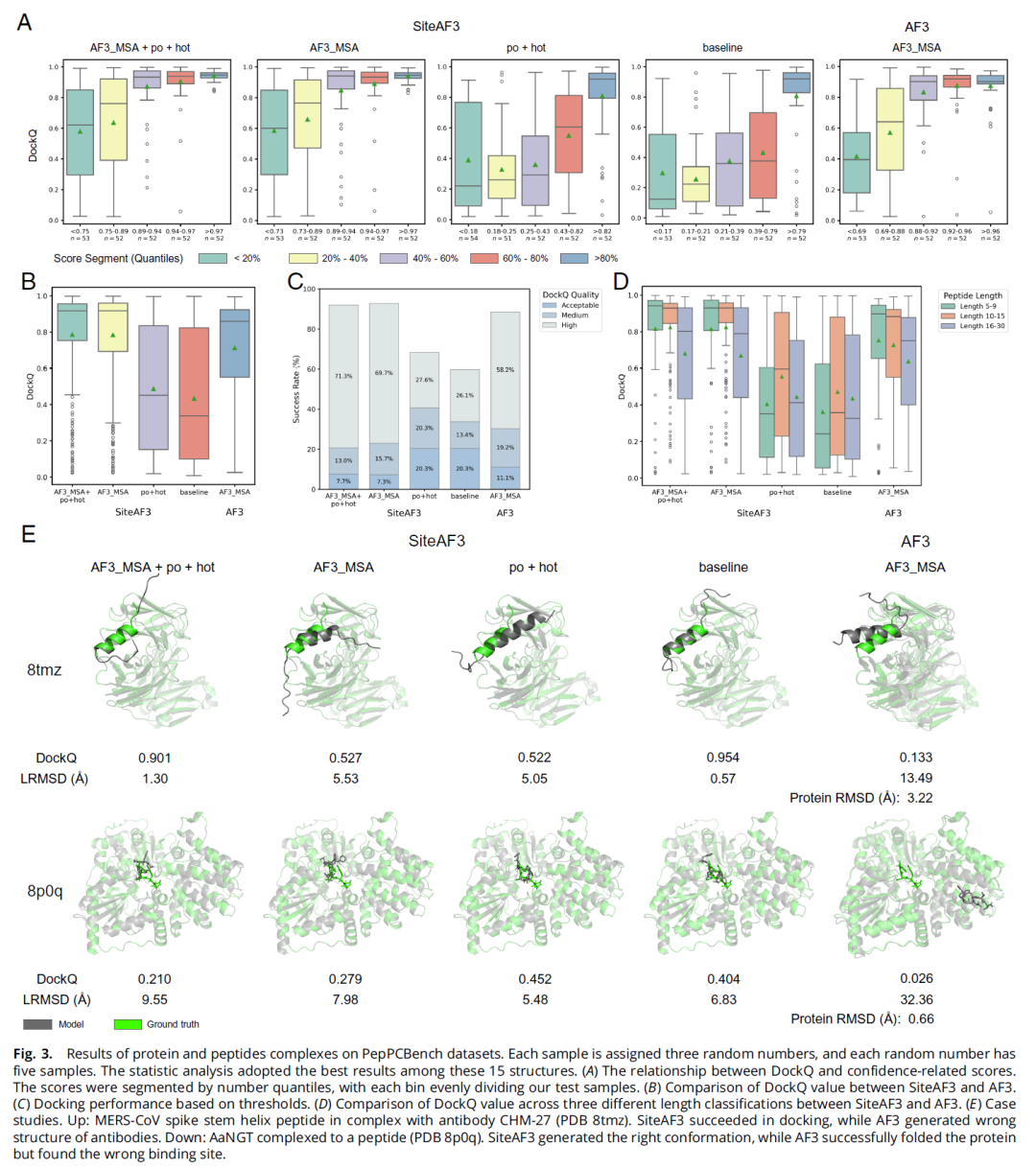
蛋白–核酸复合物预测
在蛋白–dsDNA 与蛋白–RNA 数据集中,SiteAF3 平均 DockQ 分别提高 10.2% 与 14.7%,中位数提升 13.6% 与 44.0%。结合口袋与热点残基信息可进一步增强核酸分子定位精度,尤其在 dsDNA 复合物中表现突出。
计算效率与内存优化
SiteAF3 相较 AF3 在以下方面具有显著优势:
- 显存占用减少: 局部掩码矩阵乘法仅更新配体坐标,使可处理体系规模扩大。
- 速度提升: 当不使用 MSA 遗传搜索时,推理时间显著缩短,适用于高通量虚拟筛选。
研究人员指出,在大体系中 SiteAF3 避免了 AF3 常见的显存溢出问题(>3500 tokens),并能在 RTX 6000 (48GB) 环境中稳定运行。
讨论与结论
SiteAF3 在 AlphaFold3 框架下实现了 高精度位点特异性折叠,在多类复合物预测中均显著优于原模型。
其主要优势包括:
- 对变构位点、柔性肽及孤儿蛋白预测表现突出;
- 预测不确定性高的结构中仍保持高精度;
- GPU 占用低、推理速度快、插件式兼容。
研究人员指出,SiteAF3 的条件扩散思想与口袋约束机制可推广至其他共折叠模型(如 Boltz、Chai、Protenix、HelixFold 等),并有望与亲和力预测模块结合,进一步提升结合能估计精度。
未来工作方向包括:
- 将噪声初始化改进为基于口袋形状的可学习策略;
- 引入共价结合预测模块;
- 允许局部受体构象灵活性以模拟诱导契合效应。
总之,SiteAF3 为高精度复合物预测与药物设计提供了一个通用、高效且可扩展的平台,标志着从 AlphaFold3 向“位点感知结构生成”的重要跨越。
整理 | DrugOne团队
参考资料
H. Tang, & J. Wang, Accurate site-specific folding via conditional diffusion based on AlphaFold3, Proc. Natl. Acad. Sci. U.S.A. 122 (44) e2521048122,
https://doi.org/10.1073/pnas.2521048122 (2025).
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-11-09,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

