Nat. Biotechnol. | 无需RNA三级结构的小分子–RNA相互作用预测方法
Nat. Biotechnol. | 无需RNA三级结构的小分子–RNA相互作用预测方法

DrugAI
发布于 2026-01-06 14:29:28
发布于 2026-01-06 14:29:28
DRUGONE
小分子通过结合 RNA 调控其命运与功能,为疾病治疗提供了重要机遇。然而,现有小分子–RNA 相互作用预测方法通常依赖 RNA 的三维结构信息,严重限制了适用范围。研究人员提出 SMRTnet,一种无需 RNA 三级结构、仅基于 RNA 二级结构的小分子–RNA 相互作用预测深度学习框架。该方法融合 RNA 与化学语言模型、卷积神经网络和图注意力网络,通过多模态数据融合实现高精度预测。SMRTnet 在多个实验基准上显著优于现有方法,并在十个疾病相关 RNA 靶点的筛选中验证了 40 个结合分子,亲和力覆盖纳摩尔到微摩尔范围。以 MYC IRES 为例,预测得分与实验验证率高度相关,且其中一个分子在多种癌细胞系中下调 MYC 表达、抑制增殖并促进凋亡。该工作在不依赖 RNA 三级结构的前提下,显著拓展了 RNA 靶点药物发现的可行性。

RNA 作为功能分子,在多种生命过程中发挥核心调控作用,其异常与癌症、遗传病和病毒感染密切相关。相较于蛋白质,RNA 靶点在药物开发中仍处于早期阶段,其中一个关键瓶颈在于 RNA 三级结构难以通过实验手段大规模解析。
虽然近年来已发展出基于分子对接和深度学习的方法预测小分子–RNA 相互作用,但这些方法大多依赖高质量的 RNA 三维结构,限制了其在真实生物医学场景中的适用性。因此,亟需一种摆脱 RNA 三级结构依赖、可扩展至大规模 RNA 靶点的预测方法。
方法概述
SMRTnet 以 RNA 序列与二级结构 以及 小分子 SMILES 表示 作为输入,通过多模态深度学习架构完成相互作用预测。模型主要包括四个模块:
- RNA 编码器:结合 RNA 语言模型与卷积神经网络,提取序列与碱基配对信息;
- 小分子编码器:融合化学语言模型与图注意力网络,捕获化学组成与拓扑结构特征;
- 多模态数据融合模块:利用注意力机制整合 RNA 与小分子特征,学习其相互作用表示;
- 预测模块:输出小分子与 RNA 的结合评分,并可进一步解析潜在结合位点。
模型训练基于从结构数据库与文献中整理的大规模 RNA–小分子相互作用数据,并通过严格的数据划分与集成策略提升泛化能力。
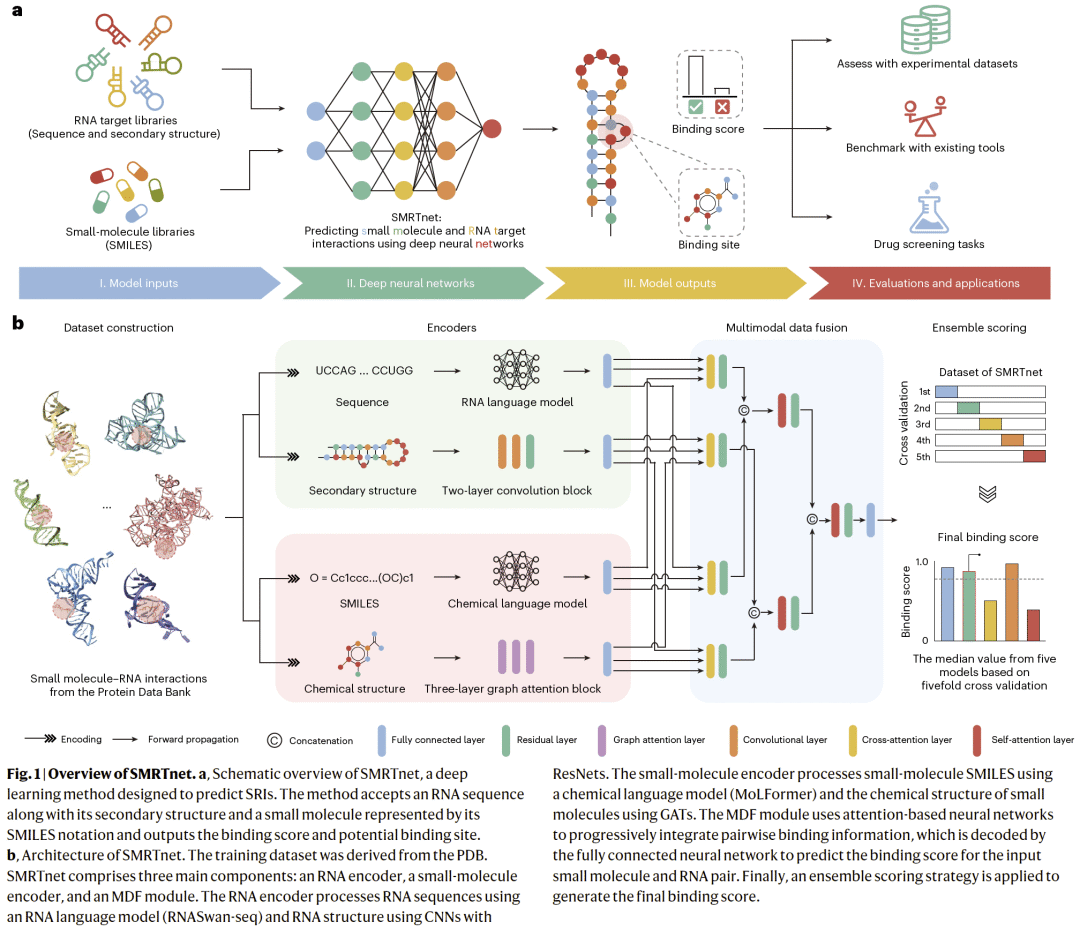
图1|SMRTnet 的整体框架。
结果
SMRTnet 的整体性能
在来源于结构数据库和多个独立实验数据集的基准测试中,SMRTnet 在区分真实结合对与非结合对方面表现稳定,整体预测性能显著优于传统分子对接方法和已有深度学习模型。
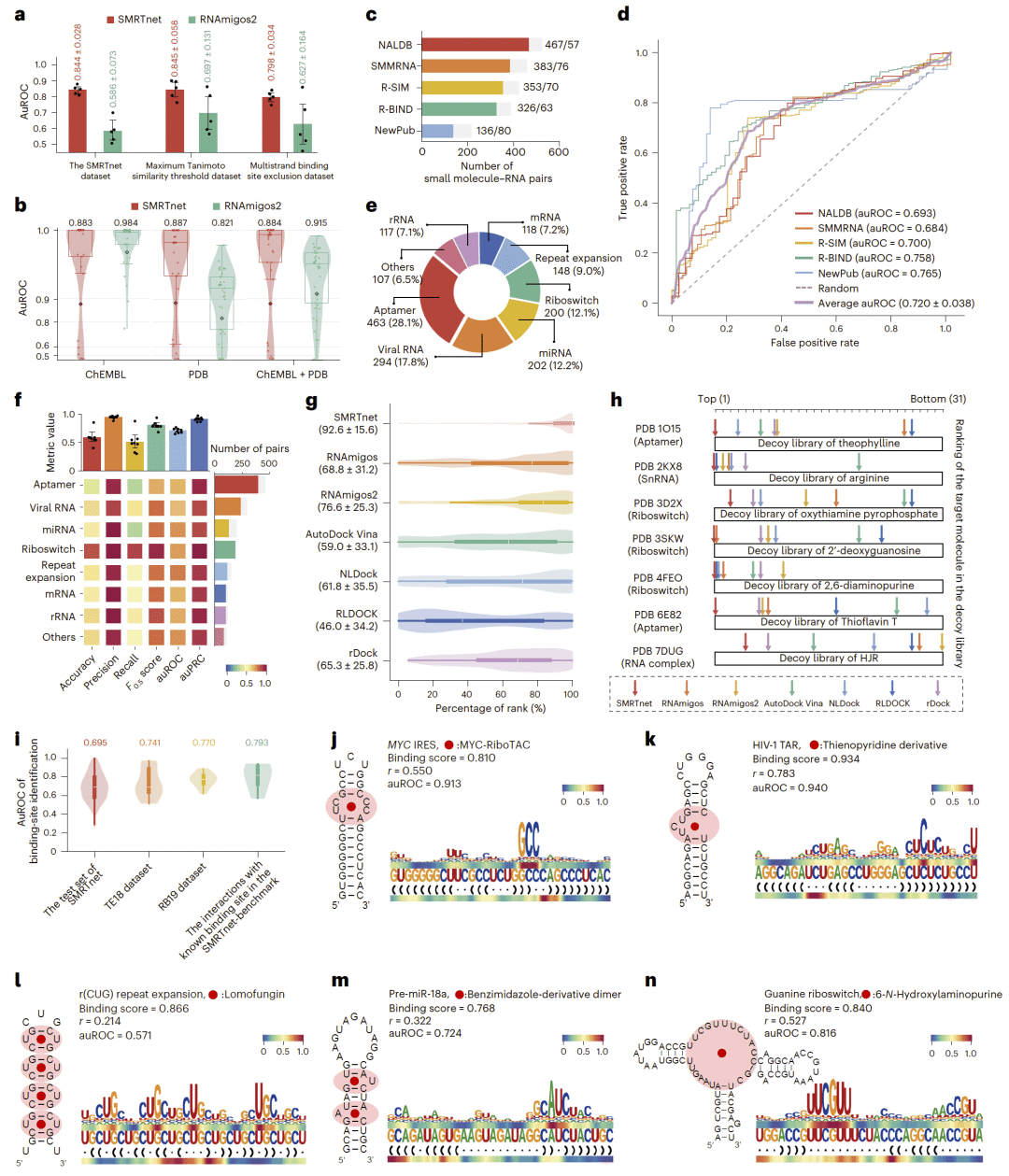
图 2|SMRTnet 的预测性能评估。
RNA 二级结构与模型组件的重要性
消融实验表明,RNA 序列与二级结构信息对预测性能至关重要;移除二级结构会显著降低模型表现。多模态融合模块也对整体性能提升具有重要贡献。
RNA 结合位点的可解释性预测
通过注意力与梯度分析,SMRTnet 可在 RNA 上定位潜在小分子结合位点。预测得到的高关注区域与多种已知实验结合位点高度一致,证明模型不仅能预测是否结合,还能提供空间层面的解释。
疾病相关 RNA 靶点的小分子筛选
研究人员利用 SMRTnet 对十个疾病相关 RNA 靶点进行虚拟筛选,并通过实验验证确认 40 个真实结合分子。不同 RNA 靶点呈现出差异化的结合分子谱,显示模型对 RNA 结构差异具有良好分辨能力。
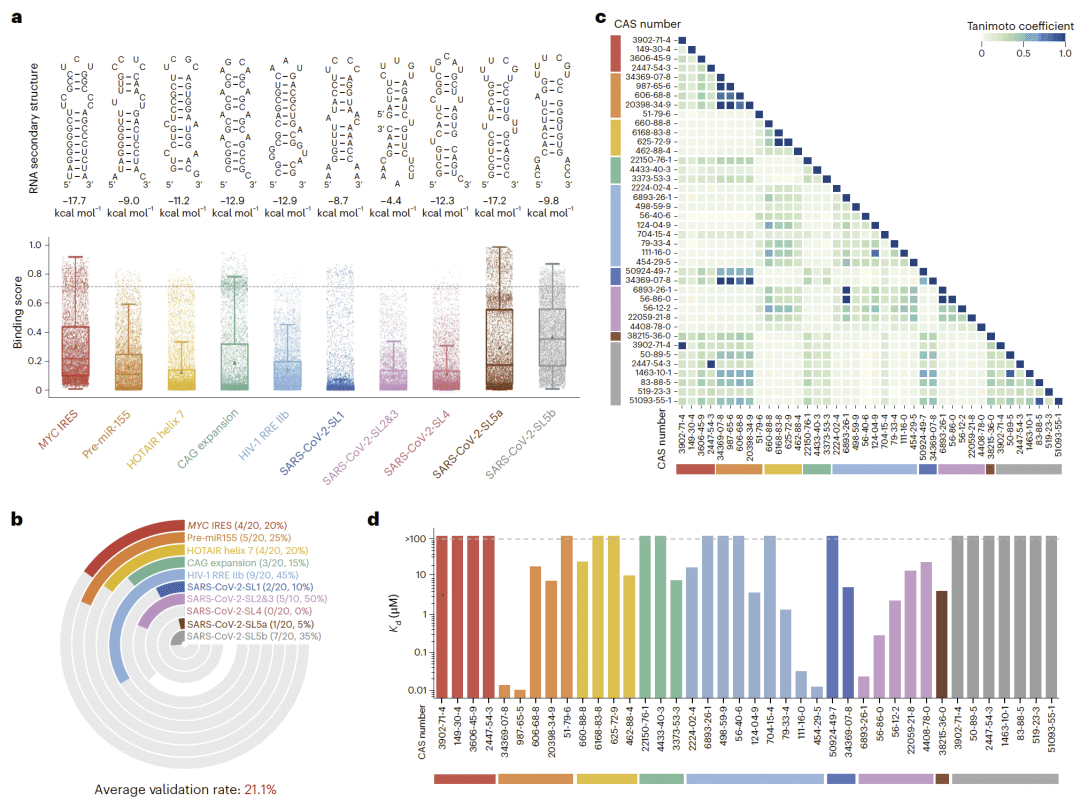
图 3|疾病相关 RNA 靶点的小分子实验验证结果。
MYC IRES 的系统验证
在 MYC IRES 案例中,预测结合评分与实验验证率呈明显正相关,表明 SMRTnet 的评分可用于有效排序真实结合分子。进一步分析显示,预测结合位点与突变实验结果高度一致。
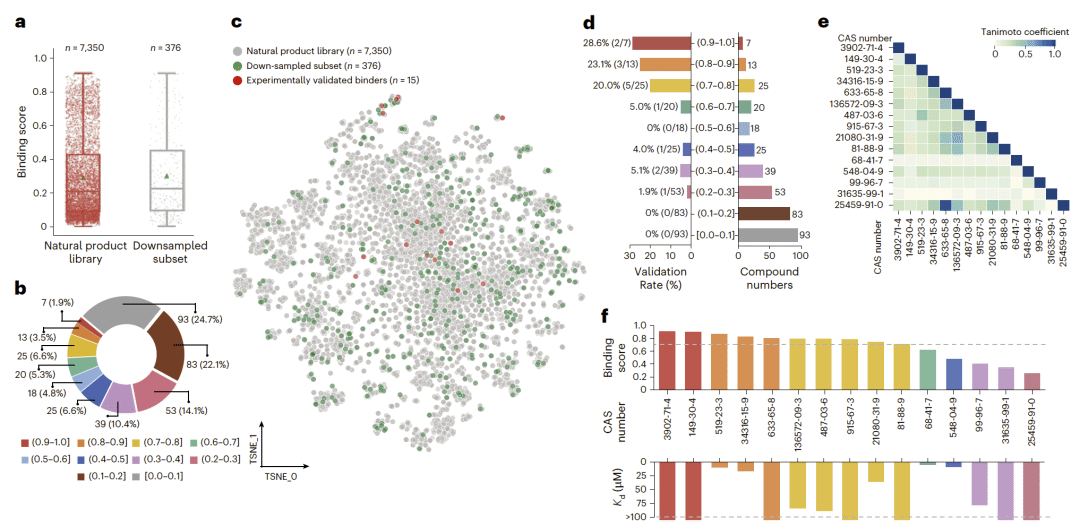
图 4|不同预测区间内 MYC IRES 靶向小分子的实验验证结果。
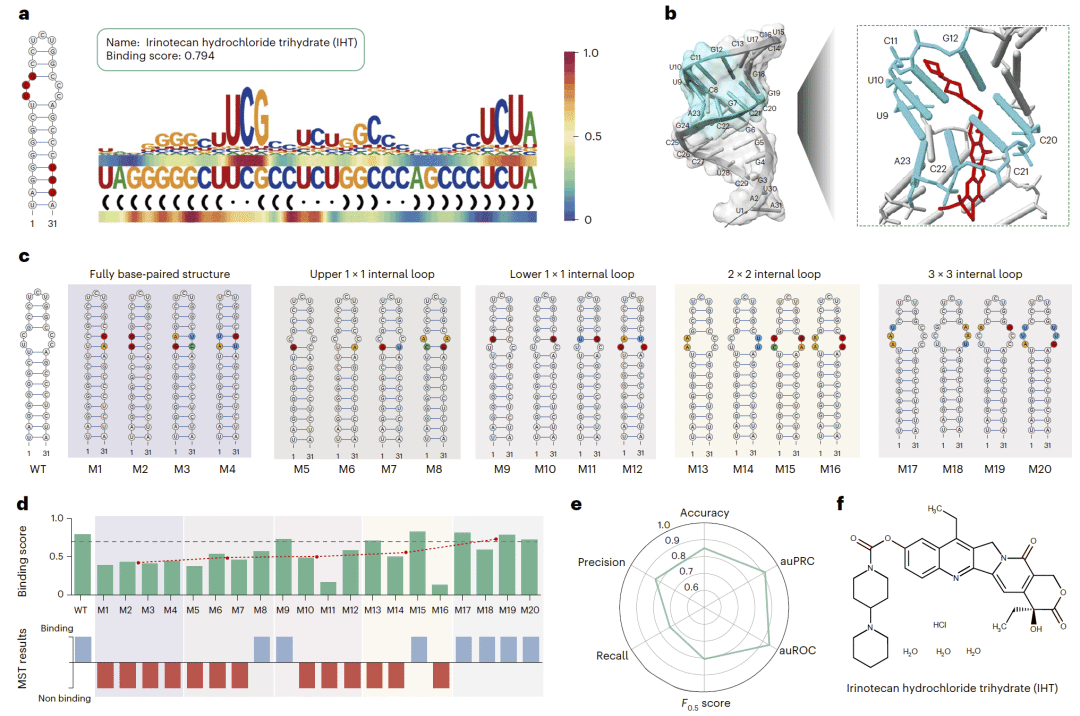
图 5|MYC IRES 上预测结合位点的实验验证。
MYC IRES 靶向小分子的功能验证
在 MYC 内部核糖体进入位点(IRES)研究中,SMRTnet 预测的小分子与实验验证结果高度一致。其中一个候选化合物能够显著下调 MYC 表达、抑制癌细胞增殖并促进细胞凋亡,显示出潜在的治疗价值。
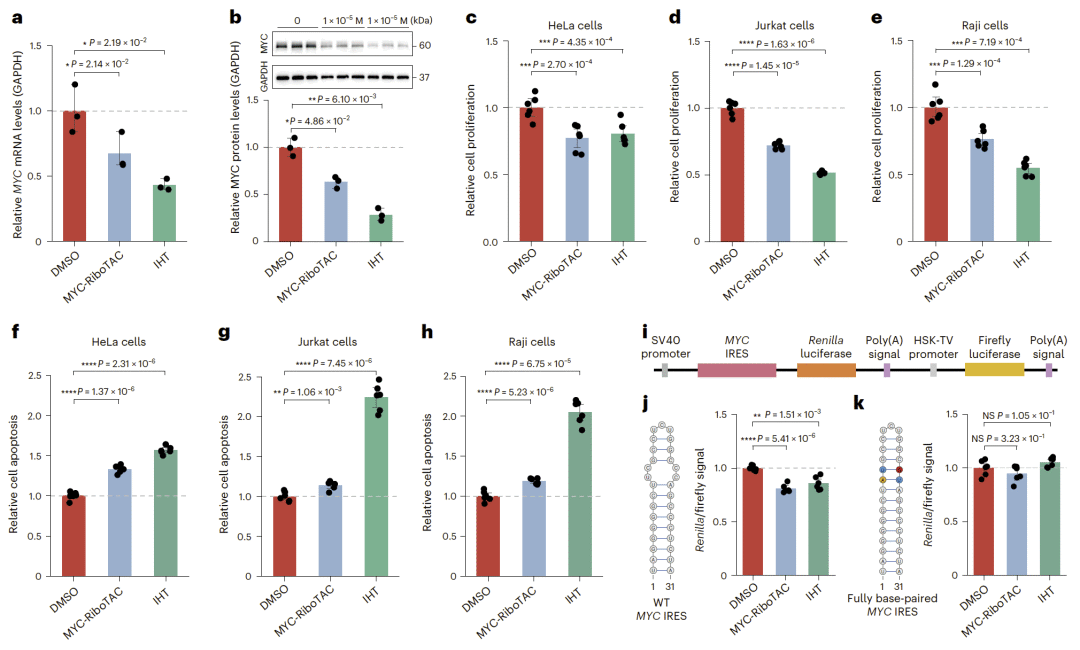
图 6|MYC IRES 靶向化合物 IHT 抑制 MYC 表达与细胞增殖。
讨论
SMRTnet 提供了一种不依赖 RNA 三级结构的小分子–RNA 相互作用预测新范式,显著扩展了可计算研究的 RNA 靶点空间。研究结果表明,RNA 二级结构结合序列信息已足以支持高质量的小分子识别,这为 RNA 靶向药物的早期发现提供了切实可行的计算工具。
该研究不仅展示了人工智能方法在 RNA 药物发现中的潜力,也为未来整合更多 RNA 动态结构信息和细胞环境因素奠定了基础。总体而言,SMRTnet 有望加速 RNA 靶向小分子药物的发现进程。
整理 | DrugOne团队
参考资料
Fei, Y., Wang, P., Zhang, J. et al. Predicting small molecule–RNA interactions without RNA tertiary structures. Nat Biotechnol (2026).
https://doi.org/10.1038/s41587-025-02942-z
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-03,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

