AI+Drug 文献速递 | Token-Mol 1.0,首个仅用标记的3D药物设计模型,为 AI 驱动药物发现提供新方向
AI+Drug 文献速递 | Token-Mol 1.0,首个仅用标记的3D药物设计模型,为 AI 驱动药物发现提供新方向

MindDance
发布于 2026-01-08 12:47:20
发布于 2026-01-08 12:47:20
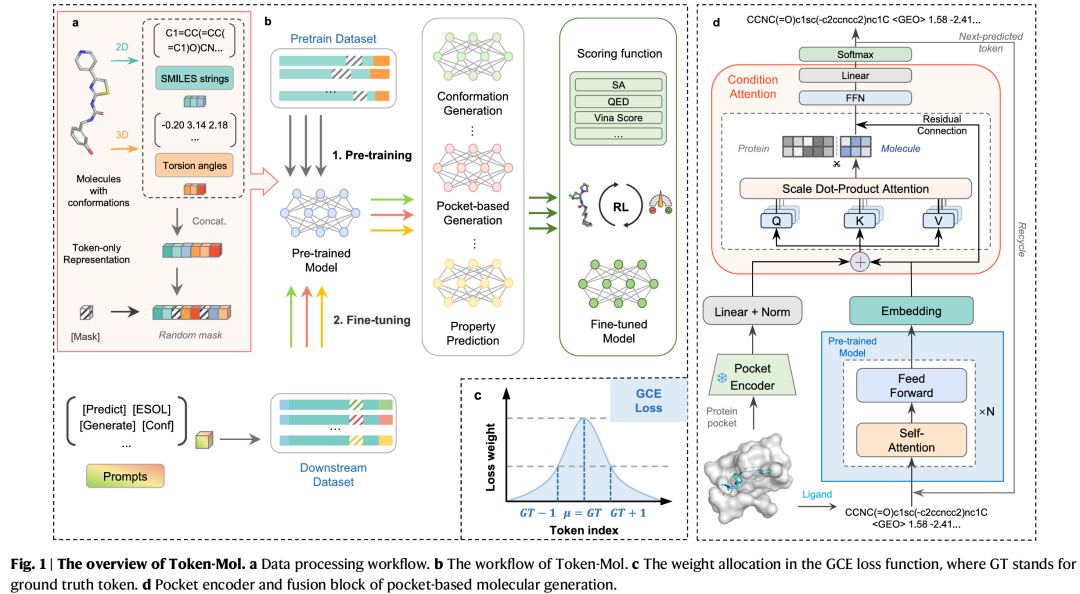
1. Token-Mol 1.0: tokenized drug design with large language models
期刊: Nature Communications 链接: https://doi.org/10.1038/s41467-025-59628-y 代码: https://github.com/jkwang93/Token-Mol https://doi.org/10.5281/zenodo.1511068
简介: 本文提出Token-Mol 1.0,这是首个仅使用标记的3D药物设计模型,通过将2D和3D结构信息及分子性质编码为离散标记,解决了现有方法难以有效整合三维分子结构的问题。模型基于Transformer解码器,采用因果掩码训练和高斯交叉熵损失函数,适用于回归任务。实验在分子构象生成、性质预测和基于口袋的分子生成等任务上进行,使用GEOM数据集、PDBBind相关数据集等,结果显示其在分子构象生成上优于现有方法,性能提升超10%和20%,且生成速度比专家扩散模型快35倍。研究表明,Token-Mol在真实药物设计场景中能提高成功率,结合强化学习可进一步优化亲和力和类药性,为AI驱动药物发现提供了新方向。
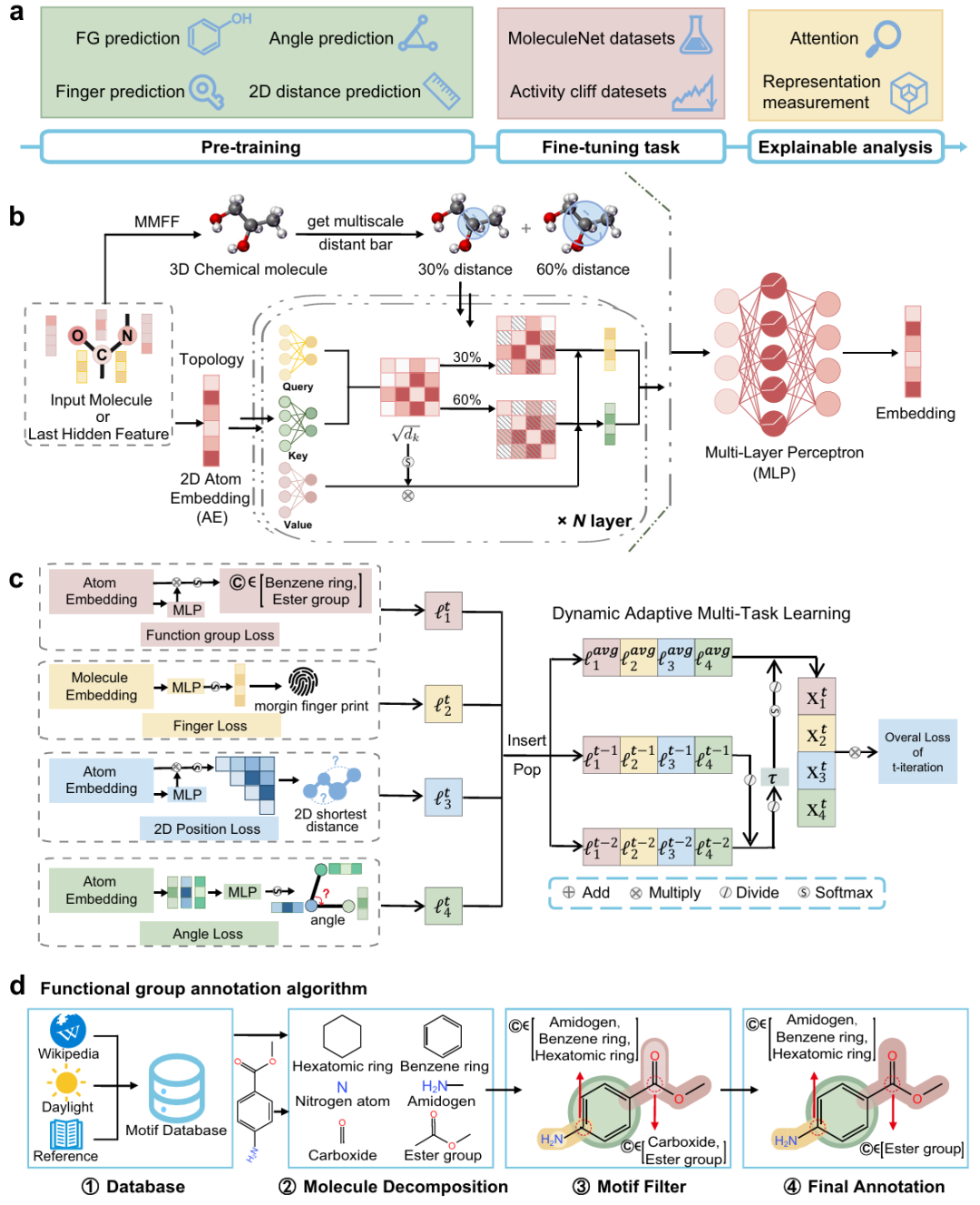
2. A self-conformation-aware pre-training framework for molecular property prediction with substructure interpretability
期刊: Nature Communications 链接: https://doi.org/10.1038/s41467-025-59628-y 代码: https://github.com/KazeDog/scage https://zenodo.org/records/15202798
简介: 本文提出自构象感知图Transformer框架SCAGE,通过多任务预训练(分子指纹预测、官能团预测、2D原子距离预测和3D键角预测)和数据驱动的多尺度构象学习策略,提升分子性质预测的泛化能力和子结构可解释性。模型在9个分子性质和30个活性悬崖基准上表现优异,使用约500万类药物化合物数据集 pretrain,在PDBBind等数据集上的AUC-ROC和RMSE等指标优于GROVER、Uni-Mol等现有方法。消融实验验证了多尺度构象学习模块和官能团注释算法的有效性,案例研究显示其能准确捕捉与分子活性相关的官能团,为定量构效关系分析提供了新工具。
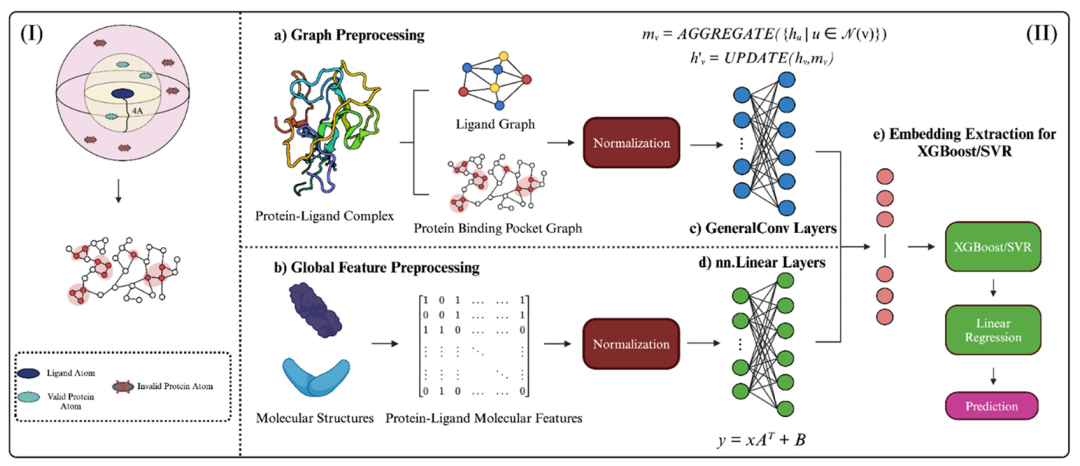
3. StructureNet: Physics-Informed Hybridized Deep Learning Framework for Protein–Ligand Binding Affinity Prediction
期刊: Bioengineering 链接: https://doi.org/10.3390/bioengineering12050505 代码: https://github.com/sivaGU/StructureNet
简介: 本文开发StructureNet,这是一种基于图神经网络的混合深度学习模型,仅使用蛋白质-配体结构描述符预测结合亲和力,避免依赖序列和相互作用数据导致的记忆化问题。模型将蛋白质和配体结构表示为图,结合几何描述符(如Voronoi镶嵌和球谐函数)和传统特征,采用GNN与XGBoost、SVR集成的架构。在PDBBind v2020数据集上,其PCC达0.68,AUC为0.75,优于MPNN(PL)等结构基模型;外部验证显示其能有效区分DUDE-Z数据集中的活性和诱饵配体。消融实验表明几何描述符是性能关键,结合分子动力学模拟生成的构象 ensemble 可进一步提升准确性,为基于结构的药物设计提供了新框架。
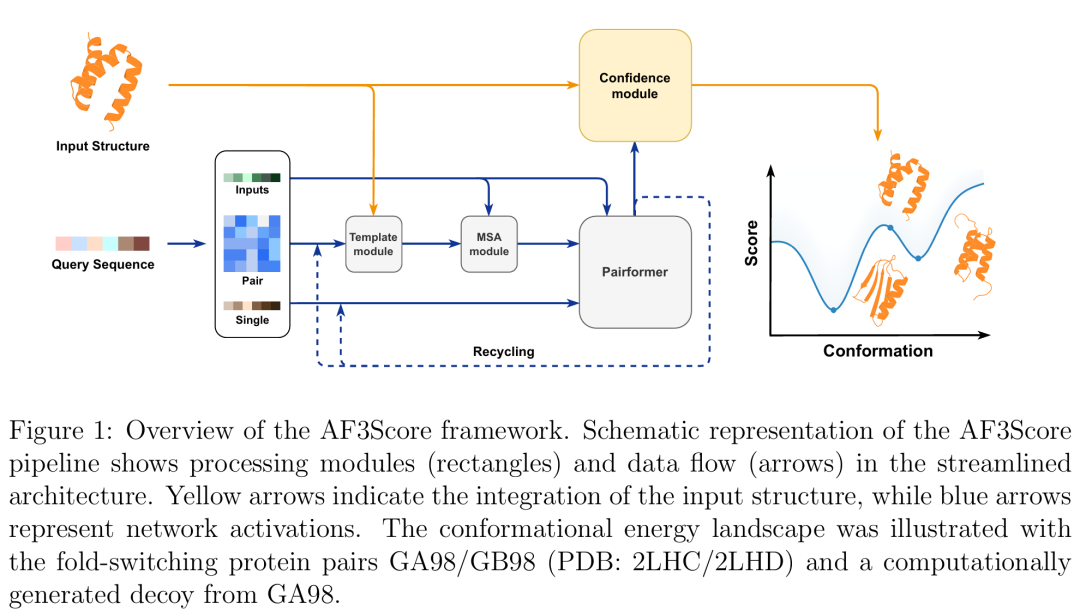
4. AF3Score: A Score-Only Adaptation of AlphaFold3 for Biomolecular Structure Evaluation
期刊: bioRxiv 链接: https://doi.org/10.1101/2025.05.10.653251 代码: https://github.com/Mingchenchen/AF3Score/
简介: 本文开发了AF3Score,这是一种仅评分的AlphaFold3改编模型,通过直接输入结构坐标到置信头而绕过扩散模块,解决了现有AlphaFold2方法需迭代优化输入结构导致评分有偏的问题。模型在单体蛋白、蛋白质-蛋白质复合物、从头设计结合剂和折叠转换蛋白等多样系统的结构质量评估中表现稳健。在设计结合剂筛选基准中,AF3Score在10个靶点中有8个优于现有方法,与AlphaFold2衍生方法结合时,实验验证结合剂的成功率从15.2%提升至31.6%,且能有效识别折叠转换蛋白的稳定构象,而AlphaFold主要预测主导构象。研究表明,AF3Score在从头结合剂设计的高通量筛选、对接生成构象和分子动力学轨迹过滤中具有广泛适用性,为生物分子结构评估提供了新工具。
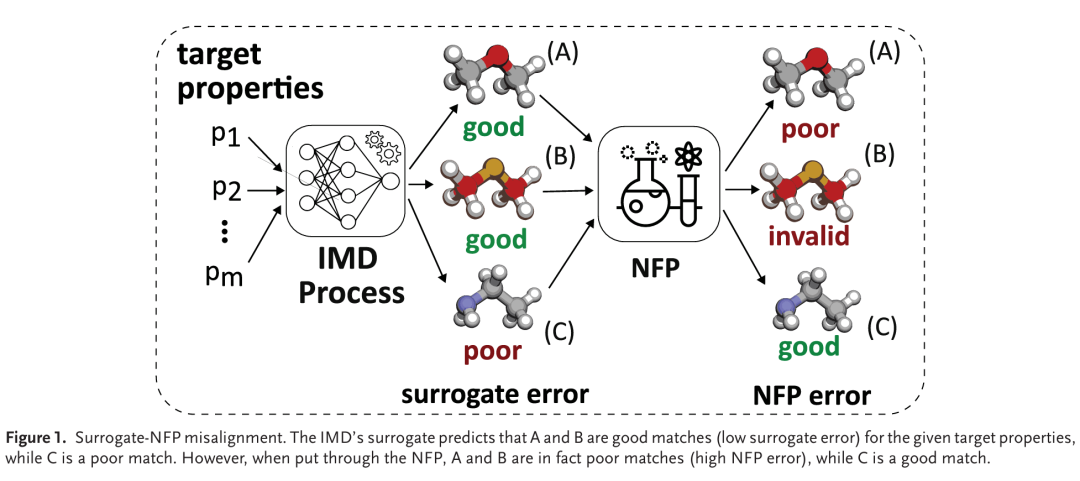
5. Trustworthy Inverse Molecular Design via Alignment with Molecular Dynamics
期刊: Advanced Science 链接: https://doi.org/10.1002/advs.202416356 代码: https://github.com/ktirta/TrustMol
简介: 本文提出TrustMol,一种通过与分子动力学基准对齐实现可信逆分子设计的方法,通过构建新型变分自动编码器(SGP-VAE)生成分子潜在空间,并训练集成属性预测器学习从潜在空间到属性空间的映射,结合重新获取方法确保训练样本的代表性,在优化时最小化预测误差和不确定性。在单目标和多目标逆分子设计任务中,TrustMol的NFP误差和NFP-代理模型偏差均优于现有基线,如在HOMO、LUMO和偶极矩预测中NFP误差最低,且在分子动力学模拟生成的构象集合中表现稳定。研究表明,TrustMol在保证设计准确性的同时提升了可信度,为逆分子设计提供了可靠框架。
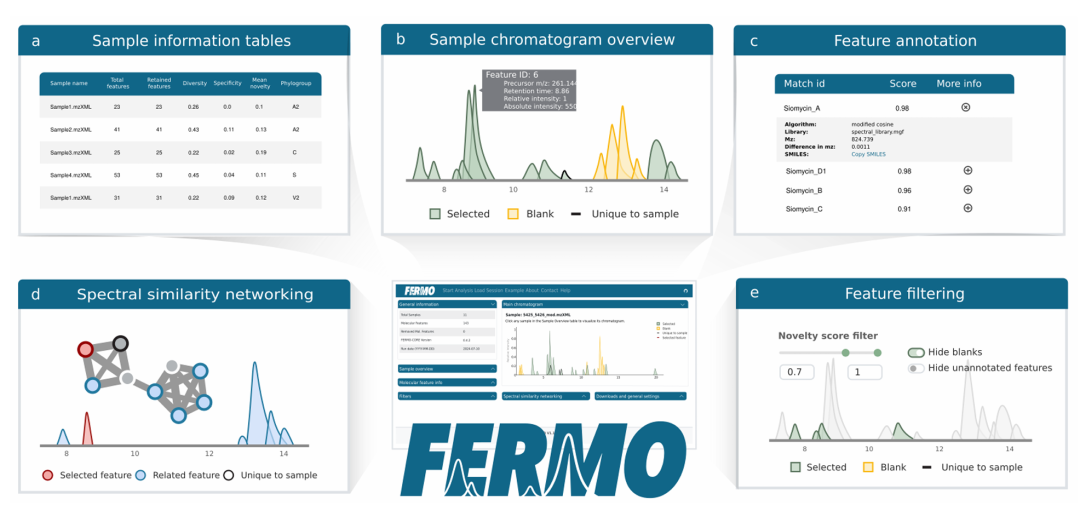
6. FERMO: a Dashboard for Automated Prioritization of Molecular Features from Mass Spectral Data
期刊: bioRxiv 链接: https://doi.org/10.1101/2022.12.21.521422 代码: https://github.com/fermo-metabolomics/fermo
简介: 本文介绍FERMO,一个基于质谱数据自动优先化分子特征的免费在线仪表盘工具,其模块化框架支持代谢组学数据与表型、基因组和样本元数据的集成,通过交互式可视化和可重复的优先化流程,实现特征和样本的筛选。在两项基准研究中,FERMO成功重现了已知生物活性化合物的优先化结果,如在大戟属植物提取物中准确识别抗病毒活性特征,在链霉菌OSMAC研究中定位放线菌素及其类似物,且优先化的特征仅占数据集的0.7%。FERMO通过标准化输入输出和参数管理,提升了假设驱动的代谢组学分析效率,适用于天然产物研究、环境科学和微生物组学等领域,但目前仅支持特定格式的LC-MS/MS数据。
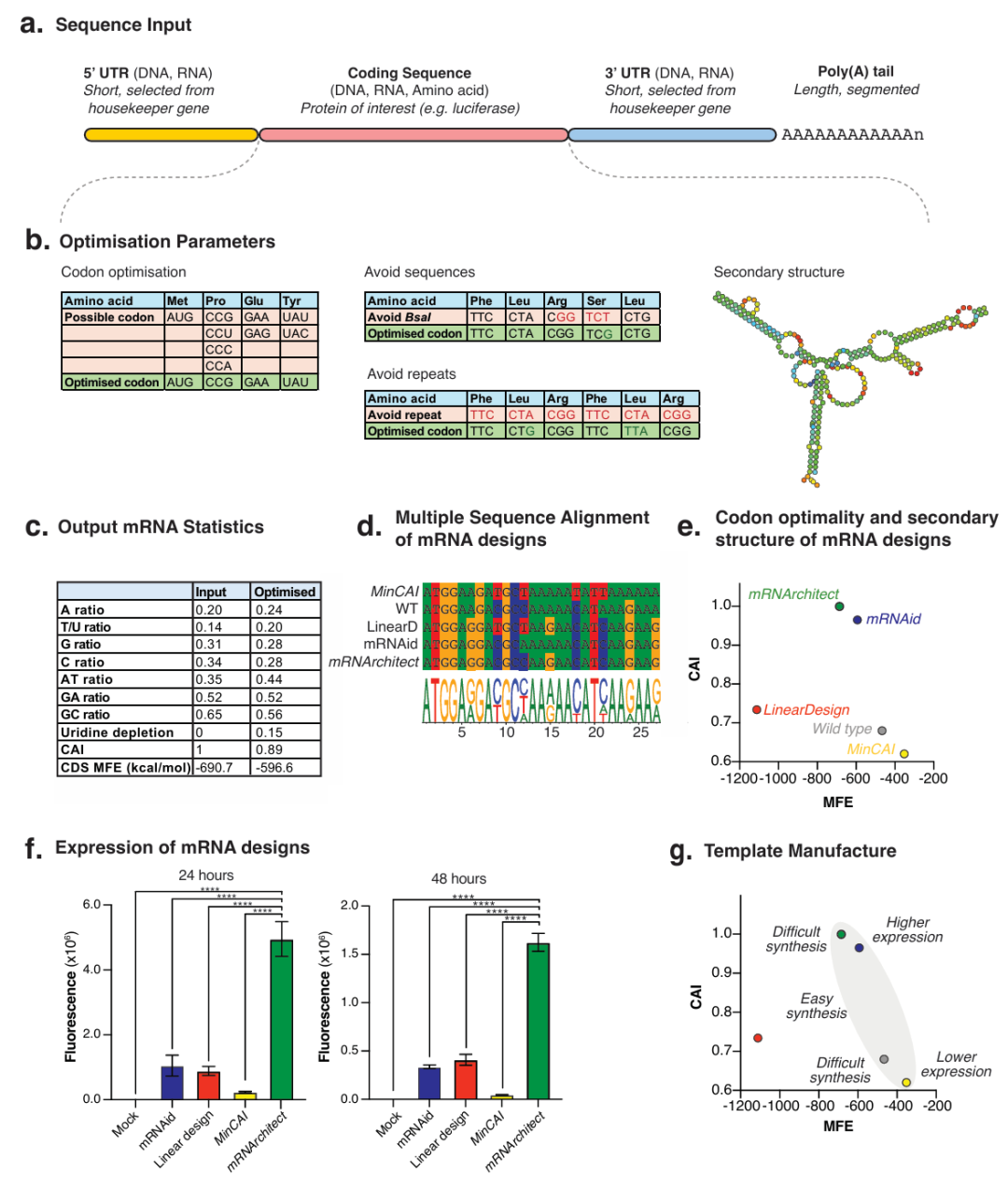
7. mRNArchitect: sequence design of mRNA medicines
期刊: bioRxiv 链接: https://doi.org/10.1101/2024.12.03.626696 代码: https://github.com/BaseUQ/mRNArchitect
简介: 本文开发了mRNArchitect软件工具包,用于辅助mRNA序列设计,可根据用户需求快速组装和优化mRNA序列,涵盖GC含量、二级结构、密码子优化和尿苷耗竭等参数,输出可直接合成且经实验验证的序列。在荧光素酶和eGFP的mRNA设计中,mRNArchitect生成的高密码子适应指数(CAI)序列在HEK293细胞中表达水平显著高于其他工具设计的序列,如最高CAI设计的荧光素酶mRNA表达量是最低CAI设计的21倍(24小时)。研究表明,mRNArchitect通过平衡序列优化参数和可制造性,为不同应用场景提供了灵活的mRNA设计方案,有助于推动mRNA疫苗和疗法的开发。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-05-17,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

