Nat. Commun. | EviDTI:基于证据深度学习的药物 - 靶点相互作用预测
Nat. Commun. | EviDTI:基于证据深度学习的药物 - 靶点相互作用预测
用户1151118
发布于 2026-01-08 13:04:19
发布于 2026-01-08 13:04:19
Evidential Deep Learning-Based Drug-Target Interaction Prediction
期刊: Nature Communications 链接: https://doi.org/10.1038/s41467-025-62235-6 代码: https://github.com/zhaoyanpeng208/EviDTI 简介: 该论文提出了一种基于证据深度学习(EDL)的药物 - 靶点相互作用(DTI)预测框架 EviDTI,其创新点在于通过 EDL 实现了预测的不确定性量化,解决了传统深度学习模型在 DTI 预测中存在的过度自信和错误预测问题。EviDTI 整合了药物的 2D 拓扑图、3D 空间结构以及靶点序列特征,利用蛋白质预训练模型 ProtTrans 和药物预训练模型 MG-BERT 等提取特征,并通过证据层输出预测概率和不确定性。实验在 DrugBank、Davis 和 KIBA 三个基准数据集上进行,与 11 个基线模型相比,EviDTI 表现出优异的预测性能,且其不确定性估计能有效校准预测错误,在冷启动场景和酪氨酸激酶调节剂的案例研究中也验证了其有效性。该论文表明,基于证据深度学习的 EviDTI 为 DTI 预测提供了可靠的不确定性量化工具,有助于加速药物发现进程。

药物-靶点相互作用(DTI)预测作为药物发现流程中的关键环节,其准确性与可靠性直接影响新药研发的效率与成本。尽管深度学习方法在该领域已展现出显著潜力,但传统模型普遍存在预测置信度不可靠、对新型未见过的DTI鲁棒性不足以及过度自信型错误预测等核心问题。近期发表于《Nature Communications》的研究论文《Evidential deep learning-based drug-target interaction prediction》提出的EviDTI框架,创新性地将证据深度学习(EDL)引入DTI预测任务,为解决上述挑战提供了突破性方案。
研究背景与核心挑战
药物发现是一个涉及靶点识别、化合物筛选、先导化合物优化等多步骤的复杂过程,其中DTI识别在化合物筛选阶段起着决定性作用。传统的体外实验虽能提供可靠的DTI验证结果,但高昂的成本与漫长的周期严重制约了药物研发的速度。为此,基于计算机模拟的DTI预测方法,尤其是深度学习技术,因其在降低成本、缩短周期和提高新药成功率方面的潜力而备受关注。
现有深度学习方法可大致分为基于网络的方法和蛋白质化学计量学(PCM)方法。前者通过整合药物-靶点、药物-药物、蛋白质-蛋白质等多种相互作用网络进行预测,后者则依赖药物与蛋白质的表征质量来提升预测精度,常用的模型包括卷积神经网络(CNNs)、循环神经网络(RNNs)、图神经网络(GNNs)和Transformer模型等。尽管这些方法在表征学习与模型架构上不断创新,如采用门控交叉注意力机制增强可解释性、利用预训练模型解决数据集规模小的问题、融合多模态数据提升表征全面性,但始终未能有效解决一个关键瓶颈:高预测概率与高置信度之间的不匹配。
传统深度学习模型缺乏对自身预测不确定性的量化能力,即使在面对分布外样本或噪声数据时,仍可能输出高概率的错误预测。这种“过度自信”现象会导致假阳性结果进入实验验证流程、在虚拟筛选中遗漏潜在活性化合物,甚至可能基于错误预测设计临床试验方案,不仅造成资源浪费,更可能延缓药物研发进程。
EviDTI框架的设计与创新
EviDTI框架的核心在于将证据深度学习(EDL)与多维度特征融合相结合,实现DTI预测的同时提供可靠的不确定性估计。与贝叶斯神经网络等传统不确定性量化方法相比,EDL无需依赖多次随机采样来近似潜在的不确定性函数,可直接学习不确定性,且能轻松整合到现有网络结构中,显著降低了计算成本与运行时间,更适用于大规模DTI预测任务。
该框架由三个关键组件构成:蛋白质特征编码器、药物特征编码器和证据层。蛋白质特征编码器采用预训练的蛋白质语言模型ProtTrans作为初始编码器,提取蛋白质序列的1024维残基嵌入特征,随后通过轻量注意力(LA)模块进一步优化特征,捕捉残基水平的局部相互作用。药物特征编码器则同时处理2D拓扑信息与3D结构信息:2D拓扑图通过预训练模型MG-BERT获取初始表征后,经1DCNN处理得到最终2D特征;3D空间结构转化为原子-键图和键-角图,通过GeoGNN模块进行编码。最后,蛋白质与药物的表征被拼接后输入证据层,该层通过全连接神经网络输出Dirichlet分布参数α,进而计算得到DTI预测概率与相应的不确定性值。
这种多维度特征融合策略与预训练模型的引入,显著提升了模型对药物与蛋白质的表征能力。消融实验表明,融合药物2D拓扑、3D结构与蛋白质序列的EviDTI模型,在所有评估指标上均优于仅使用单一维度特征的模型;而使用ProtTrans和MG-BERT等预训练模型作为初始特征提取器的架构,其性能也全面优于采用整数编码、GCN等替代方案的模型。
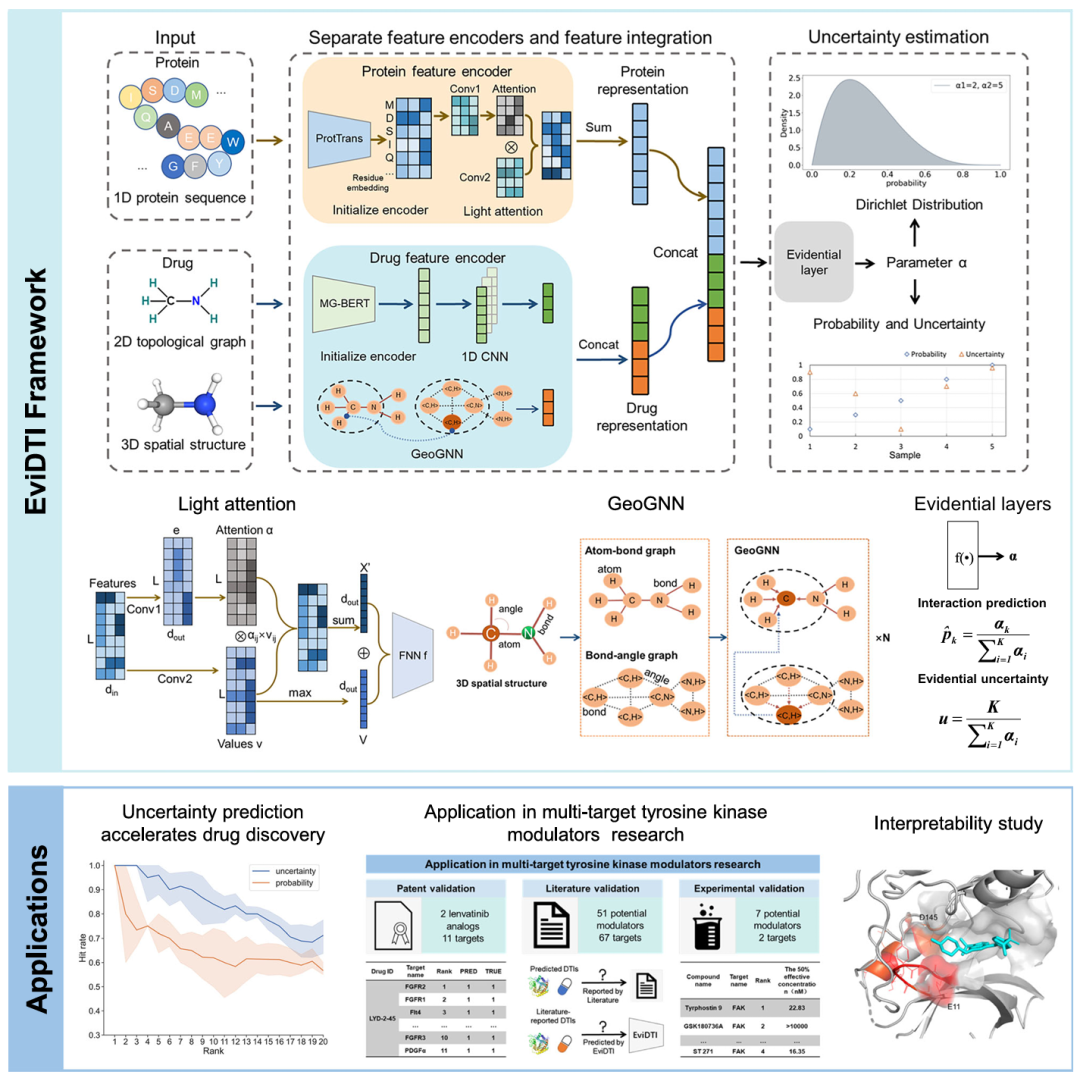
模型的流程图。该图展示了 EviDTI 框架的整体结构。对于给定的药物 - 靶点对,蛋白质特征编码器使用预训练的 ProtTrans 模型生成初始靶点表征,并通过轻量注意力(LA)模块进一步优化;药物特征编码器处理 2D 拓扑和 3D 结构表征,2D 表征来自预训练的 MG-BERT 模型并经 1D CNN 处理,3D 结构表征通过 GeoGNN 模块获得。这些表征被拼接后输入证据层,输出参数 α 以计算预测概率和不确定性。
模型的流程图。该图展示了 EviDTI 框架的整体结构。对于给定的药物 - 靶点对,蛋白质特征编码器使用预训练的 ProtTrans 模型生成初始靶点表征,并通过轻量注意力(LA)模块进一步优化;药物特征编码器处理 2D 拓扑和 3D 结构表征,2D 表征来自预训练的 MG-BERT 模型并经 1D CNN 处理,3D 结构表征通过 GeoGNN 模块获得。这些表征被拼接后输入证据层,输出参数 α 以计算预测概率和不确定性。
实验验证与性能分析
研究团队在DrugBank、Davis和KIBA三个基准数据集上对EviDTI进行了系统评估,并与3种传统机器学习方法(随机森林、支持向量机、朴素贝叶斯)和8种当前主流的DTI预测模型(如DeepConv-DTI、GraphDTA、MolTrans等)进行对比。评估指标包括准确率(ACC)、召回率、精确率、Matthews相关系数(MCC)、F1分数、ROC曲线下面积(AUC)和精确率-召回率曲线下面积(AUPR)。
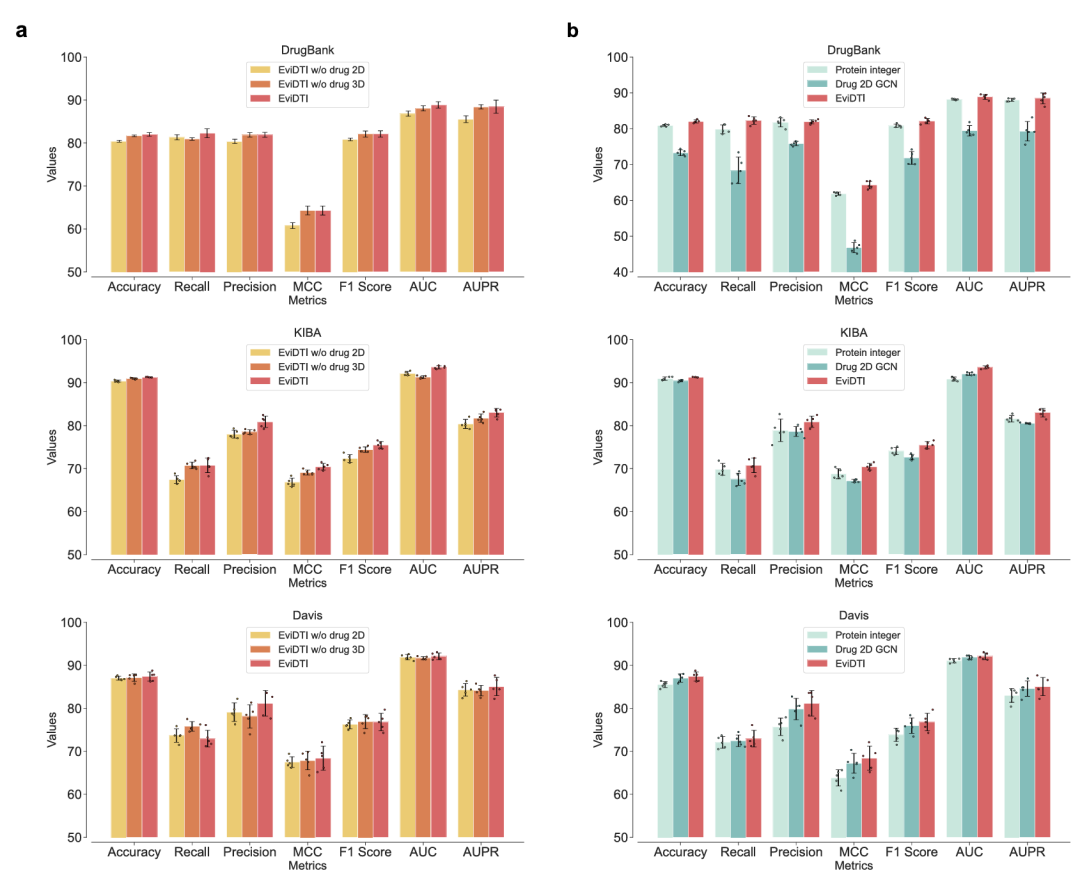
两项消融实验的结果。该图呈现了不同特征融合策略和是否使用预训练模型对预测性能的影响。(a)显示在 DrugBank、KIBA 和 Davis 数据集上,融合药物 2D 拓扑、3D 结构与蛋白质序列特征的 EviDTI 模型,在多数评估指标上优于仅使用单一维度特征的模型;(b)表明使用预训练模型进行初始特征提取的架构,在三个数据集的所有指标上均优于未使用预训练模型的架构,验证了多维度特征融合和预训练模型的价值。
两项消融实验的结果。该图呈现了不同特征融合策略和是否使用预训练模型对预测性能的影响。(a)显示在 DrugBank、KIBA 和 Davis 数据集上,融合药物 2D 拓扑、3D 结构与蛋白质序列特征的 EviDTI 模型,在多数评估指标上优于仅使用单一维度特征的模型;(b)表明使用预训练模型进行初始特征提取的架构,在三个数据集的所有指标上均优于未使用预训练模型的架构,验证了多维度特征融合和预训练模型的价值。
结果显示,EviDTI在三个数据集上均表现出卓越的综合性能。在DrugBank数据集上,其精确率达81.90%,准确率、MCC和F1分数分别为82.02%、64.29%和82.09%;在具有类别不平衡挑战的Davis和KIBA数据集上,EviDTI的优势更为明显,如在Davis数据集上,其准确率、F1分数和AUPR分别超过最佳基线模型0.8%、2%和0.3%。在冷启动场景下,EviDTI仍展现出强劲竞争力,准确率达79.96%,F1分数79.61%,仅AUC值略低于TransformerCPI。
更重要的是,EviDTI的不确定性量化能力得到了充分验证。通过分析预测结果与不确定性值的关系发现,在所有数据集上,错误预测样本(假阳性和假阴性)的不确定性显著高于正确预测样本;将样本按不确定性排序并划分置信区间后,不确定性最低的前5%样本准确率超过90%,而不确定性最高区间的准确率仅为0.5-0.6,表明模型的预测准确率随不确定性增加而显著下降,验证了不确定性估计的可靠性。
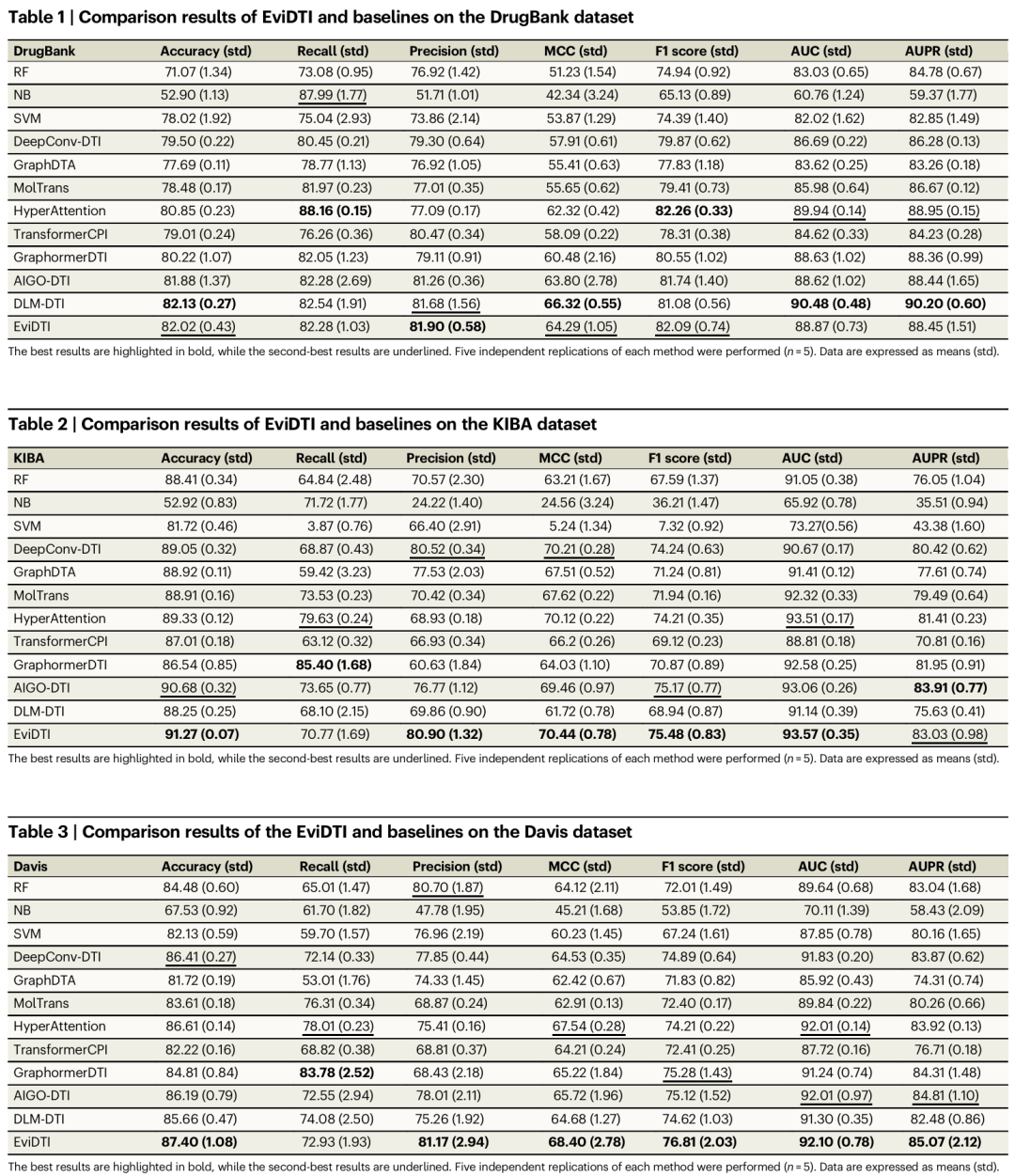
EviDTI 与基线模型在三个数据集上的比较结果
EviDTI 与基线模型在三个数据集上的比较结果
实际应用价值与案例研究
在实际药物研发场景中,EviDTI的不确定性信息展现出显著的实用价值。通过对比基于不确定性的预测方法与传统基于概率的方法发现,在高置信区间(阈值低于0.02),前者的折叠外率(OFR)更低,预测性能更优。在针对2022年FDA批准的新药物及其靶点的外部测试集中,基于不确定性的排序策略在Top3预测中命中率达100%,Top10预测中命中率超过80%,显著优于基于概率的排序策略,有效降低了假阳性率,提升了实验验证的效率。
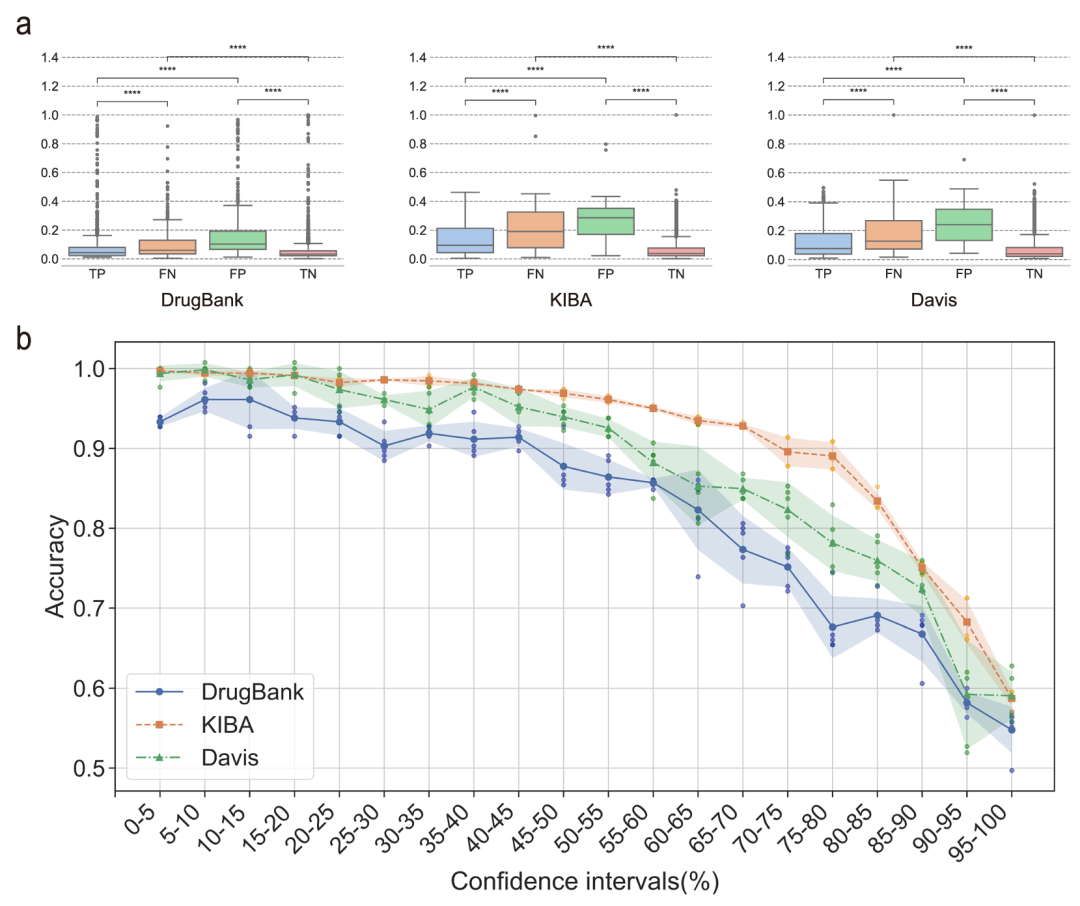
证据深度学习提供了良好的不确定性度量。该图验证了 EviDTI 的不确定性估计能力。(a)通过 Mann-Whitney 检验展示,在三个数据集上,错误预测样本(FP、FN)的不确定性显著高于正确预测样本(TP、TN);(b)显示将样本按不确定性分为 20 个置信区间后,不确定性最低的区间(前 5%)准确率超过 90%,而不确定性最高的区间准确率仅 0.5-0.6,表明预测准确率随不确定性增加而降低,验证了不确定性估计的可靠性。
证据深度学习提供了良好的不确定性度量。该图验证了 EviDTI 的不确定性估计能力。(a)通过 Mann-Whitney 检验展示,在三个数据集上,错误预测样本(FP、FN)的不确定性显著高于正确预测样本(TP、TN);(b)显示将样本按不确定性分为 20 个置信区间后,不确定性最低的区间(前 5%)准确率超过 90%,而不确定性最高的区间准确率仅 0.5-0.6,表明预测准确率随不确定性增加而降低,验证了不确定性估计的可靠性。
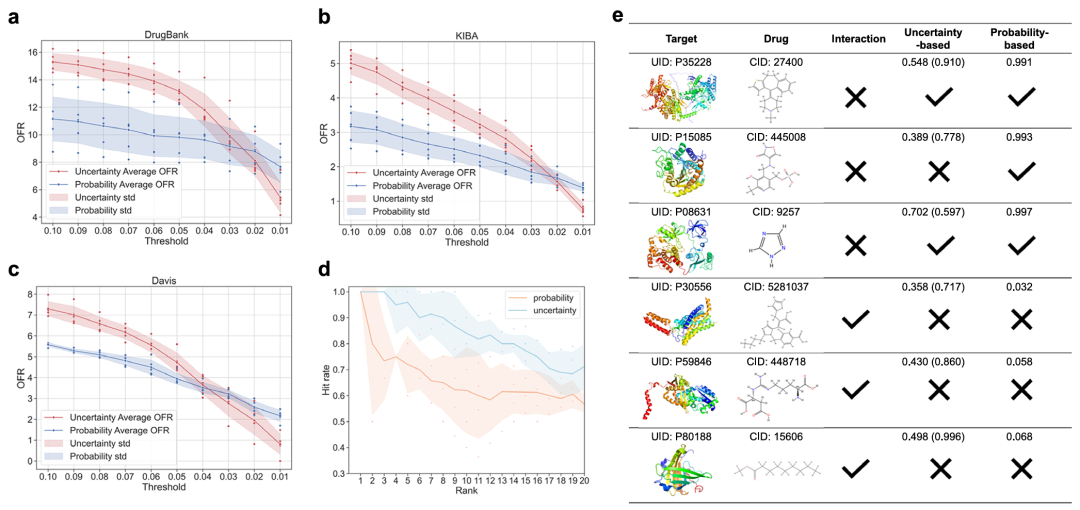
证据深度学习有助于降低决策中的错误预测风险。该图展示了不确定性预测在实际决策中的作用。(a-c)比较了基于不确定性和基于概率的框架在三个数据集上的折叠外率(OFR),表明在高置信区间(阈值 < 0.02),基于不确定性的方法 OFR 更低,性能更优;(d)显示在外部数据集上,基于不确定性的排序策略在 Top3 预测中命中率达 100%,Top10 预测中命中率超 80%,优于基于概率的排序;(e)通过案例说明不确定性方法能识别高概率错误预测,避免误导实验验证。
证据深度学习有助于降低决策中的错误预测风险。该图展示了不确定性预测在实际决策中的作用。(a-c)比较了基于不确定性和基于概率的框架在三个数据集上的折叠外率(OFR),表明在高置信区间(阈值 < 0.02),基于不确定性的方法 OFR 更低,性能更优;(d)显示在外部数据集上,基于不确定性的排序策略在 Top3 预测中命中率达 100%,Top10 预测中命中率超 80%,优于基于概率的排序;(e)通过案例说明不确定性方法能识别高概率错误预测,避免误导实验验证。
在酪氨酸激酶调节剂的研究中,EviDTI的应用价值得到进一步体现。酪氨酸激酶作为调节细胞信号传导的关键酶,在癌症治疗中具有重要作用,而多靶点酪氨酸激酶调节剂可同时调控多个癌症相关激酶,有效改善治疗效果并降低耐药性。通过专利数据与文献数据验证,EviDTI对已知DTI的预测准确率较高,如在22对来自专利的药物-靶点对中,正确预测16对;在27对文献报道的DTI中,成功预测21对,其中10对具有高置信度(不确定性分数低于0.1)。
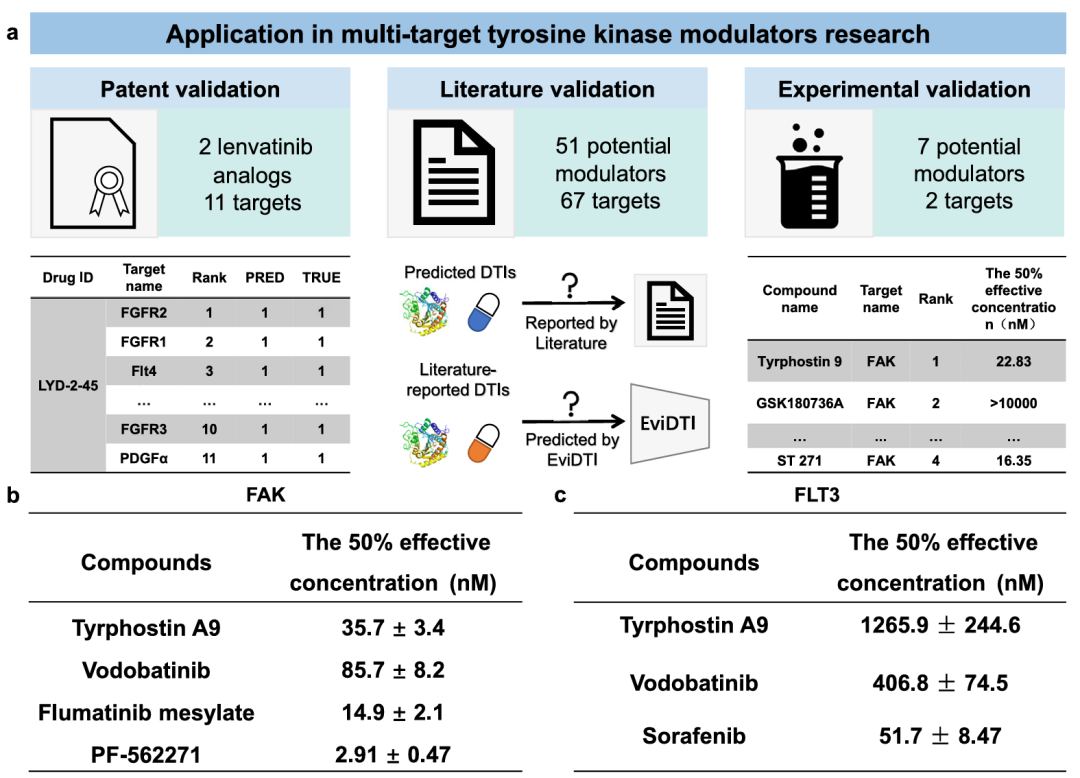
模型在多靶点酪氨酸激酶调节剂发现中的应用。该图展示了 EviDTI 在酪氨酸激酶调节剂研究中的验证与应用。(a)为验证框架,包括专利数据、文献数据验证及针对 FAK 和 FLT3 的实验验证;(b)显示 FAK 激酶实验中,Tyrphostin 9、Vodobatinib、甲磺酸氟马替尼和阳性对照 PF-562271 的半数有效浓度;(c)展示 FLT3 激酶实验中,Vodobatinib、Tyrphostin 9 和阳性对照索拉非尼的半数有效浓度,验证了 EviDTI 预测的新型调节剂的活性。
模型在多靶点酪氨酸激酶调节剂发现中的应用。该图展示了 EviDTI 在酪氨酸激酶调节剂研究中的验证与应用。(a)为验证框架,包括专利数据、文献数据验证及针对 FAK 和 FLT3 的实验验证;(b)显示 FAK 激酶实验中,Tyrphostin 9、Vodobatinib、甲磺酸氟马替尼和阳性对照 PF-562271 的半数有效浓度;(c)展示 FLT3 激酶实验中,Vodobatinib、Tyrphostin 9 和阳性对照索拉非尼的半数有效浓度,验证了 EviDTI 预测的新型调节剂的活性。
基于EviDTI的预测结果,研究团队对酪氨酸激酶FAK和FLT3的潜在调节剂进行了实验验证。ADP-Glo激酶实验表明,Tyrphostin 9、Vodobatinib和甲磺酸氟马替尼对FAK均有抑制活性,半数有效浓度分别为35.7±3.4 nM、85.7±8.2 nM和14.9±2.1 nM;Tyrphostin 9和Vodobatinib对FLT3则有激活作用,进一步证实了EviDTI在发现新型药物-靶点相互作用方面的潜力。
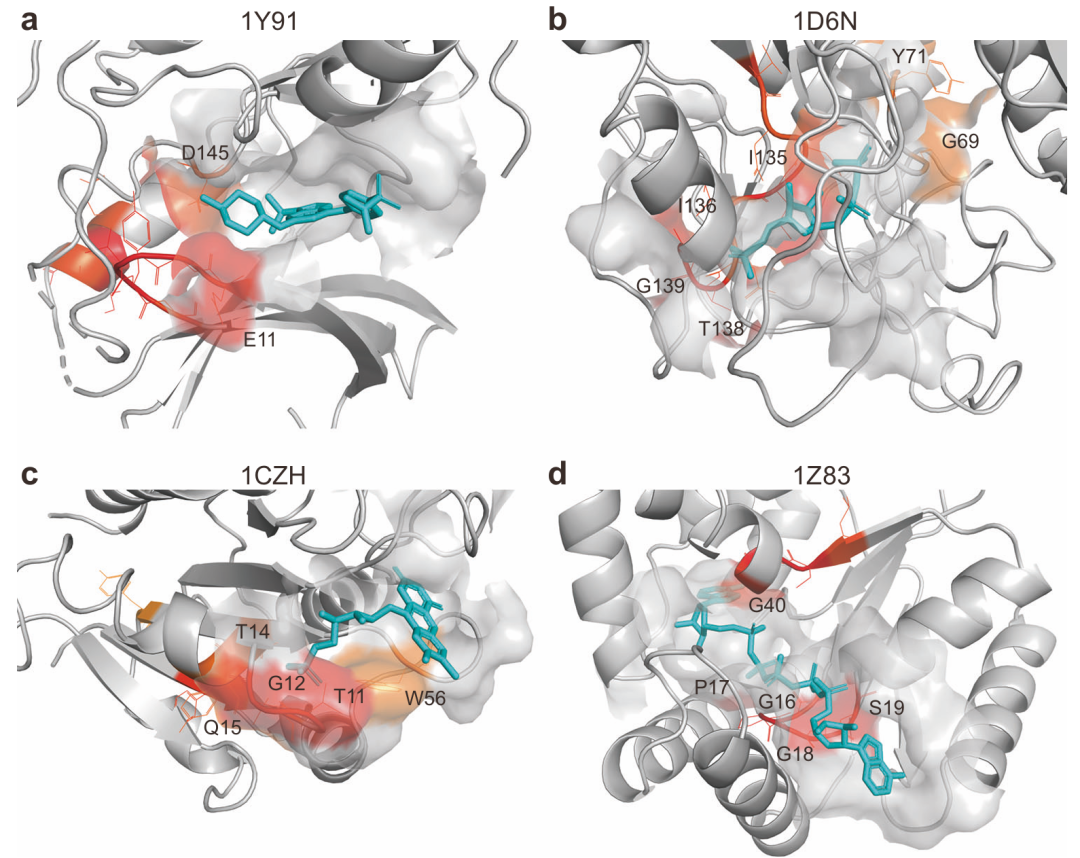
该图展示了注意力权重与结合残基的关系。从 DrugBank 数据集随机选取四个 DTI 对,通过 PyMOL 可视化其三维结构,高注意力值的残基以不同颜色标注。结果显示,所有案例中高注意力值的残基与结合位点一致,验证了轻量注意力机制在识别关键残基方面的有效性,增强了模型的可解释性。
该图展示了注意力权重与结合残基的关系。从 DrugBank 数据集随机选取四个 DTI 对,通过 PyMOL 可视化其三维结构,高注意力值的残基以不同颜色标注。结果显示,所有案例中高注意力值的残基与结合位点一致,验证了轻量注意力机制在识别关键残基方面的有效性,增强了模型的可解释性。
此外,EviDTI的轻量注意力模块还提供了残基水平的可解释性。通过将注意力权重映射到蛋白质-配体复合物的三维结构,发现高注意力值的残基与结合位点高度重合,结合位点命中率分析显示,超过70%的结合位点位于注意力值前50%的残基中,为药物设计提供了有价值的结构生物学 insights。
局限性与未来展望
尽管EviDTI展现出显著优势,但仍存在一定局限性。目前模型无法区分分子对靶点的激活或抑制作用,且对化合物与靶点及其突变体之间相互作用的识别能力有待提升。针对这些问题,未来研究可从多个方向展开:引入更多分子生化特性,开发多任务学习框架以同时预测激活与抑制作用;整合蛋白质3D结构信息与药理学扰动转录组数据,提升对突变体的表征能力;将交叉注意力机制与EDL相结合,增强模型对药物与蛋白质局部结构相互作用的学习能力;融合迁移学习以改善模型对分布外样本的泛化性能。
此外,EviDTI框架的通用性使其有望拓展至其他相互作用预测任务,如酶反应动力学参数预测和药物-药物相互作用预测等,为药物研发领域提供更广泛的支持。
综上所述,EviDTI通过创新性地融合证据深度学习与多维度特征表征,不仅实现了高精度的DTI预测,更提供了可靠的不确定性量化,有效解决了传统深度学习模型在该领域的核心局限。其在基准数据集与实际应用场景中的优异表现,充分证明了证据深度学习作为DTI预测中不确定性量化工具的巨大潜力,为加速药物发现进程、降低研发风险提供了强有力的技术支撑。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-05,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

