Chem. Sci. | PepINVENT:基于生成式 AI 的天然与非天然氨基酸肽设计新框架
Chem. Sci. | PepINVENT:基于生成式 AI 的天然与非天然氨基酸肽设计新框架
用户1151118
发布于 2026-01-08 13:30:12
发布于 2026-01-08 13:30:12
PepINVENT: generative peptide design beyond natural amino acids
期刊: Chemical Science 链接: https://doi.org/10.1039/d4sc07642g 代码: https://github.com/MolecularAI/PepINVENT/ 简介: 本文介绍了 PepINVENT,一种基于生成式 AI 的肽设计工具,其创新点在于突破天然氨基酸限制,能探索包含非天然氨基酸(NNAAs)的广阔化学空间。该工具基于 REINVENT 框架,采用 Transformer 模型,通过半合成肽数据预训练,结合强化学习实现目标导向设计。实验使用包含 100 万条肽的半合成数据集(含天然氨基酸及 10,000 种 NNAAs),验证了其生成肽的高有效性(>97%)、独特性及生成大量全新 NNAAs 的能力,且能通过强化学习精准调控肽拓扑结构和优化性质(如 Rev 结合肽的溶解度与渗透性)。该论文表明 PepINVENT 为肽类药物研发提供了强大工具,可应用于从头设计、先导优化等多个场景。

肽类分子在药物研发领域占据独特地位,其介于小分子与蛋白质之间的分子尺寸,赋予了它们高特异性、强亲和力及低毒性等优势,使其在靶向“不可成药”位点方面展现出巨大潜力。然而,传统肽设计方法受限于天然氨基酸库及预设序列空间,难以充分挖掘肽类药物的治疗潜力。前段时间,发表于《Chemical Science》的研究提出了一种名为PepINVENT的生成式AI工具,为突破这一局限提供了全新解决方案。
肽设计的挑战与PepINVENT的创新定位
天然肽由20种蛋白质ogenic氨基酸构成,其序列空间随长度呈指数级增长(20^L),但自然界中肽的翻译后修饰及微生物合成的非蛋白源氨基酸早已突破这一框架。非天然氨基酸(NNAAs)的引入能显著改善肽的代谢稳定性、结合亲和力及细胞渗透性,然而现有生成模型多局限于预设氨基酸库的序列级设计,无法实现真正的化学空间拓展。
PepINVENT作为小分子设计平台REINVENT的扩展,创新性地将生成式AI与强化学习结合,无需针对特定性质或拓扑结构训练,即可在天然与非天然氨基酸的广阔化学空间中导航。其核心优势在于:以氨基酸为基本单位学习肽的化学逻辑,保留肽结构的精细粒度;通过强化学习实现目标导向设计,支持多参数优化;能够生成全新NNAAs及多样化肽拓扑结构,为肽类药物研发提供了前所未有的设计自由度。
技术框架:从数据构建到模型训练
半合成训练数据的构建
鉴于天然肽及已知NNAAs数据的稀缺性,研究团队构建了包含100万条独特肽的半合成数据集。该数据集融合了20种天然氨基酸及来自Amarasinghe等人虚拟库的10,000种可合成NNAAs,涵盖线性、头-尾环化、侧链-尾环化及二硫键桥接等多种拓扑结构。数据生成过程中,肽链长度控制在6-18个氨基酸,NNAAs占比通过左偏正态分布控制在30%左右,同时引入立体异构(D型)及backbone N-甲基化等修饰,确保模型学习到丰富的化学多样性。
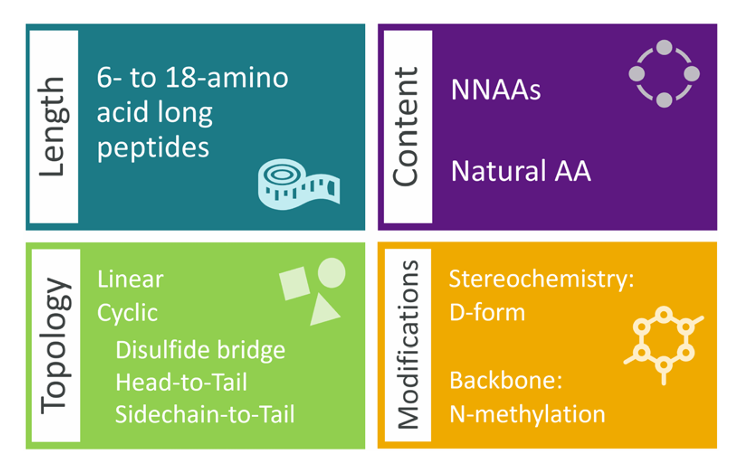
图 1:半合成数据的特征。该图展示了半合成数据集的主要特征,包括肽链长度(6 到 18 个氨基酸)、天然与非天然氨基酸的比例、肽的拓扑结构(线性、环化等)以及立体化学和骨架修饰(如 D 型异构体、骨架 N - 甲基化)。这些特征共同构成了多样化的训练数据,确保模型能学习到丰富的肽结构信息。
图 1:半合成数据的特征。该图展示了半合成数据集的主要特征,包括肽链长度(6 到 18 个氨基酸)、天然与非天然氨基酸的比例、肽的拓扑结构(线性、环化等)以及立体化学和骨架修饰(如 D 型异构体、骨架 N - 甲基化)。这些特征共同构成了多样化的训练数据,确保模型能学习到丰富的肽结构信息。
化学语言表示与预训练目标
PepINVENT采用CHUCKLES编码方式,将氨基酸以标准化SMILES格式表示,从氨基到羧基严格遵循N-to-C方向,使肽链的化学结构得以精确编码。预训练任务设计为“文本填充”:随机掩盖肽序列中30%的氨基酸(天然与非天然氨基酸按比例选择),模型需生成恰好数量的氨基酸填补空位,且生成产物必须符合化学规则。这种设计使模型能够理解肽的上下文逻辑,同时偏向于生成更多NNAAs以避免对天然氨基酸模式的过拟合。
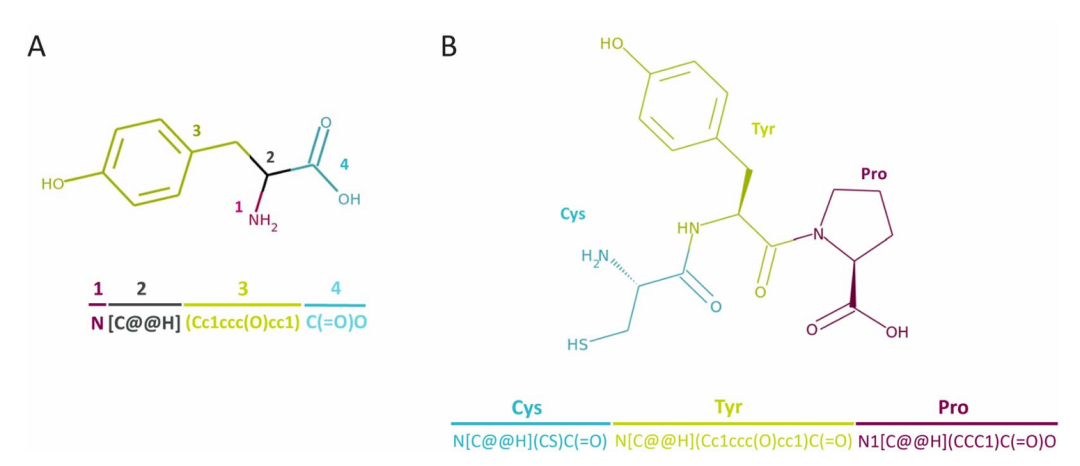
图 2:CHUCKLES 表示法示例。此图以酪氨酸(T)和三肽 CTP(半胱氨酸 - 酪氨酸 - 脯氨酸)为例,展示了 CHUCKLES 表示法。该方法按 N 到 C 的方向,以标准化 SMILES 格式编码氨基酸,包含氨基、α- 碳、侧链和剩余骨架结构,使肽的化学结构能被精准表示,且氨基酸的串联可形成完整肽的有效 SMILES 字符串。
图 2:CHUCKLES 表示法示例。此图以酪氨酸(T)和三肽 CTP(半胱氨酸 - 酪氨酸 - 脯氨酸)为例,展示了 CHUCKLES 表示法。该方法按 N 到 C 的方向,以标准化 SMILES 格式编码氨基酸,包含氨基、α- 碳、侧链和剩余骨架结构,使肽的化学结构能被精准表示,且氨基酸的串联可形成完整肽的有效 SMILES 字符串。
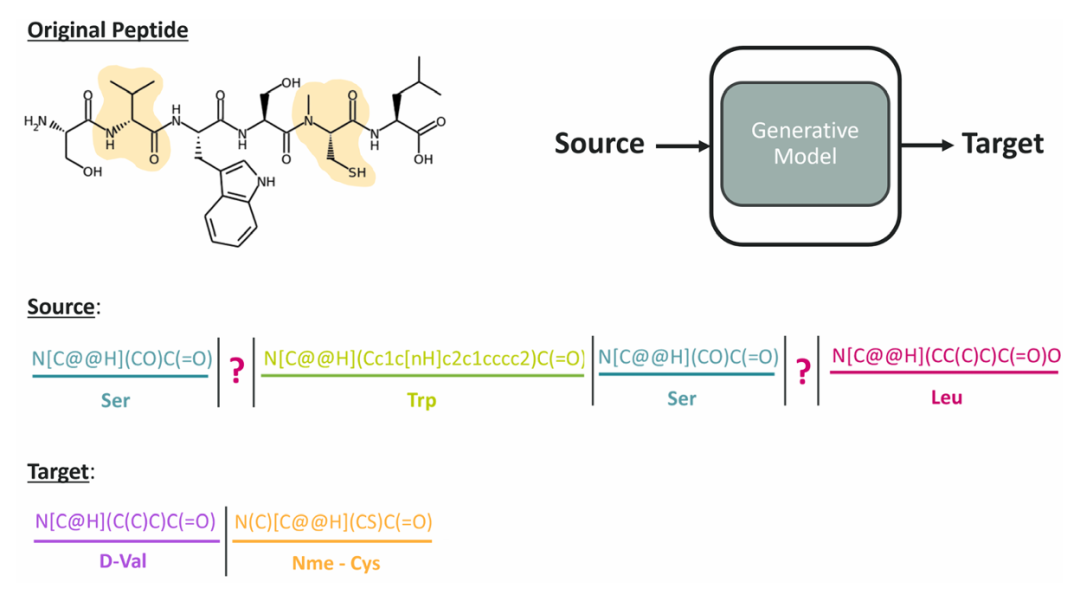
图 3:文本填充任务的源 - 目标对示例。图中展示了一个 6 肽的源 - 目标对,用于说明模型的预训练任务。源序列是掩盖了 2 号和 5 号位置氨基酸的肽,目标序列则是这两个位置对应的填充氨基酸(D - 缬氨酸和甲基化半胱氨酸)。模型通过学习此类对,掌握生成符合化学规则的氨基酸以填补肽中 masked 位置的能力,同时理解肽的上下文复杂性。
图 3:文本填充任务的源 - 目标对示例。图中展示了一个 6 肽的源 - 目标对,用于说明模型的预训练任务。源序列是掩盖了 2 号和 5 号位置氨基酸的肽,目标序列则是这两个位置对应的填充氨基酸(D - 缬氨酸和甲基化半胱氨酸)。模型通过学习此类对,掌握生成符合化学规则的氨基酸以填补肽中 masked 位置的能力,同时理解肽的上下文复杂性。
模型架构与训练策略
模型基于Transformer架构,包含编码器与解码器,参数设置与REINVENT保持一致。训练过程中,源序列(含掩码)与目标序列(填充氨基酸)经SMILES分词器处理后分别输入编码器与解码器,采用负对数似然损失函数优化参数。模型在NVIDIA V100显卡上训练24个epoch,批次大小为16,采用Adam优化器(学习率0.0001,4000步预热),确保在学习肽化学语言的同时避免过拟合。
性能评估:有效性、多样性与创新性的全面验证
肽水平的有效性与独特性
测试结果显示,PepINVENT生成肽的有效性表现优异:束搜索(beam search)方法的总体有效性达99%,多项式采样(multinomial sampling)达98%,其中线性肽与头-尾环化肽的有效性均超过98%。独特性方面,束搜索生成的肽序列独特性为100%,多项式采样约为98%,表明模型能够生成丰富多样的肽结构。值得注意的是,二硫键桥接肽在多项式采样中的独特性略低(94%),这与训练集中含硫侧链氨基酸数量有限相关,反映了模型对训练数据分布的合理响应。
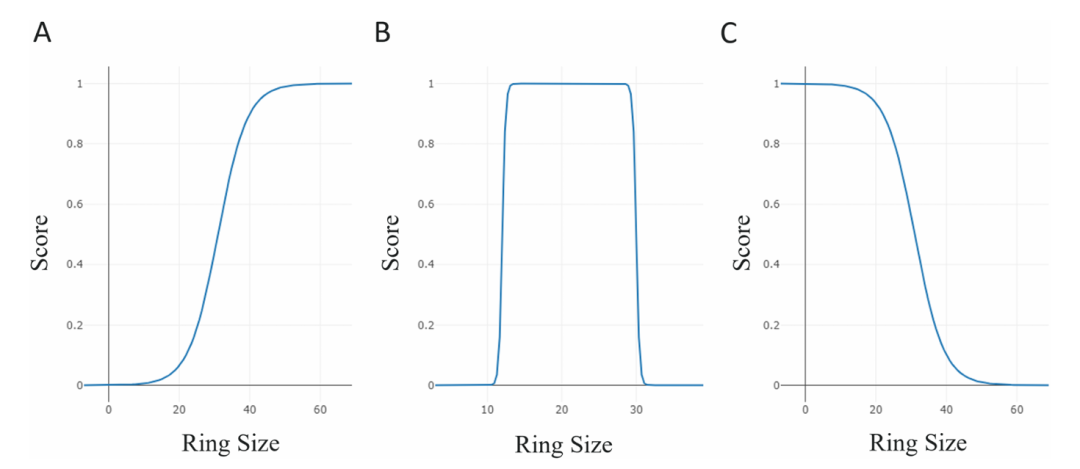
图 4:环大小评分函数的转换曲线。该图呈现了三种用于评分最大环尺寸的函数转换曲线:(A)sigmoid 函数,用于最大化环尺寸(12 到 60 个原子);(B)双 sigmoid 函数,用于限定头 - 尾环化的环尺寸范围;(C)反向 sigmoid 函数,用于最小化环尺寸以生成线性肽。这些曲线指导强化学习实现对肽拓扑结构的定向调控。
图 4:环大小评分函数的转换曲线。该图呈现了三种用于评分最大环尺寸的函数转换曲线:(A)sigmoid 函数,用于最大化环尺寸(12 到 60 个原子);(B)双 sigmoid 函数,用于限定头 - 尾环化的环尺寸范围;(C)反向 sigmoid 函数,用于最小化环尺寸以生成线性肽。这些曲线指导强化学习实现对肽拓扑结构的定向调控。
氨基酸水平的多样性与新颖性
在氨基酸层面,多项式采样生成的独特氨基酸数量(约1400种)显著多于束搜索(约200种),表明随机性采样策略更有利于化学空间探索。立体化学修饰对多样性贡献显著:去除手性信息后, canonical SMILES水平的独特性明显下降,证实了立体异构在拓展化学空间中的关键作用。
模型生成的氨基酸中,除天然氨基酸及训练集中的NNAAs外,还包含约91,826种全新NNAAs。t-SNE可视化显示,这些新颖氨基酸与训练集中的NNAAs在化学空间中分布相似,且在合成可及性、类天然产物性、重原子数量等分子特征上表现合理,证明其并非随机生成,而是基于学习到的化学规则产生。
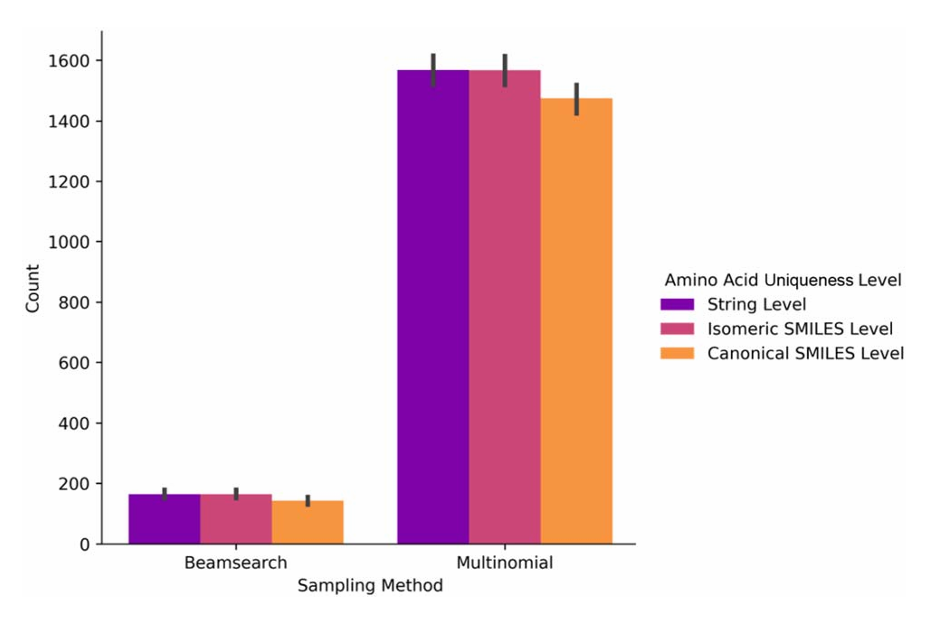
图 5:氨基酸的三种独特性水平对比。该图比较了束搜索和多项式采样在三种独特性水平(字符串水平、异构 SMILES 水平、规范 SMILES 水平)下生成的独特氨基酸数量。结果显示多项式采样生成的独特氨基酸显著更多,且去除立体化学信息后(规范 SMILES 水平),独特性下降,体现了立体修饰对多样性的贡献。
图 5:氨基酸的三种独特性水平对比。该图比较了束搜索和多项式采样在三种独特性水平(字符串水平、异构 SMILES 水平、规范 SMILES 水平)下生成的独特氨基酸数量。结果显示多项式采样生成的独特氨基酸显著更多,且去除立体化学信息后(规范 SMILES 水平),独特性下降,体现了立体修饰对多样性的贡献。
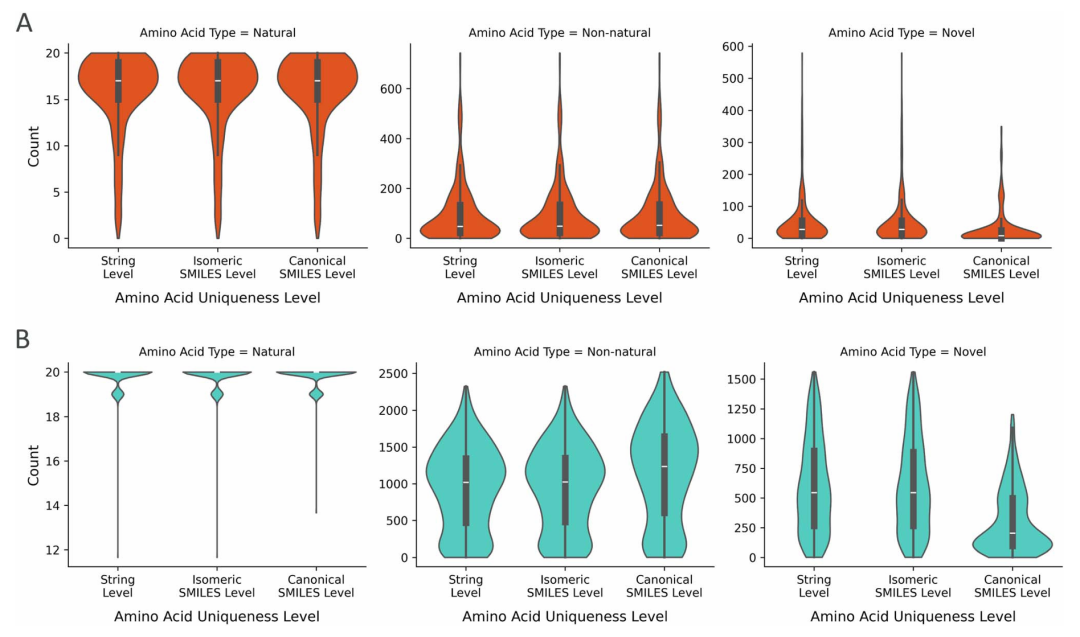
图 6:不同类型氨基酸的独特性分布。图中按氨基酸类型(天然、训练集中的非天然、全新),展示了两种采样方法在三种独特性水平上的分布。束搜索和多项式采样均能生成三类氨基酸,其中多项式采样生成的数量更丰富,且规范 SMILES 水平下非天然氨基酸占比增加、全新氨基酸占比下降,再次凸显立体化学对多样性的影响。
图 6:不同类型氨基酸的独特性分布。图中按氨基酸类型(天然、训练集中的非天然、全新),展示了两种采样方法在三种独特性水平上的分布。束搜索和多项式采样均能生成三类氨基酸,其中多项式采样生成的数量更丰富,且规范 SMILES 水平下非天然氨基酸占比增加、全新氨基酸占比下降,再次凸显立体化学对多样性的影响。
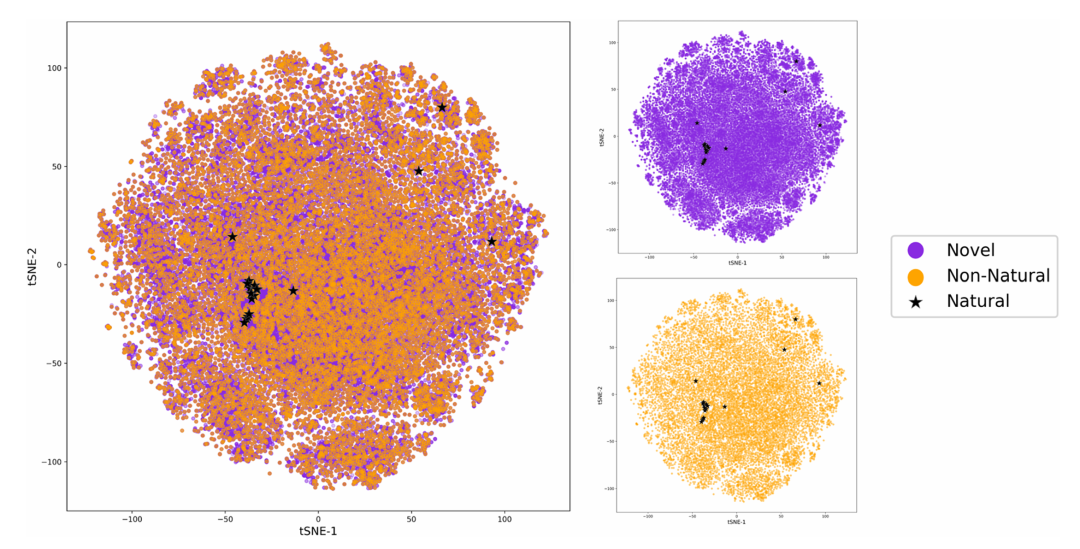
图 7:氨基酸化学空间的 t-SNE 可视化。该图通过 t-SNE 降维,展示了天然氨基酸、训练集中的非天然氨基酸和全新氨基酸在化学空间的分布。三类氨基酸分布趋势相似,全新氨基酸与训练集中的非天然氨基酸重叠度高,表明模型生成的新氨基酸符合学习到的化学规则,来自合理的化学空间。
图 7:氨基酸化学空间的 t-SNE 可视化。该图通过 t-SNE 降维,展示了天然氨基酸、训练集中的非天然氨基酸和全新氨基酸在化学空间的分布。三类氨基酸分布趋势相似,全新氨基酸与训练集中的非天然氨基酸重叠度高,表明模型生成的新氨基酸符合学习到的化学规则,来自合理的化学空间。
拓扑语境理解能力
针对肽拓扑结构的完整性测试表明,即使输入肽缺少环化所需的关键氨基酸(如二硫键桥接所需的含巯基氨基酸),模型仍能生成正确的氨基酸完成拓扑构建,总体有效性达98.3%。这一结果证实PepINVENT不仅能生成氨基酸,更能理解肽的整体结构逻辑,为复杂拓扑肽的设计提供了可靠支持。
强化学习驱动的定向优化:从拓扑控制到多参数优化
拓扑结构的精准调控
通过强化学习,PepINVENT可实现对肽拓扑结构的定向设计。实验中,研究团队基于最大环尺寸设计了三种评分函数:sigmoid函数最大化环尺寸,双sigmoid函数限定头-尾环化范围,反向sigmoid函数抑制环形成以生成线性肽。结果显示,模型在100步内即可收敛到目标拓扑:最大化环尺寸策略生成了含二硫键的大环肽(环原子数30-35),甚至出现训练集中未有的双环结构;限定范围策略稳定生成头-尾环化肽;反向策略则高效生成线性肽,且所有情况下的肽有效性均保持在90%以上。
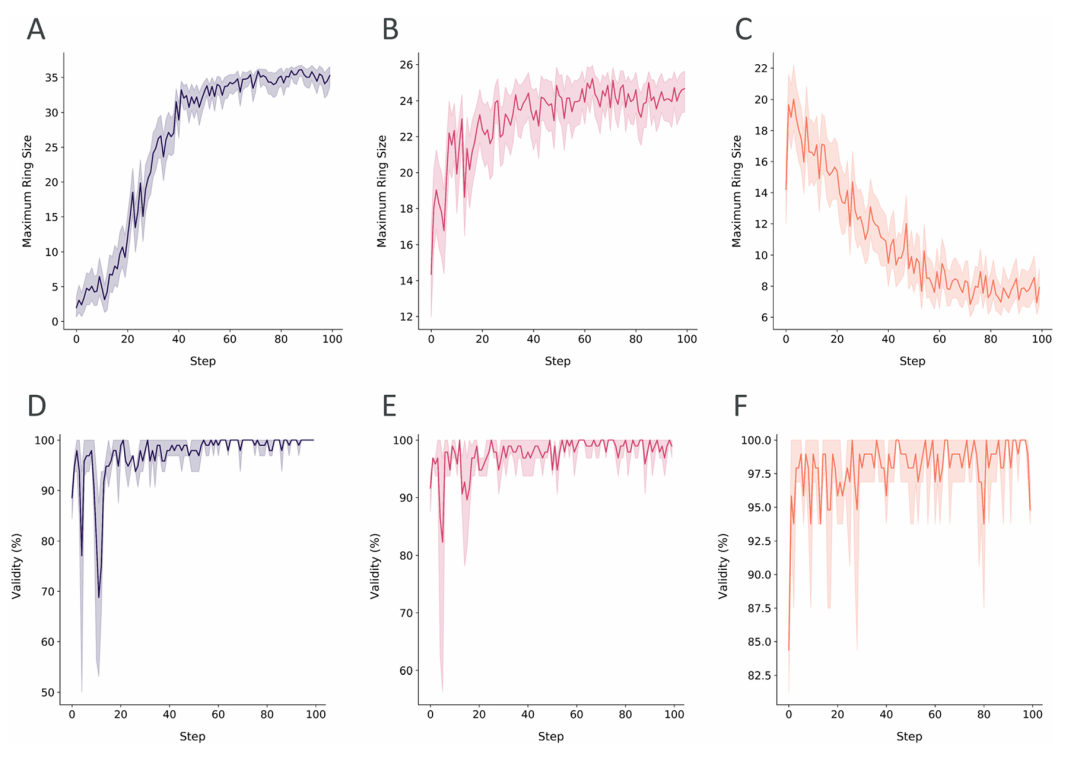
图 8:强化学习中环尺寸和有效性的变化。图中展示了强化学习过程中,不同目标(最大化环尺寸、限定环尺寸范围、最小化环尺寸)下的平均环尺寸(A-C)和肽的有效性(D-F)变化。结果显示模型在约 40 步内可收敛到目标拓扑,且生成肽的有效性始终保持在较高水平(>90%),体现了对拓扑结构的灵活调控能力。
图 8:强化学习中环尺寸和有效性的变化。图中展示了强化学习过程中,不同目标(最大化环尺寸、限定环尺寸范围、最小化环尺寸)下的平均环尺寸(A-C)和肽的有效性(D-F)变化。结果显示模型在约 40 步内可收敛到目标拓扑,且生成肽的有效性始终保持在较高水平(>90%),体现了对拓扑结构的灵活调控能力。
Rev结合肽的多参数优化案例
在实际药物设计场景中,研究团队针对HIV治疗靶点Rev结合肽(RBP)进行了优化。天然RBP膜渗透性差,团队掩盖了对生物活性影响较小的氨基酸(如丙氨酸、甘氨酸),通过强化学习同时优化四个目标:维持大环拓扑、提升溶解度(CAMSOL-PTM预测)、增强渗透性(XGBoost分类器)、规避毒性亚结构。
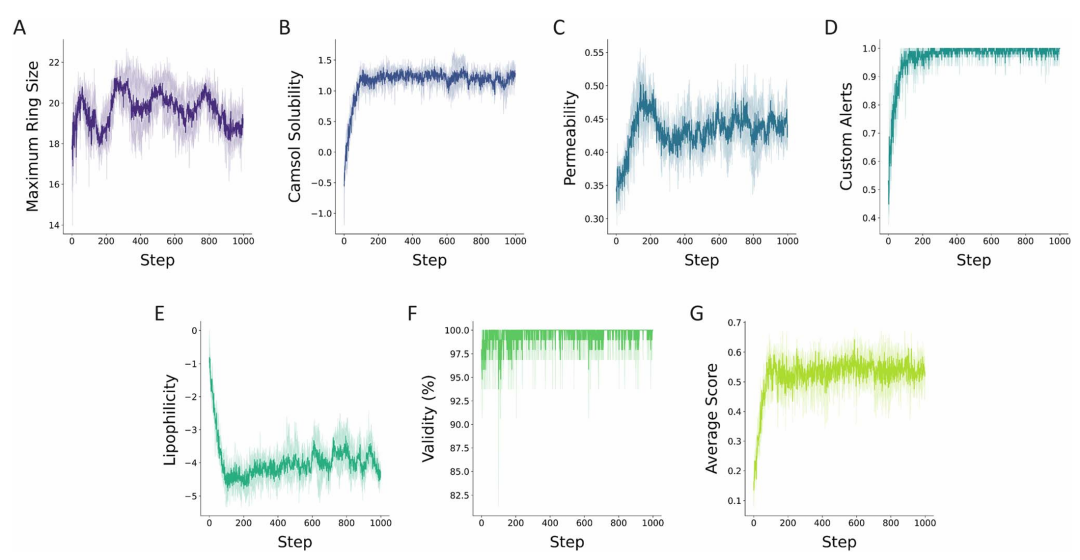
图 9:Rev 结合肽多参数优化的评分变化。此图跟踪了强化学习优化 Rev 结合肽时各评分组件的变化,包括拓扑约束(大环结构)、溶解度、渗透性、自定义警报(规避毒性亚结构),以及脂溶性、有效性和综合评分。过程显示模型先优化溶解度,再提升渗透性,最终实现多参数平衡。
图 9:Rev 结合肽多参数优化的评分变化。此图跟踪了强化学习优化 Rev 结合肽时各评分组件的变化,包括拓扑约束(大环结构)、溶解度、渗透性、自定义警报(规避毒性亚结构),以及脂溶性、有效性和综合评分。过程显示模型先优化溶解度,再提升渗透性,最终实现多参数平衡。
学习过程显示,模型在初始100步内优先优化溶解度,随后在可溶性空间中提升渗透性,最终生成了5146种高渗透性(评分>0.6)、高溶解度的大环肽。分析表明,模型倾向于通过引入杂环结构改善渗透性,这与已知的肽修饰策略一致,证明其设计结果具有化学合理性。
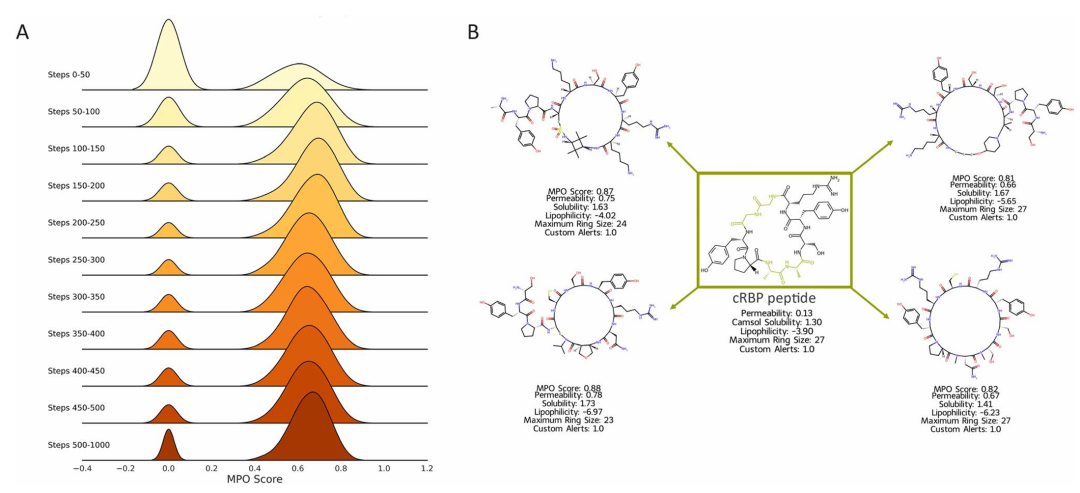
图 10:多参数优化(MPO)得分分布及示例肽。(A)脊线图展示了前 500 步及后续过程中生成肽的 MPO 得分分布,显示模型在 200 步左右聚焦于高得分肽,随后因多样性过滤探索更多设计;(B)展示了生成的示例肽及其 MPO 各组件得分,这些肽围绕输入的环化 Rev 结合肽(cRBP)进行设计,体现了优化效果。
图 10:多参数优化(MPO)得分分布及示例肽。(A)脊线图展示了前 500 步及后续过程中生成肽的 MPO 得分分布,显示模型在 200 步左右聚焦于高得分肽,随后因多样性过滤探索更多设计;(B)展示了生成的示例肽及其 MPO 各组件得分,这些肽围绕输入的环化 Rev 结合肽(cRBP)进行设计,体现了优化效果。
结论与展望
PepINVENT作为开源框架,首次实现了超越预设氨基酸库的肽类生成设计,其核心价值在于:以化学感知的方式学习肽的构建逻辑,支持天然与非天然氨基酸的灵活生成;结合强化学习可实现拓扑控制、多参数优化等目标导向设计;生成的全新NNAAs及肽结构具有化学有效性与合成潜力。
该工具为肽类药物研发提供了强大助力,可广泛应用于从头设计、肽模拟物开发、先导化合物优化等场景。未来通过整合合成可行性评估等模块,PepINVENT有望进一步缩短从设计到实验验证的流程,加速肽类新药的发现进程。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-15,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

