Nat. Commun. | 原子、键双扩散+性质引导的靶点感知3D分子生成模型
Nat. Commun. | 原子、键双扩散+性质引导的靶点感知3D分子生成模型
用户1151118
发布于 2026-01-08 13:30:21
发布于 2026-01-08 13:30:21
在 AI 辅助药物设计的赛道上,3D 分子生成模型常陷入 “两难困境”—— 要么结构离谱(比如出现能量不稳的三元环),要么类药性拉胯(结合力强却难合成)。【现在的扩散模型,很难同时有效地提升亲和力和成药性】近期,由华东师范大学与临港实验室李洪林教授团队发表于《Nature Communications》的研究 “Target-aware 3D molecular generation based on guided equivariant diffusion” 提出的 DiffGui 模型,创新性地将键扩散与性质引导融入 E (3) 等变扩散框架,为解决上述难题提供了系统性方案,其在从头药物设计、先导化合物优化及突变靶点适配等场景的优异表现,有望重塑 SBDD 的技术路径。

传统药物研发中,先导化合物的发现是决定项目成败的关键环节,但依赖随机筛选或基于配体的设计(LBDD)存在明显局限——前者需面对的庞大化学空间,计算成本极高且假阳性率突出;后者因未纳入靶蛋白结构信息,对无已知配体的新靶点束手无策。近年来兴起的3D分子生成模型虽转向SBDD思路,但仍有两大瓶颈:一是自回归模型(如ResGen、PocketFlow)需按固定顺序生成原子,易丢失全局信息,导致分子片段化或误差累积;二是现有扩散模型(如GCDM、TargetDiff)虽采用非自回归生成,却因仅建模原子扩散、忽略键的动态变化,常出现键型误判(如基于静态键长推断键型,微小坐标偏差即导致结构失效),且未明确融入类药性属性,生成分子难以满足临床转化需求。
DiffGui的核心创新在于构建了“原子-键协同扩散”与“多属性引导”的双机制框架,从根本上解决了上述问题。在模型架构上,DiffGui将正向扩散过程分为两个阶段:第一阶段仅对原子施加微量噪声以保持其动态性,重点将键型逐步扩散至无键先验分布,使键型推断能基于原子间的动态距离而非静态值,避免键长与键型的错配;第二阶段再将原子类型与坐标扰动至先验分布,确保扩散过程中键型学习始终贴合真实键长规律。同时,模型采用E(3)-等变图神经网络(GNN)更新原子与键的表征,严格保留分子的旋转、平移与置换对称性——这一设计对3D分子生成至关重要,意味着无论靶蛋白口袋如何旋转,生成的配体都能保持正确的空间构象与结合模式。
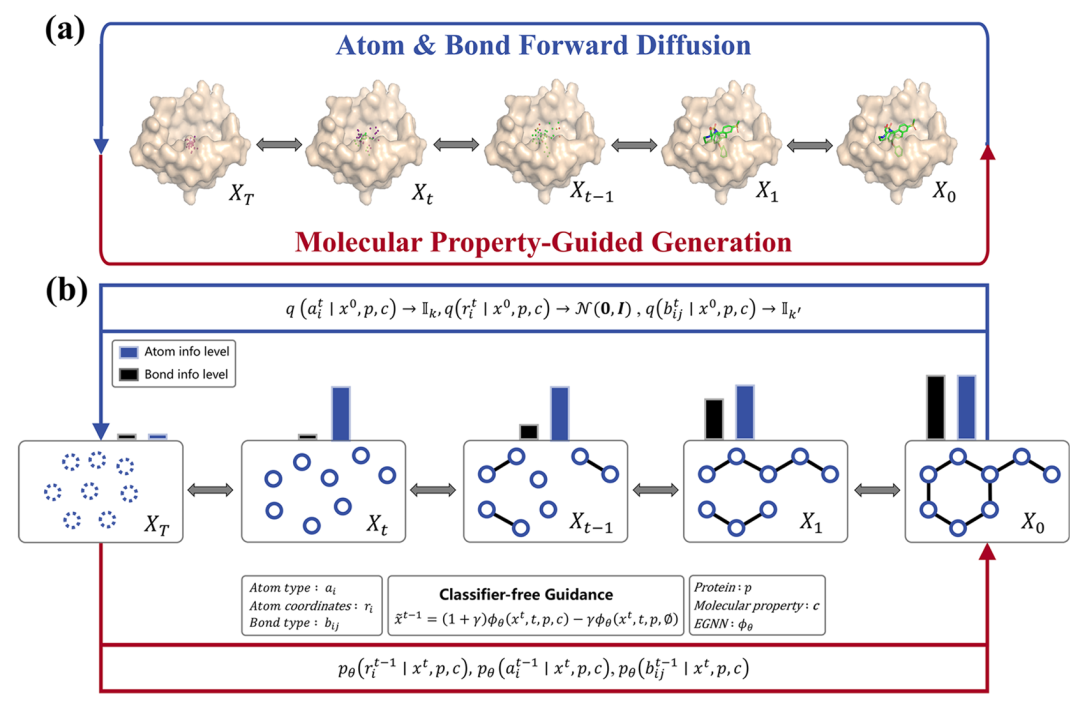
<b>DiffGui 框架概览。</b>该图分为 (a) 和 (b) 两个子图,整体展示了 DiffGui 模型的扩散与生成流程及结构细节。子图 (a) 呈现了 DiffGui 中原子与键的正向扩散过程(从初始状态 X₀逐步添加噪声至 Xᵀ),以及基于分子性质引导的反向生成过程,清晰体现了 “正向扰动 - 反向去噪” 的核心逻辑;子图 (b) 详细拆解了模型结构,包括原子与键的信息层级变化、分类器 - free 引导的数学表达式(通过线性组合条件与无条件模型得分估计实现性质引导),以及原子类型、键类型与原子坐标在反向过程中的联合预测分布,直观揭示了键扩散与性质引导如何协同作用于分子生成。
DiffGui 框架概览。该图分为 (a) 和 (b) 两个子图,整体展示了 DiffGui 模型的扩散与生成流程及结构细节。子图 (a) 呈现了 DiffGui 中原子与键的正向扩散过程(从初始状态 X₀逐步添加噪声至 Xᵀ),以及基于分子性质引导的反向生成过程,清晰体现了 “正向扰动 - 反向去噪” 的核心逻辑;子图 (b) 详细拆解了模型结构,包括原子与键的信息层级变化、分类器 - free 引导的数学表达式(通过线性组合条件与无条件模型得分估计实现性质引导),以及原子类型、键类型与原子坐标在反向过程中的联合预测分布,直观揭示了键扩散与性质引导如何协同作用于分子生成。
更具突破性的是DiffGui的“属性引导”机制。不同于传统扩散模型仅追求高结合亲和力,该模型将结合亲和力(Vina评分)、类药性(QED)、合成可及性(SA)、辛醇-水分配系数(LogP)及拓扑极性表面积(TPSA)等5类关键属性,通过无分类器引导(classifier-free guidance)融入训练与采样过程。具体而言,模型同时训练无条件生成与属性条件生成两个分支,训练时以一定概率将属性标签设为空值,采样时通过线性组合两个分支的得分估计,实现对属性引导强度的灵活调控。这种设计无需额外训练属性分类器,既简化了流程,又能确保生成分子在“高亲和力”与“类药性”间达到平衡——例如,在PDBbind数据集上,DiffGui生成分子的QED值显著高于GCDM、DiffSBDD等基线模型,同时SA值(0.678)在扩散模型中最优,证明其生成分子兼具活性与合成可行性。
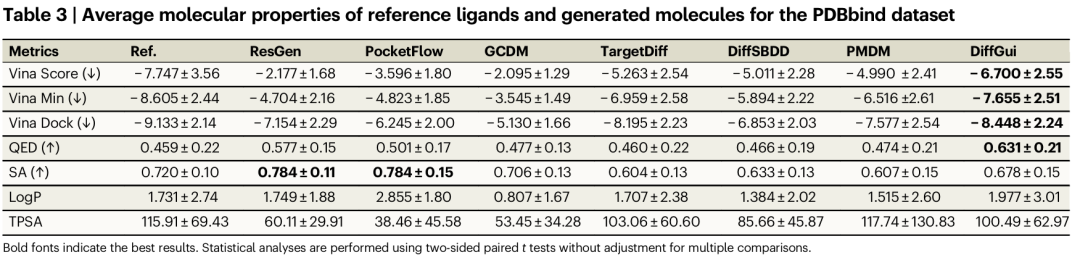
<b>PDBbind 数据集参考配体与生成分子的平均分子性质。</b>DiffGui 的三种 Vina 得分(-6.700、-7.655、-8.448)均为最低(得分越低亲和力越高),QED 值最优,LogP 值(1.782-3.642)处于 1-3 的理想药物范围,SA 值(0.678)在扩散模型中最高,仅略低于生成片段化分子的 ResGen(0.784)。
PDBbind 数据集参考配体与生成分子的平均分子性质。DiffGui 的三种 Vina 得分(-6.700、-7.655、-8.448)均为最低(得分越低亲和力越高),QED 值最优,LogP 值(1.782-3.642)处于 1-3 的理想药物范围,SA 值(0.678)在扩散模型中最高,仅略低于生成片段化分子的 ResGen(0.784)。
DiffGui在多维度评估中均展现出SOTA性能。在分子结构合理性上,通过Jensen-Shannon(JS)散度衡量生成分子与参考分子的键长、键角、二面角分布相似度——在PDBbind数据集上,DiffGui的C-C键距离JS散度仅为0.1815,全原子对距离JS散度低至0.0486,远低于ResGen(0.4034/0.1860)、TargetDiff(0.2265/0.0604)等模型,且生成分子中五、六元环占比超95%,与真实药物分子的环系分布高度一致。在全局构象准确性上,DiffGui生成分子与RDKit优化构象的RMSD值稳定在1Å左右,既避免了ResGen等自回归模型因片段化导致的RMSD偏低(实际为分子不完整),又克服了GCDM因计算成本限制导致的构象简化问题。
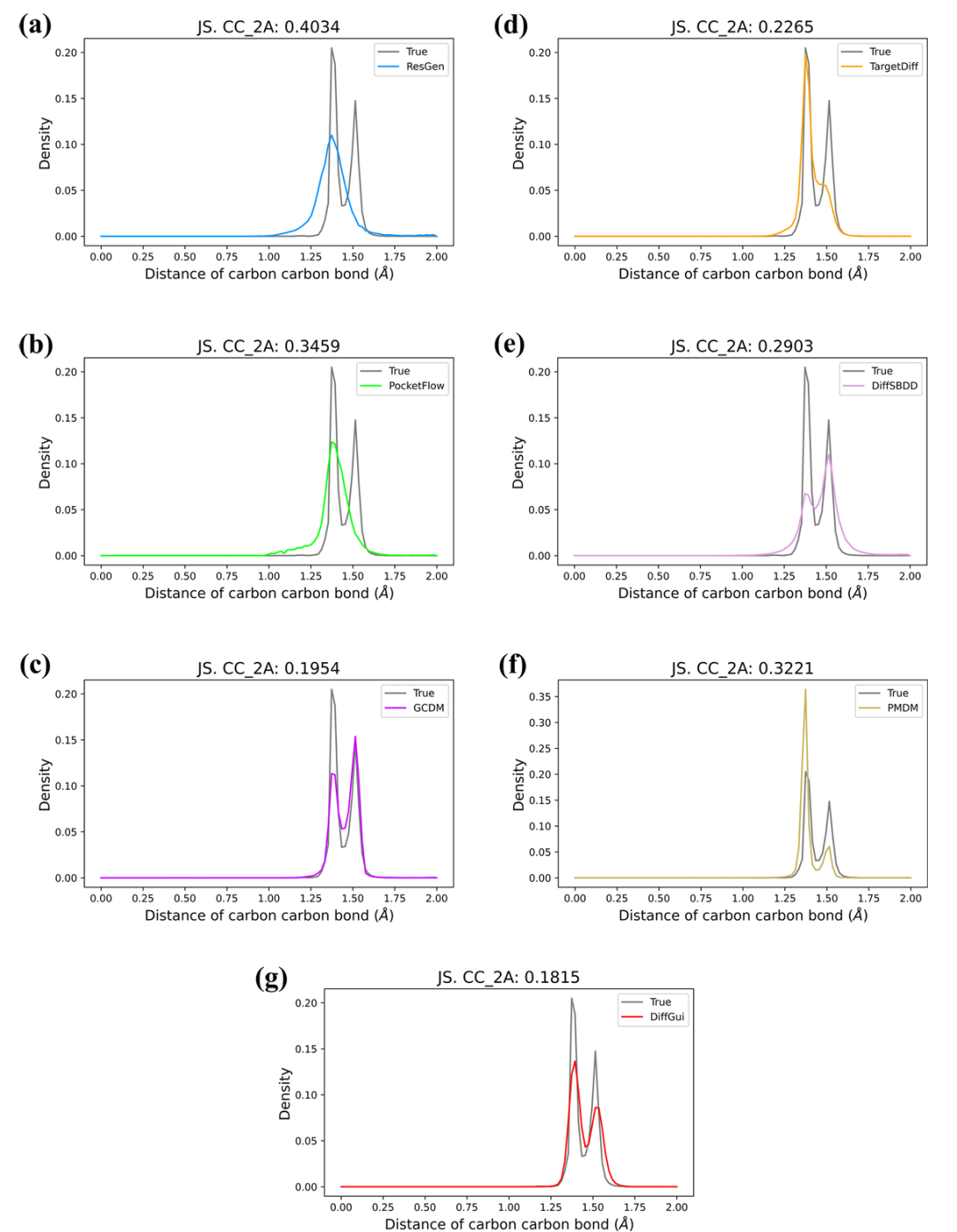
<b>PDBbind 数据集上 C-C 键距离分布对比。</b>该图包含 7 个子图(a-g),分别对比了 ResGen、PocketFlow、GCDM、TargetDiff、DiffSBDD、PMDM 及 DiffGui 七种模型生成分子的 C-C 键距离分布与参考分子(灰色线)的差异,每个子图顶部标注了 2.0Å 范围内 C-C 键距离的 Jensen-Shannon(JS)散度值。参考分子的 C-C 键距离主要分布在 1.3-1.6Å,且在 1.4Å 和 1.5Å 处有两个明显峰值;ResGen、PocketFlow 等模型或仅出现单一峰值(如 ResGen 在 1.4Å 附近),或峰值与真实分布偏差较大(如 PMDM),而 DiffGui(子图 g)生成的分布与参考分子高度重合,JS 散度低至 0.1815,为所有模型中最低,证明其在键长分布建模上的优越性。
PDBbind 数据集上 C-C 键距离分布对比。该图包含 7 个子图(a-g),分别对比了 ResGen、PocketFlow、GCDM、TargetDiff、DiffSBDD、PMDM 及 DiffGui 七种模型生成分子的 C-C 键距离分布与参考分子(灰色线)的差异,每个子图顶部标注了 2.0Å 范围内 C-C 键距离的 Jensen-Shannon(JS)散度值。参考分子的 C-C 键距离主要分布在 1.3-1.6Å,且在 1.4Å 和 1.5Å 处有两个明显峰值;ResGen、PocketFlow 等模型或仅出现单一峰值(如 ResGen 在 1.4Å 附近),或峰值与真实分布偏差较大(如 PMDM),而 DiffGui(子图 g)生成的分布与参考分子高度重合,JS 散度低至 0.1815,为所有模型中最低,证明其在键长分布建模上的优越性。
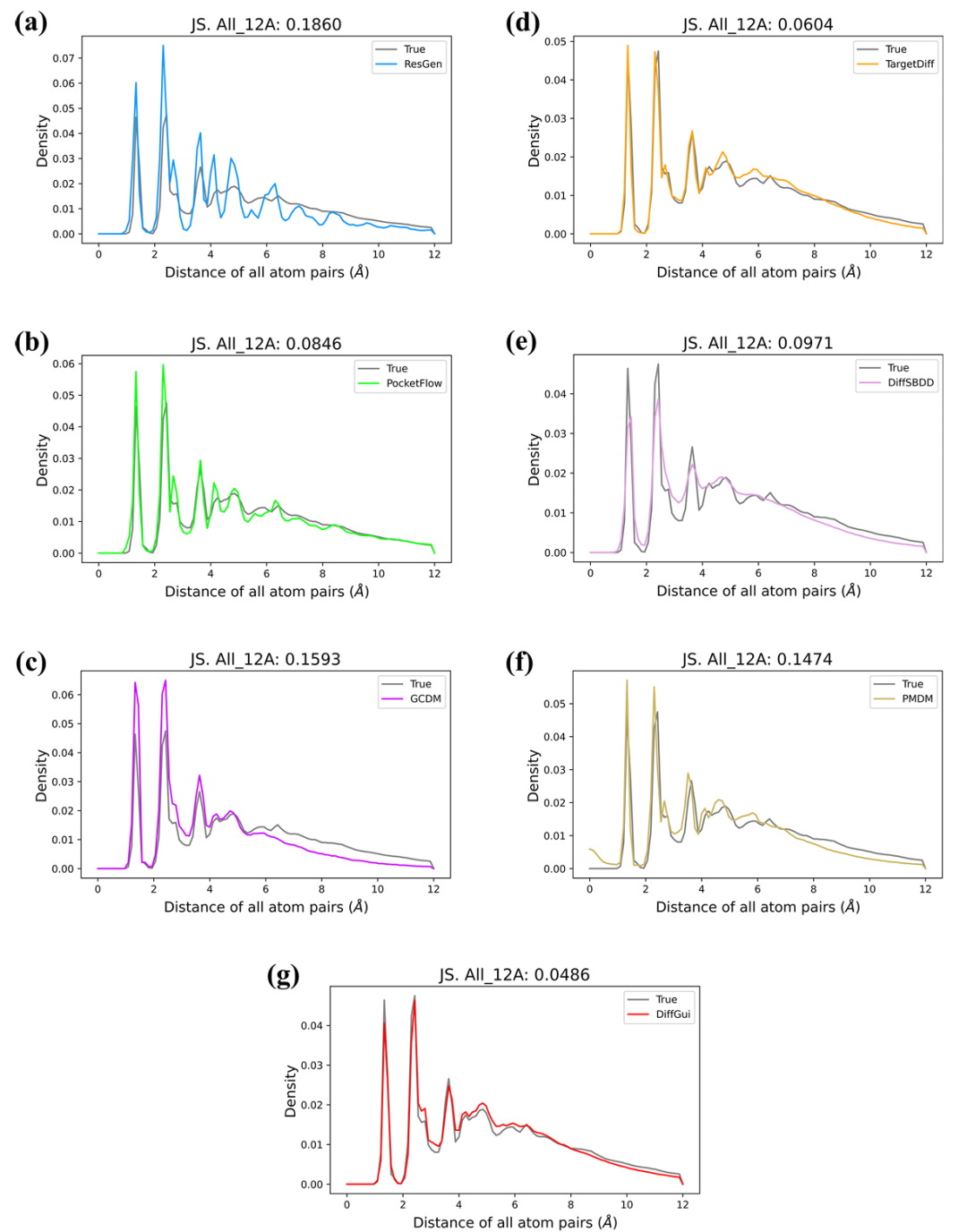
<b>PDBbind 数据集上全原子对距离分布对比。</b>该图同样包含 7 个子图(a-g),对比七种模型生成分子的全原子对距离分布与参考分子(灰色线)的差异,子图顶部标注了 12.0Å 范围内全原子对距离的 JS 散度值。参考分子的全原子对距离在 1.5Å、2.5Å、3.5Å 处有三个显著峰值,且在 4-6.5Å 处有延伸的肩峰;ResGen、GCDM 等模型难以同时拟合短程与长程分子相互作用(如 ResGen 无法复现多峰值特征),而 DiffGui(子图 g)生成的分布能精准匹配参考分子的峰值位置与强度,JS 散度仅为 0.0486,显著优于其他模型,表明其能有效捕捉分子全局几何信息。
PDBbind 数据集上全原子对距离分布对比。该图同样包含 7 个子图(a-g),对比七种模型生成分子的全原子对距离分布与参考分子(灰色线)的差异,子图顶部标注了 12.0Å 范围内全原子对距离的 JS 散度值。参考分子的全原子对距离在 1.5Å、2.5Å、3.5Å 处有三个显著峰值,且在 4-6.5Å 处有延伸的肩峰;ResGen、GCDM 等模型难以同时拟合短程与长程分子相互作用(如 ResGen 无法复现多峰值特征),而 DiffGui(子图 g)生成的分布能精准匹配参考分子的峰值位置与强度,JS 散度仅为 0.0486,显著优于其他模型,表明其能有效捕捉分子全局几何信息。
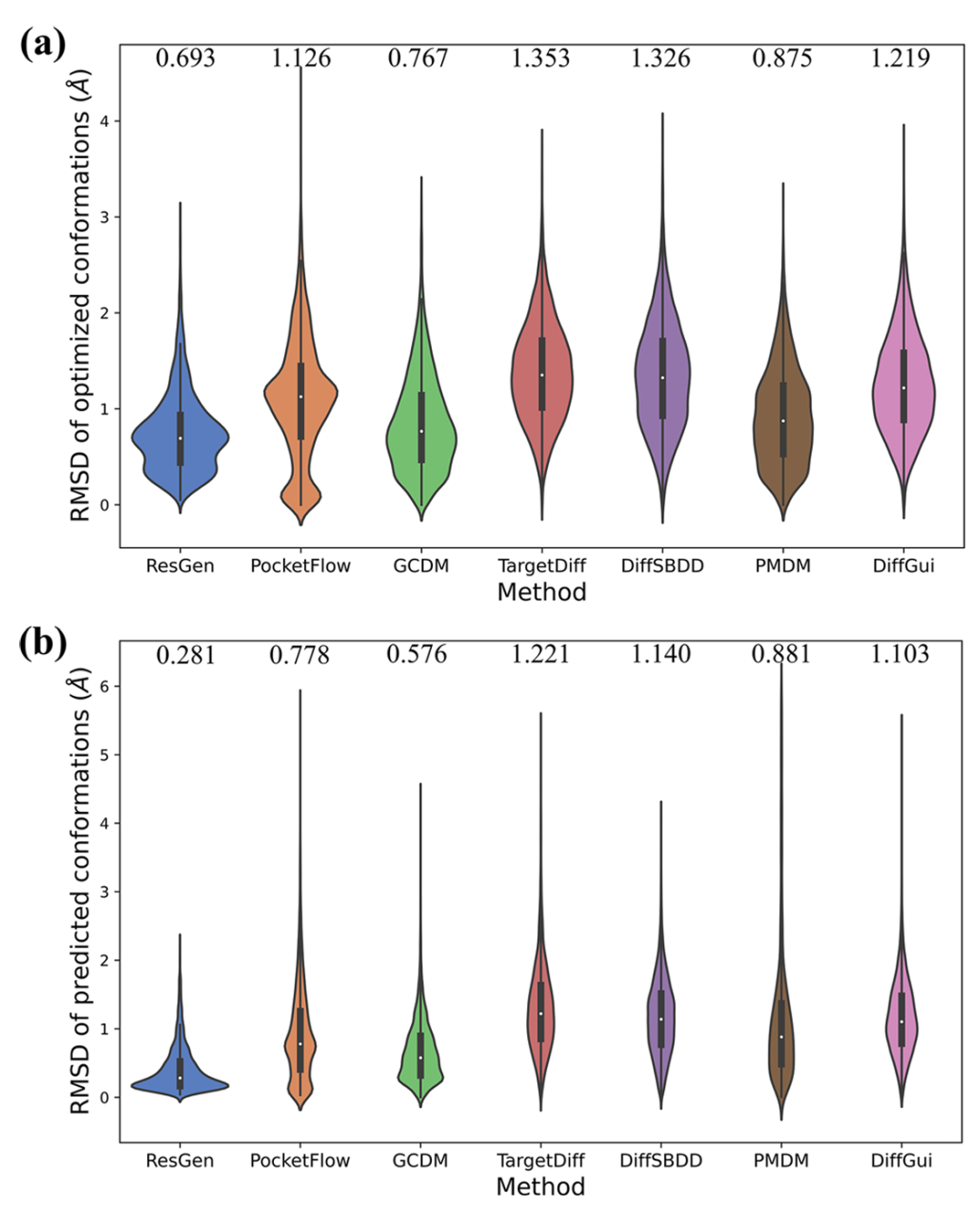
<b>PDBbind 数据集上生成构象与优化 / 预测构象的 RMSD 分布对比。</b>该图包含 2 个子图(a-b),以小提琴图形式展示七种模型生成分子的 RMSD(均方根偏差)分布,子图顶部标注了中位数 RMSD 值,RMSD 用于衡量生成构象与 RDKit 优化构象(a)、生成构象与 ETKDG 预测构象(b)的全局几何相似度,值越低表示构象越合理。ResGen 和 GCDM 虽展现出较低的中位数 RMSD,但这源于其生成小分子片段(ResGen 易提前终止,GCDM 因计算成本简化生成);而 DiffGui 在两种场景下均稳定实现约 1Å 的中位数 RMSD,且分布集中,证明其生成的分子尺寸适宜、3D 构象能良好适配蛋白质结合口袋,避免了片段化或构象失真问题。
PDBbind 数据集上生成构象与优化 / 预测构象的 RMSD 分布对比。该图包含 2 个子图(a-b),以小提琴图形式展示七种模型生成分子的 RMSD(均方根偏差)分布,子图顶部标注了中位数 RMSD 值,RMSD 用于衡量生成构象与 RDKit 优化构象(a)、生成构象与 ETKDG 预测构象(b)的全局几何相似度,值越低表示构象越合理。ResGen 和 GCDM 虽展现出较低的中位数 RMSD,但这源于其生成小分子片段(ResGen 易提前终止,GCDM 因计算成本简化生成);而 DiffGui 在两种场景下均稳定实现约 1Å 的中位数 RMSD,且分布集中,证明其生成的分子尺寸适宜、3D 构象能良好适配蛋白质结合口袋,避免了片段化或构象失真问题。
在分子属性与功能上,DiffGui的优势更为显著。PDBbind数据集结果显示,其生成分子的Vina评分(-6.700)、Vina Min(-7.655)与Vina Dock(-8.448)均为最低(评分越低亲和力越强),且蛋白质-配体相互作用指纹相似度最高,2D结构相似度最低——这意味着DiffGui能在保持关键结合作用的同时,生成具有新颖骨架的分子,有效规避现有模型“生成分子与参考配体高度相似”的局限。更重要的是,湿实验验证进一步证实了模型的实用价值:针对RSK4激酶(PDB ID 6g77)生成的化合物,IC50值分别达到215.0 nM与111.1 nM;针对二氢乳清酸脱氢酶(DHODH)的先导优化中,优化后化合物的IC50值从8.02 μM降至4.27 μM,从32.20 nM降至10.45 nM,且通过引入羧基、氟原子等基团,增强了与靶蛋白关键残基的相互作用(如与R136形成盐桥、与Q47形成氢键)。
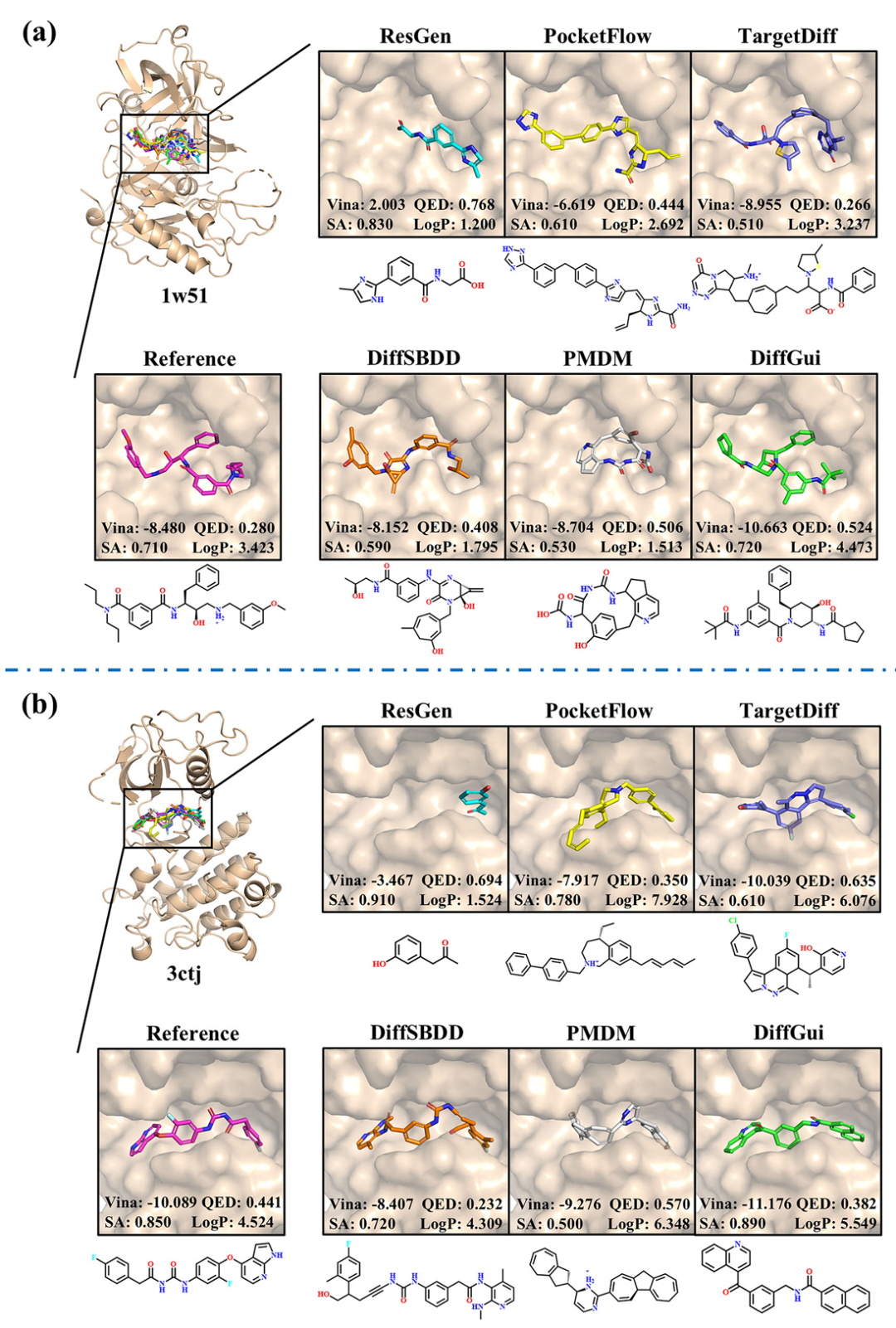
<b>PDBbind 测试集靶点生成分子展示。</b>该图包含 2 个子图(a-b),分别展示针对 PDBbind 测试集中 1w51(a,靶点为 β- 分泌酶 1)和 3ctj(b,靶点为酪氨酸激酶)两个蛋白靶点生成的最优分子。每个子图中选取各模型生成的 PB-valid 分子中对接得分最佳的配体进行可视化,对比显示:ResGen 生成的小分子片段局限于子口袋易致脱靶,PocketFlow 生成的线性分子柔性高、类药性差,TargetDiff 生成的非芳香稠环稳定性低,而 DiffGui 生成的分子化学结构明确,能完全占据结合口袋,且与参考配体的结合模式高度相似,同时具备更优的 Vina 得分与成药性指标(如 QED、SA),部分分子的结合自由能(ΔG)甚至低于参考配体。
PDBbind 测试集靶点生成分子展示。该图包含 2 个子图(a-b),分别展示针对 PDBbind 测试集中 1w51(a,靶点为 β- 分泌酶 1)和 3ctj(b,靶点为酪氨酸激酶)两个蛋白靶点生成的最优分子。每个子图中选取各模型生成的 PB-valid 分子中对接得分最佳的配体进行可视化,对比显示:ResGen 生成的小分子片段局限于子口袋易致脱靶,PocketFlow 生成的线性分子柔性高、类药性差,TargetDiff 生成的非芳香稠环稳定性低,而 DiffGui 生成的分子化学结构明确,能完全占据结合口袋,且与参考配体的结合模式高度相似,同时具备更优的 Vina 得分与成药性指标(如 QED、SA),部分分子的结合自由能(ΔG)甚至低于参考配体。
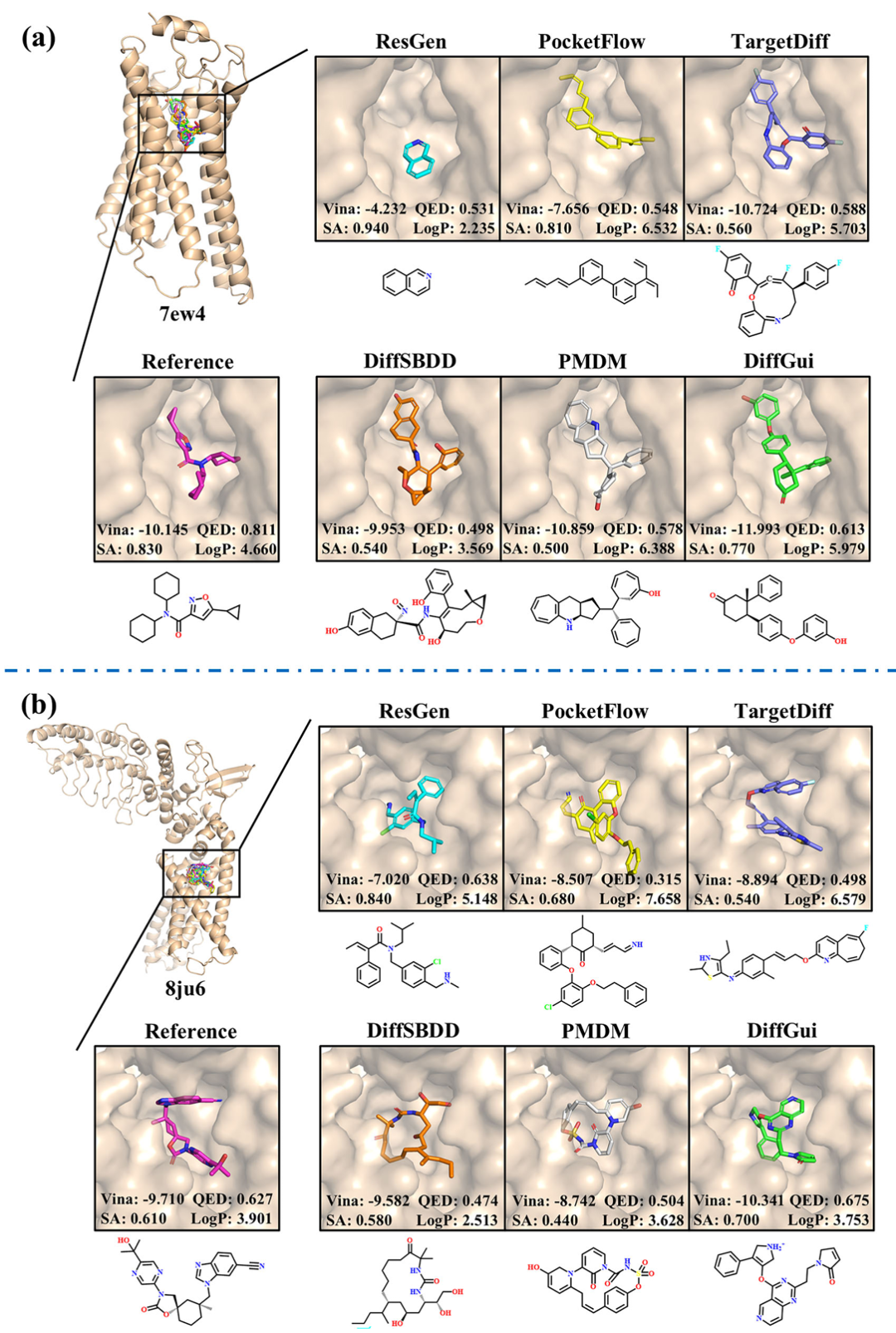
<b>PDBbind 数据集外靶点生成分子展示。</b>该图包含 2 个子图(a-b),展示针对 PDBbind 数据集外 7ew4(a,靶点为 G 蛋白偶联受体)和 8ju6(b,靶点为离子通道)两个跨膜蛋白靶点生成的最优分子。由于这两个靶点的参考配体与口袋残基相互作用较少,其他模型(如 TargetDiff、DiffSBDD)仍试图复制参考配体的稀疏结合模式,导致结合效果不佳;而 DiffGui 通过生成新颖的药效团结构,与关键残基建立新的相互作用,生成分子的对接得分虽未显著超越所有模型,但化学结构合理性与成药性(QED、SA)更优,且能适配跨膜蛋白口袋的特殊几何环境,体现了模型对非训练集靶点的泛化能力。
PDBbind 数据集外靶点生成分子展示。该图包含 2 个子图(a-b),展示针对 PDBbind 数据集外 7ew4(a,靶点为 G 蛋白偶联受体)和 8ju6(b,靶点为离子通道)两个跨膜蛋白靶点生成的最优分子。由于这两个靶点的参考配体与口袋残基相互作用较少,其他模型(如 TargetDiff、DiffSBDD)仍试图复制参考配体的稀疏结合模式,导致结合效果不佳;而 DiffGui 通过生成新颖的药效团结构,与关键残基建立新的相互作用,生成分子的对接得分虽未显著超越所有模型,但化学结构合理性与成药性(QED、SA)更优,且能适配跨膜蛋白口袋的特殊几何环境,体现了模型对非训练集靶点的泛化能力。
DiffGui的应用场景还延伸至药物研发的关键环节:在从头药物设计中,针对β-分泌酶1(1w51)、G蛋白偶联受体(7ew4)等不同类型靶点,生成分子均能完全占据结合口袋,且结合自由能(ΔG)低于参考配体;在先导优化中,通过“片段去噪”与“片段条件”两种采样策略,实现片段生长、骨架跃迁等任务——例如针对PI3K(3l13),以噻吩并嘧啶为种子片段生成的分子,既保留了与Lys802、Asp841的关键相互作用,又将吗啉环替换为呋喃、吡唑等基团,提升了类药性;在突变靶点适应上,针对KRAS G12D突变体(7rpz),DiffGui能敏锐捕捉口袋残基变化(如Asp12→Ala),调整生成分子的官能团(用羧酸替代胺基以适应电荷变化),而传统模型往往无法区分野生型与突变型口袋的细微差异。
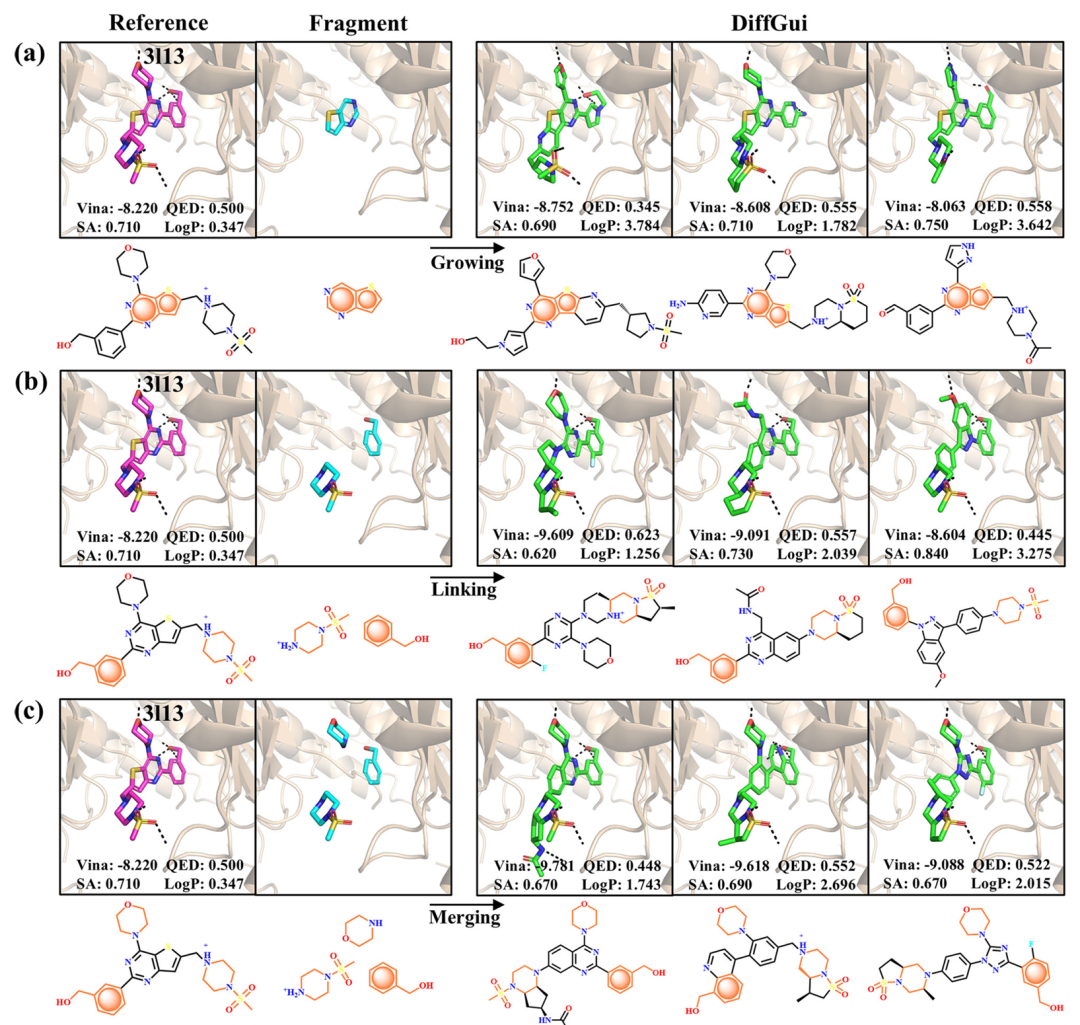
<b>基于片段去噪法的 3l13 靶点先导化合物优化展示。</b>该图包含 3 个子图(a-c),以 PDBid 3l13(靶点为 PI3K 激酶)为对象,展示 DiffGui 通过片段去噪法实现先导优化的三种场景,橙色标注固定种子片段。子图 (a) 为片段生长:以噻吩并嘧啶为种子,生成分子保留了参考配体与 Lys802、Asp841 等残基的相互作用,同时将吗啉环替换为呋喃、吡唑等更优基团,Vina 得分从 - 8.220 提升至 - 8.752;子图 (b) 为片段链接:输入两个片段实现骨架跃迁,生成分子的 Vina 得分达 - 9.609,QED 值提升至 0.623;子图 (c) 为片段融合:输入三个片段,生成分子的 Vina 得分低至 - 9.781,且 QED、SA 值均优于或持平参考配体,验证了 DiffGui 在片段驱动先导优化中的有效性。
基于片段去噪法的 3l13 靶点先导化合物优化展示。该图包含 3 个子图(a-c),以 PDBid 3l13(靶点为 PI3K 激酶)为对象,展示 DiffGui 通过片段去噪法实现先导优化的三种场景,橙色标注固定种子片段。子图 (a) 为片段生长:以噻吩并嘧啶为种子,生成分子保留了参考配体与 Lys802、Asp841 等残基的相互作用,同时将吗啉环替换为呋喃、吡唑等更优基团,Vina 得分从 - 8.220 提升至 - 8.752;子图 (b) 为片段链接:输入两个片段实现骨架跃迁,生成分子的 Vina 得分达 - 9.609,QED 值提升至 0.623;子图 (c) 为片段融合:输入三个片段,生成分子的 Vina 得分低至 - 9.781,且 QED、SA 值均优于或持平参考配体,验证了 DiffGui 在片段驱动先导优化中的有效性。
当然,DiffGui仍有可优化空间,例如生成分子中七元环占比约10%(虽低于PMDM的21.2%,但仍高于真实药物分子),未来可通过片段级扩散建模进一步降低;针对跨膜蛋白(如7ew4、8ju6),其3D药效团重叠度未显著优于基线模型,提示需加强对疏水性口袋结合模式的学习。但瑕不掩瑜,DiffGui通过原子-键协同扩散解决了结构合理性问题,通过属性引导平衡了活性与类药性,通过多场景适配验证了实用价值,为SBDD提供了兼具理论创新与应用潜力的新工具。
对于从事AI药物设计、计算生物学及药物化学的研究者而言,这篇文献的价值不仅在于提出了一个高性能模型,更在于确立了“结构合理性-属性可控性-场景适配性”三位一体的3D分子生成设计思路。随着后续模型在片段级生成、药代动力学属性融入等方向的优化,DiffGui有望进一步缩短从靶点到先导化合物的研发周期,为创新药物的高效发现提供有力支撑。建议相关领域同仁仔细研读原文,尤其关注其扩散过程设计、等变GNN实现及无分类器引导的细节,这对推动自身研究具有重要的借鉴意义。
参考文献
Hu, Q., Sun, C., He, H. et al. Target-aware 3D molecular generation based on guided equivariant diffusion. Nat Commun 16, 7928 (2025). https://doi.org/10.1038/s41467-025-63245-0
代码: https://github.com/QiaoyuHu89/DiffGui
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-26,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

