Soft-introspective VAEs:超越AlphaFold2,揭示K-Ras蛋白新视野
Soft-introspective VAEs:超越AlphaFold2,揭示K-Ras蛋白新视野

今天我们介绍华盛顿大学的David baker课题组发表在bioRxiv上的工作。探索蛋白质构象的整体,这些构象对功能有贡献,并且可以被小分子药物所靶向,仍是一个未解决的挑战。本文探讨了使用软自省变分自编码器(Soft-introspective Variational Autoencoders)来简化蛋白质结构集合生成问题中的维度挑战。通过将高维度的蛋白质结构数据转化为连续的低维表示,在此空间中进行由结构质量指标指导的搜索,接着使用RoseTTAFold来生成3D结构。本文使用这种方法为与癌症相关的蛋白质K-Ras生成集合,训练VAE使用部分可用的K-Ras晶体结构和MD模拟快照,并评估其对从训练中排除的晶体结构的采样范围。本文发现,潜在空间采样程序可以迅速生成具有高结构质量的集合,并能够在1埃范围内采样保留的晶体结构,其一致性高于MD模拟或AlphaFold2预测。

本文的创新点与贡献:
- 提出了利用软自省变分自动编码器潜在空间采样的方法来生成蛋白质集合。该方法将高维蛋白质结构数据转化为低维表示,通过在该空间中进行搜索,并引导结构质量指标,快速生成具有高结构质量的集合。
- 使用VAE对一部分可用的K-Ras晶体结构和MD模拟快照进行训练,并利用训练好的VAE快速生成结构集合。所生成的结构集合能够在与保留的晶体结构相比的1埃范围内进行采样,表现优于MD模拟和AlphaFold2预测。这些采样的结构足够重现保留的K-Ras结构中的隐藏口袋,可以用于小分子对接。
- 本文的方法为生成合理的蛋白质结构集合提供了一种经济高效的途径。相比于传统方法,VAE潜在空间采样方法能够快速生成高质量的结构集合,从而解决了在蛋白质功能和药物可及性研究中的集合生成问题。
在药物发现中,一个主要的挑战是识别可以被小分子药物靶向的隐匿性结合口袋。尽管近年来利用AlphaFold和RoseTTAFold在原始蛋白结构预测上取得了显著进展,但生成在结合小分子或蛋白质功能过程中可能的结构集合仍是一个问题,因为AlphaFold和RoseTTAFold主要生成单一结构,而不是集合。分子动力学(MD)轨迹通过模拟蛋白质在本机构附近的运动来生成蛋白质集合,但在进行小分子对接计算之前的集合生成中常常失败或需要长时间的计算密集型模拟。其他传统方法,如Rosetta和动力闭合(KIC)的循环取样,未能取样涉及隐匿口袋形成的构象变化。在深度学习方面,变分自动编码器已被用于生成一般蛋白质设计任务的替代主干,例如64残基主干的de novo设计和基于图的蛋白质设计。VAEs曾被用于取样蛋白质的构象空间,但需要视觉检查已训练的潜在空间或重点映射目标蛋白的apo和holo状态的MD模拟中的相关波动。
我们提出了采样变分自编码器的潜在空间可以为特定蛋白序列的集成生成提供答案。尽管以往的VAE方法主要关注众多不同的蛋白质,但特定蛋白质的VAE面临的挑战是有限的训练数据。我们认为,通过短MD轨迹快照和已有的晶体结构可以扩充所需的数据。我们以癌症关键靶点K-Ras为例进行了研究,因其具有高治疗价值和丰富的可用结构。
在探索不同的VAE架构后,我们使用K-Ras的MD模拟集成进行了训练,并评估了3D重建的质量。我们选择了二维RoseTTAFold模板特征来编码3D结构信息。重建的模板特征随后被用于RoseTTAFold的3D结构生成。我们使用RMSD衡量了重建的准确性,并在潜在空间中产生新的样本,最后使用RoseTTAFold生成3D坐标(见图1)。
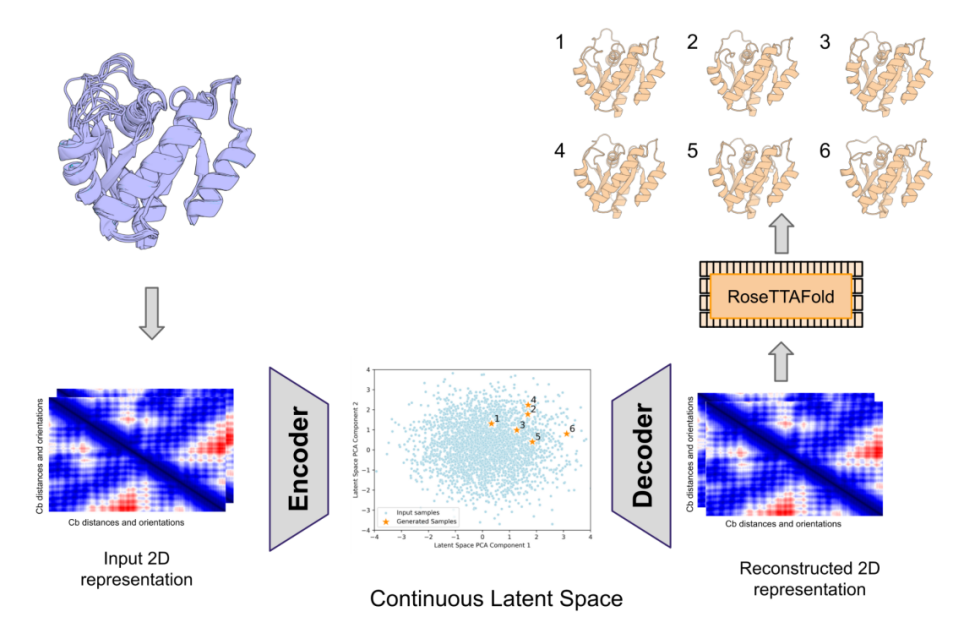
图1:基于VAE的集成生成方法。晶体结构和MD模拟的3D坐标被转换为RoseTTAFold 2D模板特征。解码后的模板特征通过RoseTTAFold转换为三维结构,并给出氨基酸序列。通过在潜在空间中采样,然后解码和射频结构生成来生成集成。
未纳入训练集的晶体结构的重建精度为我们提供了一个大概的精度界限,显示该方法可以有效地概括目标构象。我们对每个K-Ras结构训练了一个VAE,排除了该结构和其他结构,并评估了RoseTTAFold的3D坐标重建准确性。最佳结果来自软内省VAE架构,其重建精度在约256个潜在空间维度上稳定。对于大部分目标(13/20),VAE的重建精度达到亚埃级(RMSD <1),而AF2的结构预测只有2/20达到此精度(见图2)。
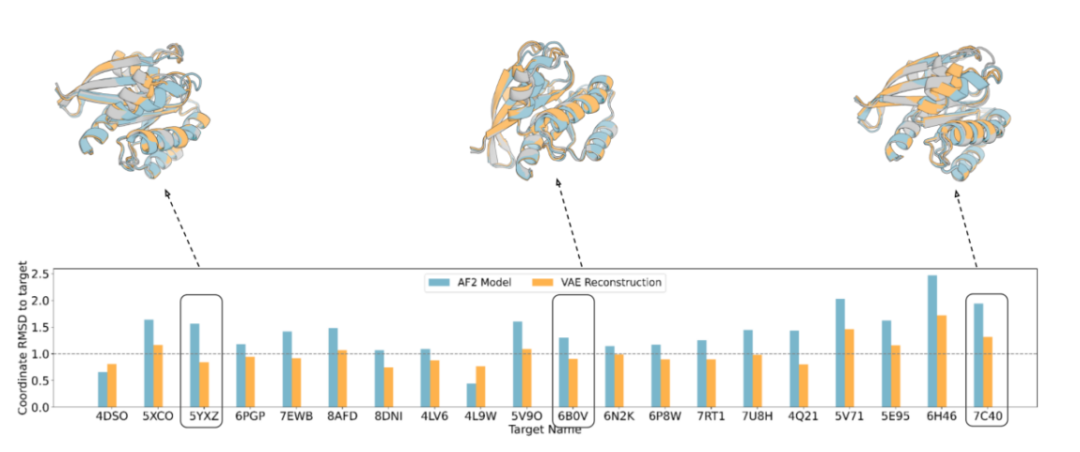
图2:VAE结构重建精度。最接近的AF2预测模型与利用RoseTTAFold生成的VAE解码模板特征重构模型的坐标RMSD。上面突出显示了3个目标的结构叠加,其中目标晶体为灰色,AF2预测为蓝色,VAE重建为橙色。
我们进一步探讨了通过在训练好的VAE的潜在空间中采样来生成K-Ras集合的可能性。为确保采样结构与序列大致一致且物理上合理,我们根据AF2预测的氨基酸序列距离分布来引导采样。从均值为0,方差为1的正态分布中生成样本,解码为相应的Cb距离图,计算与AF2预测的序列距离图的CCE,并通过对CCE值进行梯度下降进行潜在空间的局部优化,以限制从起点开始的总距离,防止收敛。解码和RF结构生成后,使用坐标RMSD评估样本,以目标晶体对整体结构和隐蔽口袋环境(定义为与配体结合口袋内5埃的残基)进行重构。
利用这种VAE引导的采样方法,我们生成了K-Ras结构集合,并再次留出单独的K-Ras晶体结构和从中导出的MD模拟快照,以及其他在1埃RMSD内的K-Ras晶体结构(及其MD快照)。我们通过确定它们如何接近留出结构来评估这些集合。本文方法的优势是可以快速生成集合(与MD模拟相比),且与增加的样本数相比,与留出结构的最近RMSD当然会减少。我们发现,对于大多数目标,3000个结构的集合比最近的训练集晶体结构、训练集MD模拟快照和最近的AF2模型更接近于留出结构(见图3)。
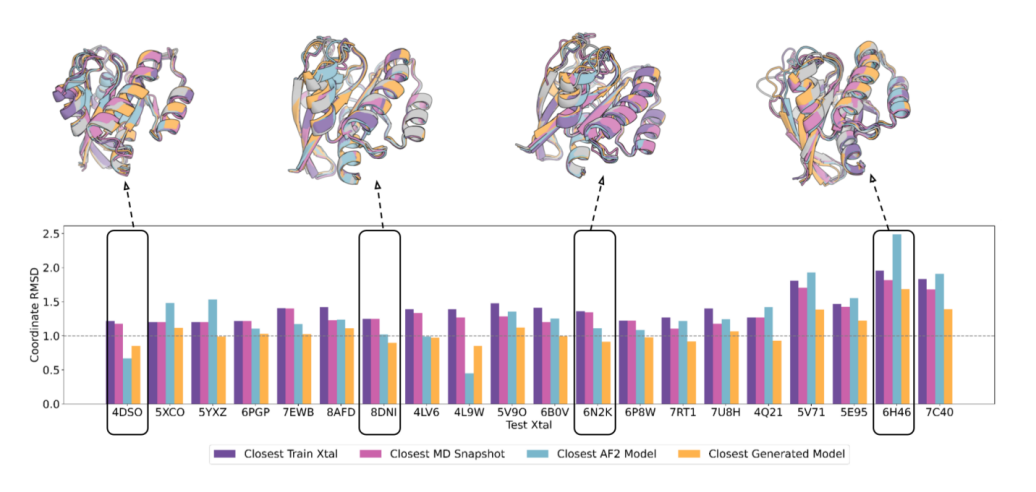
图3:与MD或AlphaFold生成的结构相比,VAE使采样更接近于K-Ras晶体结构。对于每个测试晶体结构(名称见条形图),使用RMSD大于1A的所有晶体结构的MD模拟数据训练VAE,并用于生成结构集合。条形表示最接近的训练晶体、最接近的训练样本、最接近的AF2模型和最接近的VAE生成样本与测试晶体的坐标误差。
对于小分子对接计算,采样替代配体结合口袋的几何结构尤为重要。比较生成集合中最近采样构象与留出结构之间的配体结合口袋残基的RMSD,显示集合在大多数情况下都比最近的训练MD快照或晶体结构更接近。结构叠加显示,生成的样本与目标结构的叠加配体不冲突,突出显示为橙色,因此可以无障碍地对接;而对于最接近的训练晶体和最接近的AF2模型,存在显著的冲突(见图4)。
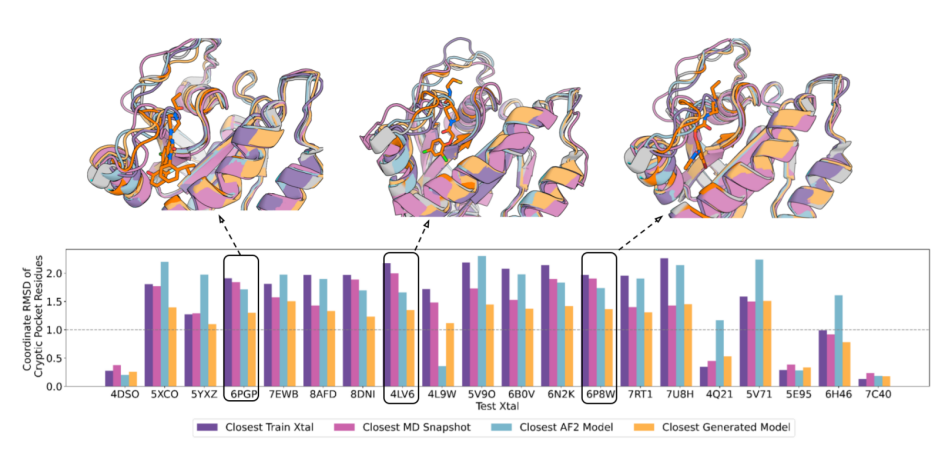
图4:K-Ras隐袋几何的VAE采样。图3对测试晶体结构的坐标误差仅计算结合位点残基(定义为配体结合袋5埃范围内的残基)。结构叠加图(上)显示配体抑制剂仅停靠在目标晶体上,其中隐式结合袋和配体在目标晶体结构上以橙色突出显示。
我们使用基于物理的GA-配体对接方法将配体对接到从VAE、训练示例和AF2模型生成的所有模型上。与上述观察结果一致,与AF2预测相比,配体原子的RMSD对于生成的样本集合始终较低,在大多数情况下,与MD集合的对接相比也较低(见图5)。
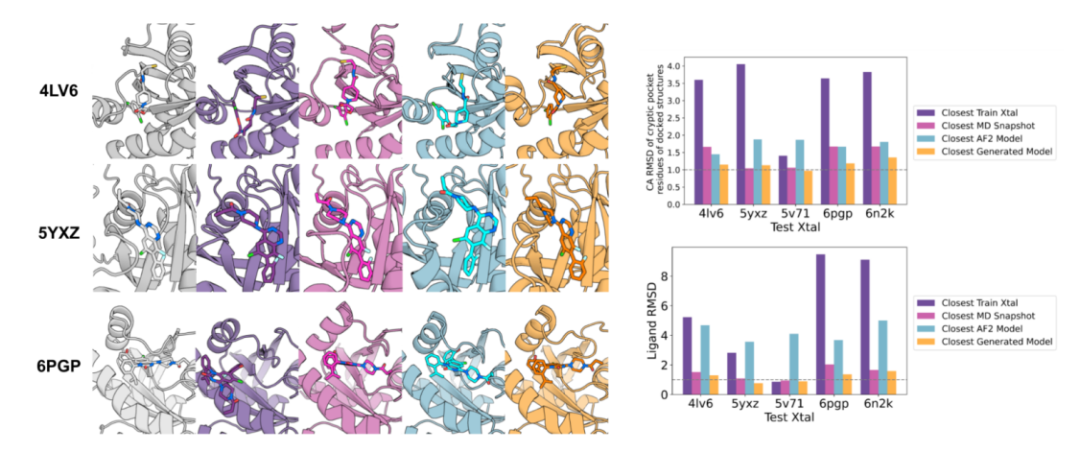
图5:小分子对接到VAE生成的集合中。利用GA-配体对接,将来自外延晶体结构的配体与蛋白质构象对接。左:训练集晶体结构(第2列)、MD快照(第3列)、AlphaFold模型(第4列)和VAE集合(第5列)中持有的晶体结构配合物(第1列)和最近的对接配合物(就配体上的RMSD而言)。右边的条形图显示了C-alpha坐标的RMSD的最近RMSD和配体原子上的最低RMSD(配体RMSD)。
讨论
我们基于VAE的采样方法允许从多个已知晶体结构的MD模拟快照中外推,生成更接近留出晶体结构的构象集合。这些集合足够准确地采样替代配体结合位点几何,以实现小分子配体的对接。我们的方法为从不同晶体结构起点的多个经典MD模拟轨迹中概括,生成几乎无限数量的可能样本提供了一种方法,且计算成本极低。我们超越了使用VAE模拟MD模拟采样空间的先前研究,通过两种方式利用AF2和RF深度神经网络中隐含的蛋白质序列-结构关系:首先,我们使用AF预测的距离分布来将潜在空间的采样集中在与氨基酸序列一致的区域;其次,我们使用RF从输出距离图生成3D坐标,确保物理真实性和局部序列-结构兼容性。
我们的方法还有明确的改进方向。首先,已知晶体结构的重建误差约为1埃是合理的,但挑战是许多不同构象之间的差异也是这个数量级,限制了该方法精确采样替代状态的能力。还有更低重建误差的VAE架构可以改进我们的方法,微调经过训练的VAE的FAPE损失也有可能(我们在初步测试中没有观察到这一点,但这值得进一步探讨)。其次,虽然AF2 CCE指标提供了合理的参考,但AF2是训练来生成单个结构的,因此使用这个指标来指导采样可能会限制多样性。通过预测给定目标的结构集合或为RoseTTAFold结构生成子采样目标MSA来引入更多的输出结构中的多样性,可以获得更好的结果。尽管存在这些局限性,我们的结果显示了深度生成模型在模拟确定蛋白质功能和药物化学性的构象集合方面的实用性。
参考资料
Mansoor, S., Baek, M., Park, H., Lee, G.R., & Baker, D. (2023). Protein Ensemble Generation through Variational Autoencoder Latent Space Sampling. bioRxiv.
