单细胞与bulk转录组联合分析构建并验证了一个非小细胞肺癌的预后模型
单细胞与bulk转录组联合分析构建并验证了一个非小细胞肺癌的预后模型
生信技能树jimmy
发布于 2024-03-05 10:58:50
发布于 2024-03-05 10:58:50
文章概述
基本信息
文章标题:Integrated Bulk and Single-cell RNA Sequencing Data Constructs and Validates a Prognostic Model for Non-small Cell Lung Cancer 发表时间:2024-01-01 发表杂志:Journal of Cancer 影响因子:3.9 在线阅读链接:https://www.jcancer.org/v15p0796.htm

文章摘要
背景:目前大多数关于非小细胞肺癌(NSCLC)预后模型构建的研究仅涉及批量RNA-seq数据,而没有整合单细胞RNA-seq(scRNA-seq)数据。此外,大多数预后模型都是由预测基因构建的,忽略了其他预测变量,如临床特征。
方法:从GEO数据库获得scRNA-seq数据,从TCGA数据库获得bulk RNA-seq数据。文中通过 LASSO 和 Cox 回归构建了一个预后模型。此外,还进行了 ESTIMATE、CIBERSORT、免疫检查点相关分析,并使用 pRRophetic 方法评估不同风险组之间的IC50来比较药物敏感性。
结果:提取14个肿瘤相关基因进行模型构建。TCGA 和三个验证队列中 1 年、3 年和 5 年总生存期预测的 AUC 几乎都高于 0.65,其中一些甚至高于 0.7,甚至 0.8。此外,校准曲线表明模型预测和完美拟合之间没有偏离。免疫相关和药物敏感性结果揭示了潜在的治疗靶点和治疗策略,可提供临床指导。
结论:整合了传统的bulk RNA-seq和scRNA-seq数据,以及预测性的临床特征,为NSCLC患者建立了预后模型。根据构建的模型,不同组的患者可以根据免疫特征和药物敏感性拟定精确和个性化的治疗方案。
疾病简介与实验设计
疾病简介
肺癌是全球癌症相关死亡的主要原因,非小细胞肺癌(NSCLC)占肺癌的85%,其中肺腺癌(LUAD)和肺鳞状细胞癌(LUSC)是最常见的亚型。近年来,越来越多的非小细胞肺癌患者有前途的治疗策略被提出并实施,包括免疫治疗和化疗药物。然而,并非所有NSCLC患者都能从这些有前途的治疗策略中受益,其中一些对免疫检查点抑制剂(如程序性死亡1(PD1)、程序性死亡配体1(PD-L1)等几乎没有反应。对治疗的反应与许多因素密切相关,例如特定基因的表达、临床特征、免疫细胞浸润等。因此,有必要在考虑基因表达和临床特征的情况下构建患者分层的预测模型。基于患者分层,我们可以找出患者对不同治疗策略的反应,并针对不同群体的患者采取合适的策略,这符合精准治疗和合理用药的原则。
通过对NSCLC预后模型开发的文献回顾,发现其中大多数集中在免疫、代谢等方面。然而,目前的研究大多只涉及 bulk RNA-seq数据,而没有整合单细胞RNA-seq(scRNA-seq)数据,忽略了细胞异质性的影响。此外,大多数预后模型都是由预测基因构建的,忽略了其他预测变量,如临床特征,包括年龄、性别、肿瘤分期等,这可能导致预后模型效率低下。
传统的bulk RNA-seq主要集中在一名患者中所有细胞的平均表达相比,scRNA-seq可以检测肿瘤细胞的细胞和分子变化。此外,由于scRNA-seq突出了肿瘤内异质性和不同的亚群,可以量化正常和肿瘤组织中免疫细胞浸润的异质组成,这是NSCLC治疗反应和预后的关键因素。
样本
如文中Table 1所示,单细胞训练集为GSE117570;bulk RNA-seq 训练集来源于 TCGA 数据库。有三个验证集:GSE42127;GSE13213;GSE157009。

研究结果
使用 scRNA-seq 数据鉴定肿瘤相关簇的标志物
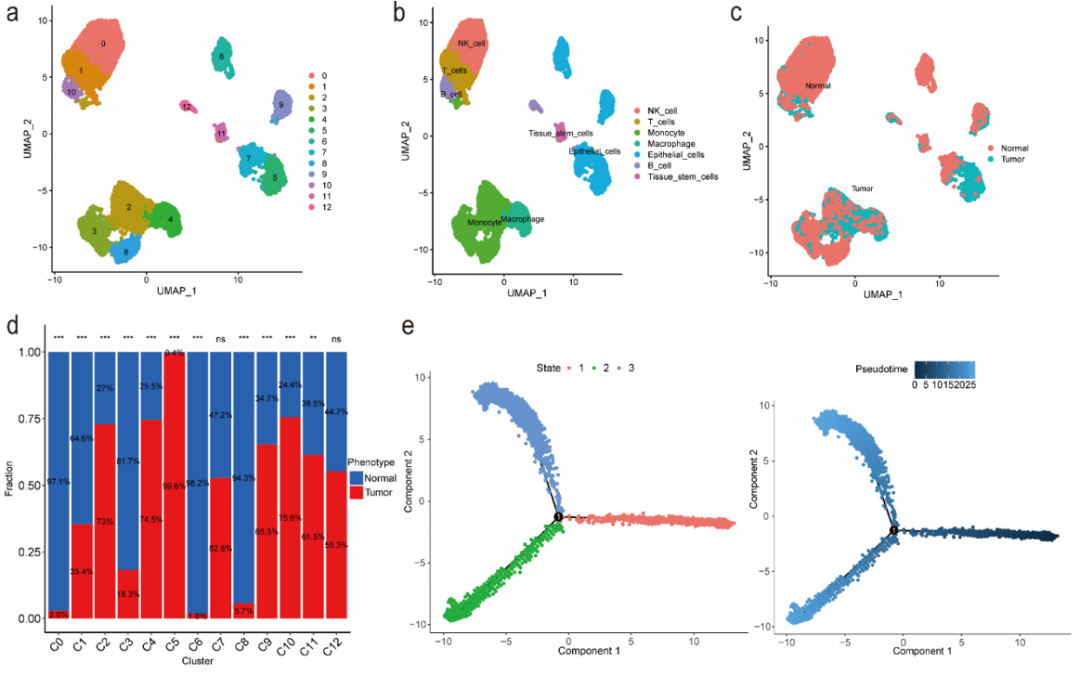
作者鉴定了 13 个簇,并进一步自动注释了 7 种细胞类型。包括 NK 细胞覆盖的簇 0;T 细胞覆盖的簇 1;单核细胞覆盖的簇 2、簇 3 和簇 8;巨噬细胞覆盖的簇 4;上皮细胞覆盖的簇 5、簇 6、簇 7 和簇 9;B 细胞覆盖的簇 10、簇 12 和组织干细胞覆盖的簇 11(图 1a, 此外,结合表型(即样本是癌症组织来源还是正常组织来源),发现肿瘤细胞主要分布在簇2、簇4、簇5和簇10,而正常细胞主要分布在簇0、簇3、簇6和簇8(图1c,1d)。
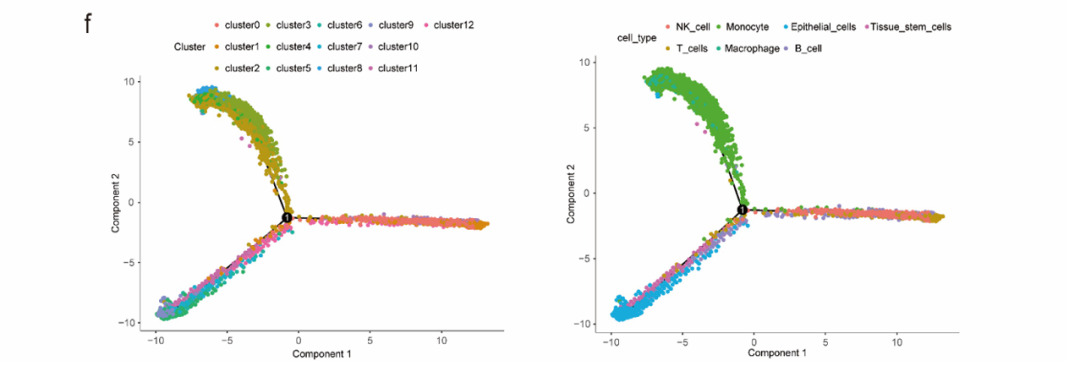
然后进行基因集变异分析(GSVA),结果表明,在上调通路中,第4簇巨噬细胞和第5簇上皮细胞富集在内质网等细胞成分相关通路中,并参与免疫效应、细胞活化和有机磷生物合成过程,而在下调通路中, 第4簇巨噬细胞富集在核糖体组成相关途径中,并参与蛋白质定位到内质网、肽代谢、病毒基因表达、翻译起始和共翻译蛋白靶向膜的过程。此外,根据细胞轨迹和伪时间分析结果,分别覆盖簇 2 和簇 4 的单核细胞和巨噬细胞仅对应状态 3,而覆盖簇 5 的上皮细胞仅对应状态 2。状态 2 和状态 3 都处于细胞轨迹发育的后期阶段(图 1e、1f)。最后,将簇2、簇4、簇5和簇10作为肿瘤相关簇,提取预后模型开发的标志物。
使用bulk RNA-seq 数据鉴定肿瘤相关模块基因
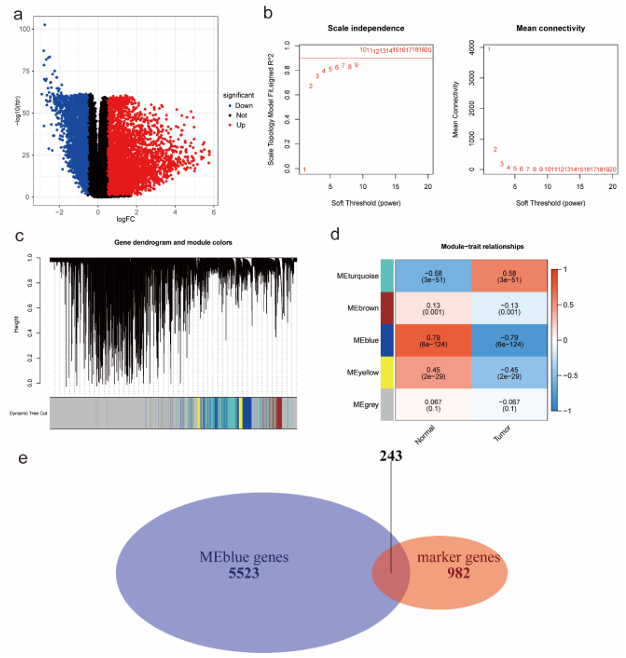
根据差异表达分析结果,有5942个上调基因和3172个下调基因。图2a为差异表达基因(DEGs)的火山图。此外,GO富集分析结果表明,DEGs主要富集在离子跨膜转运调控、通道和信号受体活性,以及细胞顶端部分的成分、突触膜、顶端质膜和转运蛋白复合物。KEGG富集分析结果显示,DEGs主要参与神经活性配体-受体相互作用和细胞因子-细胞因子受体相互作用、钙信号通路和cAMP信号通路、酒精中毒和中性粒细胞胞外陷阱的形成。最后,进行了WGCNA分析,以鉴定肿瘤相关模块基因。作者观察到,在共表达网络构建过程中,当无标度拓扑的拟合指数达到0.9时,软阈值功率 β 为10(图2b)。基于软阈值能力以及平均连锁层次聚类,最终确定了 5 个基因模块(图 2c)。表型相关性分析显示,根据相关系数和p值,“蓝色”模块与肿瘤表型的相关性最显著(图2d)。结合scRNA-seq数据中来自肿瘤相关簇的1225个标记物和bulk RNA-seq数据中来自“蓝色”模块的5766个基因,提取了243个共有基因用于预后模型构建(图2e)。
使用来自scRNA-seq数据和bulk RNA-seq数据的共同基因进行预后模型开发和验证
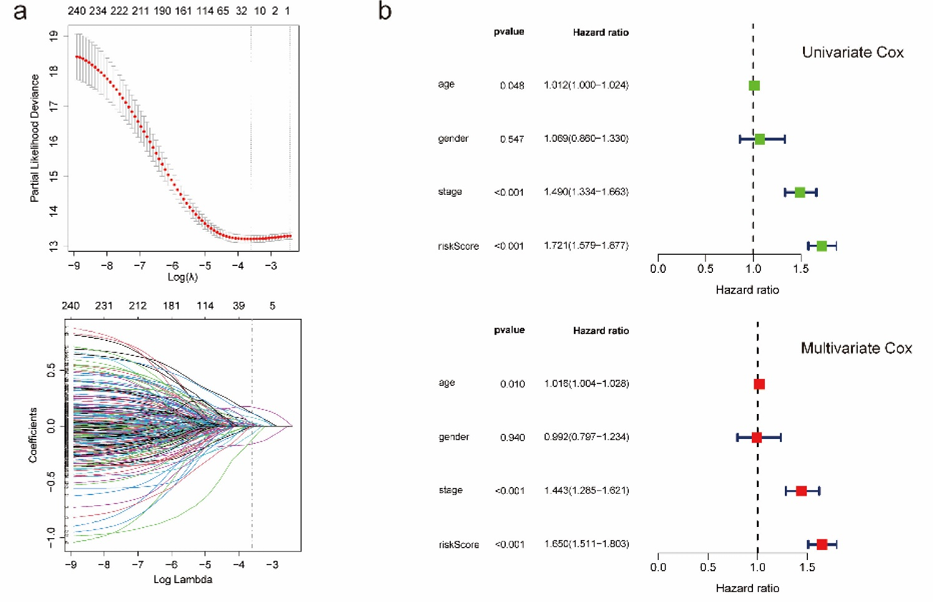
根据LASSO Cox回归分析的结果,从上述共有基因中鉴定出20个潜在的预后基因(图3a)。作者通过多因素Cox回归分析进一步提取了14个预后基因,然后构建了一个基因评分。

然后,确定了变量“基因评分”、“年龄”和“阶段”,这些变量同时在单变量和多变量Cox回归分析中与预后显着相关(图3b)。
接着,利用选定的变量来开发预后模型并计算风险评分,用于将患者分为高风险组和低风险组:
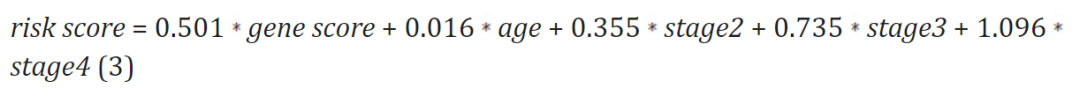
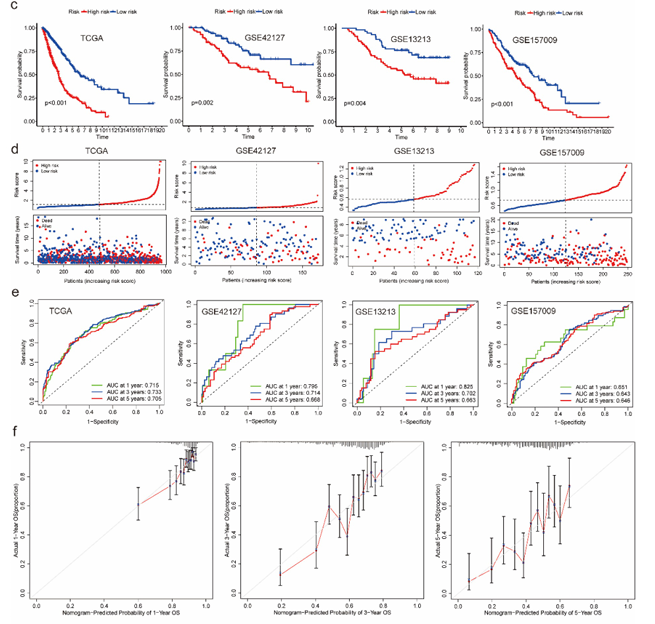
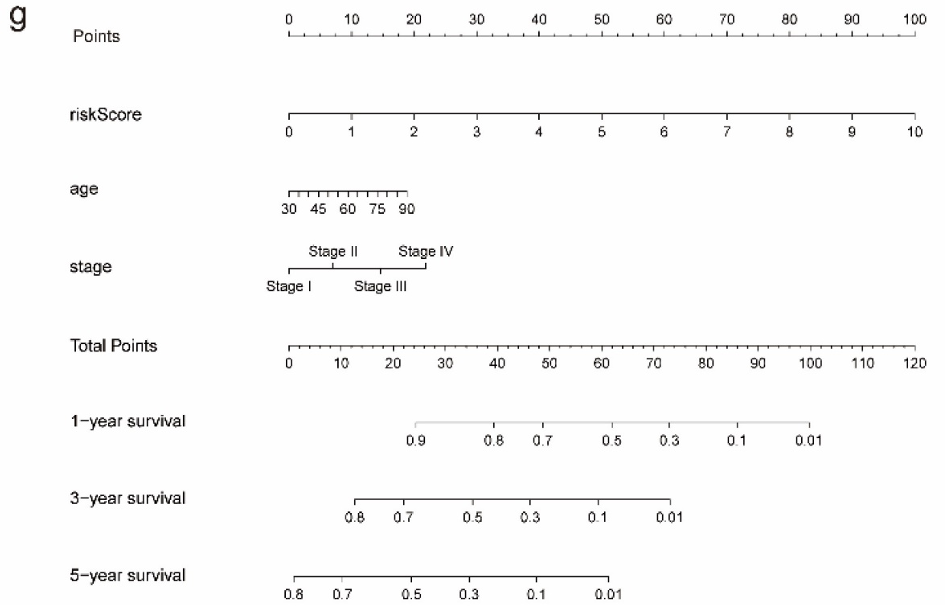
与TCGA队列以及三个验证队列中高风险组和低风险组之间的显著生存差异相比,作者发现低风险组的患者预后更好(图3c)。此外,还观察到高风险组患者与死亡的生存状态相关,而低风险组患者与存活的生存状态相关(图3d)。此外,在分类方面,预后模型在预测每个患者的总生存期(OS)方面表现出良好的性能。TCGA 队列和三个验证队列中 1 年、3 年和 5 年 OS 预测的 AUC 几乎都高于 0.65,其中一些甚至高于 0.7,甚至 0.8(图 3e)。至于校准,1年、3年和5年的曲线表明模型预测和完美拟合之间没有偏离(图3f)。为了更好地应用,作者将模型转换为列线图,方便用于预测 1 年、3 年和 5 年的 OS(图 3g)。
高风险和低风险人群的特征相关性、基因集富集分析
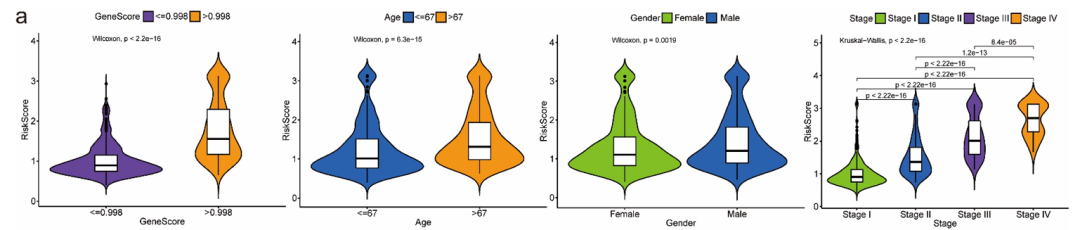
根据箱形小提琴图,发现风险评分与基因评分以及年龄、性别和肿瘤分期等临床特征显著相关。具体来说,作者分别根据基因评分的中位数和年龄的中位数将患者分为两组,观察到老年患者和基因评分较高的患者具有较高的风险评分。此外,男性患者和IV期患者的风险评分更高(图4a)。对于GO通路以及KEGG通路结果详见补充图(图S6、S7,图S8)。
高风险和低风险人群之间的免疫相关分析结果
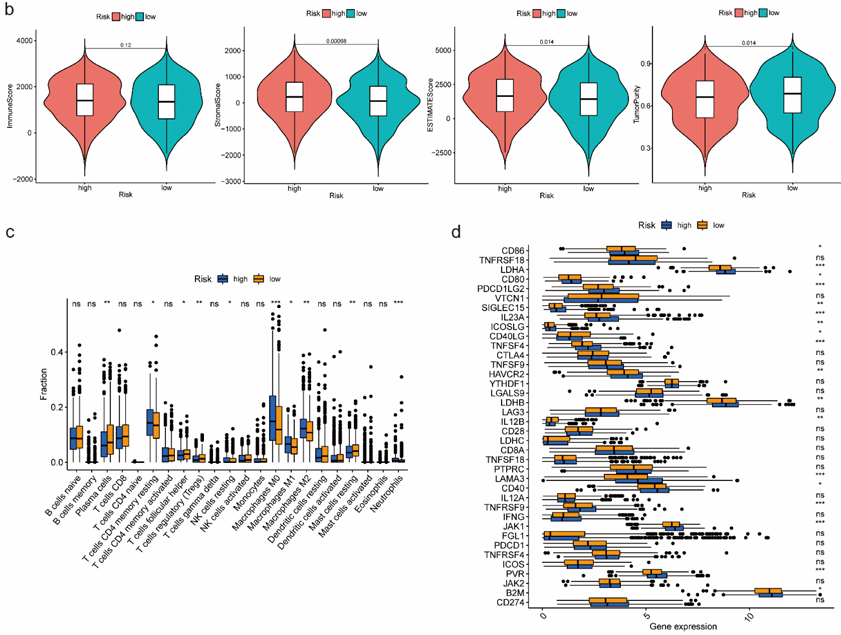
肿瘤免疫微环境(TIME)中各类免疫细胞浸润水平的估计显示,高风险组和低风险组的免疫评分差异无统计学意义。高风险组的基质评分和ESTIMATE评分较高,而低风险组的肿瘤纯度较高(图4b)。CIBERSORT分析结果表明,高风险组组织中T细胞CD4记忆静息、NK细胞静息、巨噬细胞M0、巨噬细胞M1、巨噬细胞M2和中性粒细胞的相对分数较高。而在低风险组中,浆细胞、T细胞滤泡辅助细胞、T细胞调节细胞和肥大细胞静息的相对分数较高(图4c)。通过比较38个免疫检查点相关基因的表达,发现高风险组与CD86、LDHA、CD80、PDCD1LG2、SIGLEC15、IL23A、ICOSLG、TNFSF4、HAVCR2、LDHB、LAMA3、CD40、TNFRSF9、JAK1、PVR和B2M的上调显著相关,而低风险组与CD40LG和IL12B的上调显著相关(图4d)。
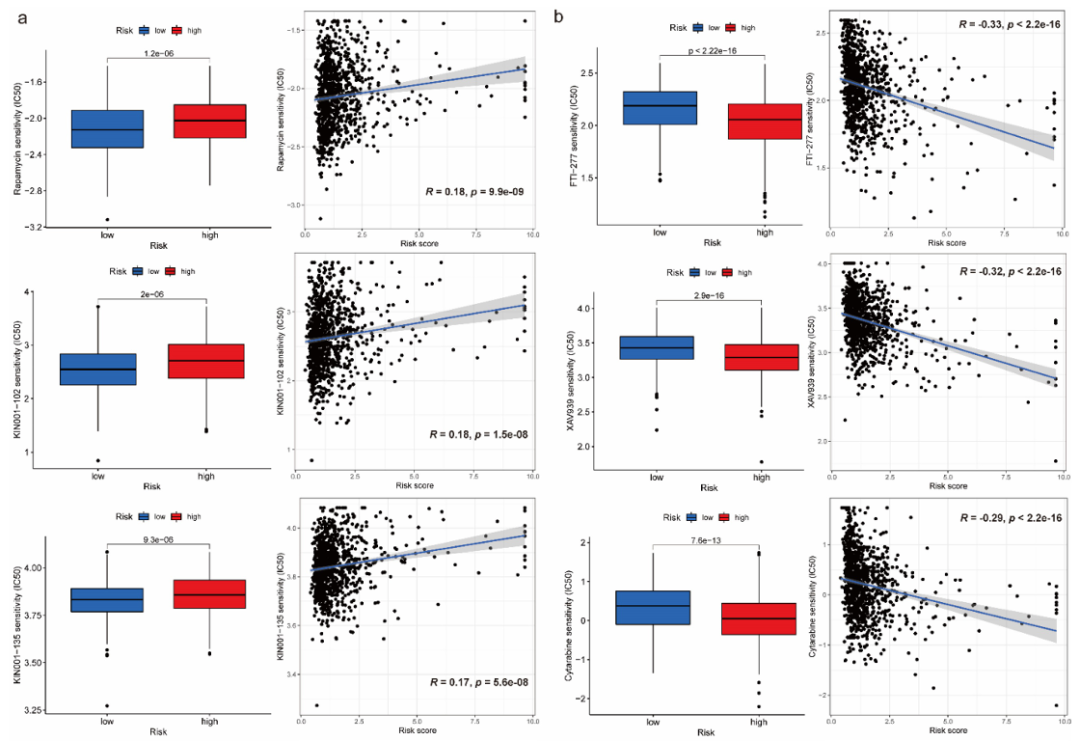
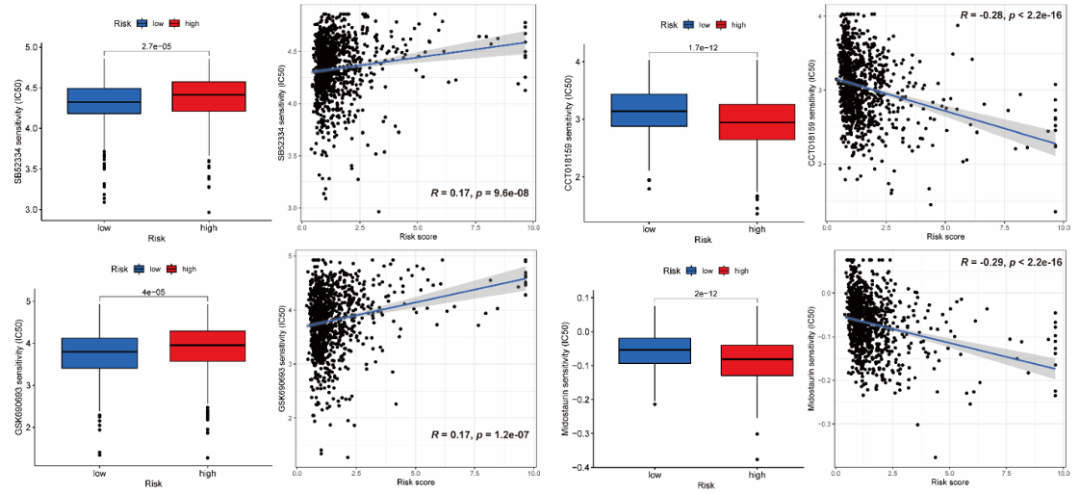
高风险组和低风险组的药敏比较
作者从GDSC 2016药物数据集中获得了102种高风险组和低风险组IC50差异显著的药物。较低的 IC50 表示对药物的反应更好。低风险组有8种较低IC50的药物,高风险组有94种IC50较低的药物。此外,根据IC50差异p值、相关值和相关p值分别提取了在低风险组的五种药物:雷帕霉素、KIN001-102、KIN001-135、SB52334、GSK690693;以及在高风险组的五种药物:FTI-277、XAV939、阿糖胞苷、CCT018159、米多斯尿素(图5a、5b)。
小结
- 首先,结合scRNA-seq数据中来自肿瘤相关簇的1225个标记物和bulk RNA-seq数据中来自“蓝色”模块的5766个基因,提取了243个共有基因用于预后模型构建。
- 然后,243个共有基因作为输入,结合LASSO Cox回归分析和多因素Cox回归分析得到14个潜在的预后基因,开发预后模型并计算风险评分,并用验证集进行验证。
- 上述预后模型的风险评分用于将患者分为高风险组和低风险组。最后,对高风险组和低风险组进行比较,分别进行了 ESTIMATE、CIBERSORT、免疫检查点,药物敏感性相关分析。
读后感想
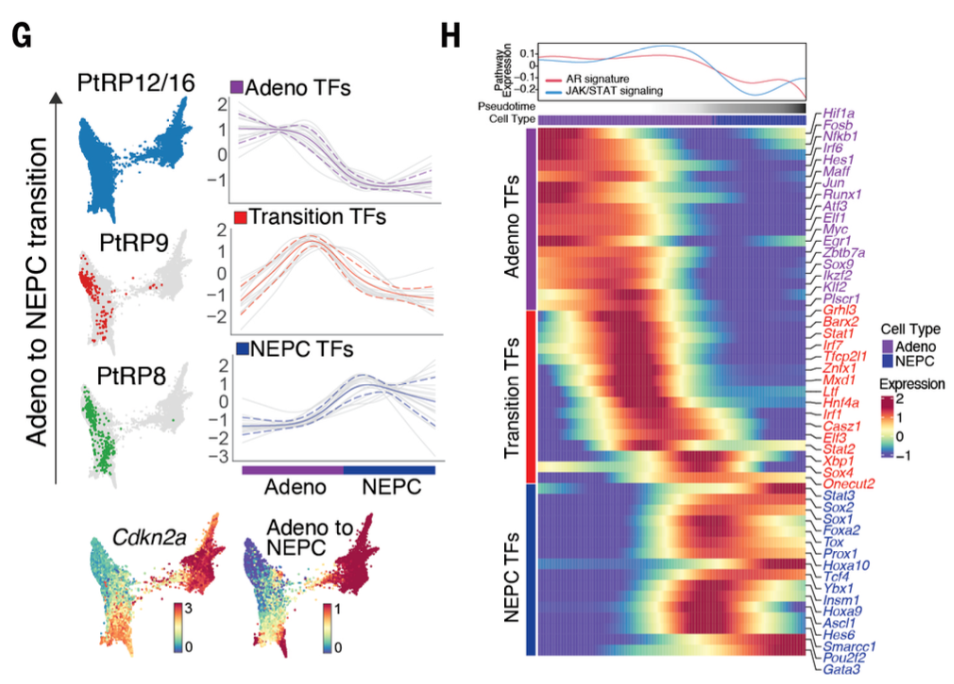
这篇文献中,关于单细胞分析的拟时序部分,分析的方法其实不是很合适。因为拟时序分析(轨迹推断)是为了找到不同细胞身份、分化或者生物过程中渐进式非同步的变化,构建出的一个动态模型。所以不适合将所有的细胞类型都进行拟时序分析,而是只对可能有分化演变过程的细胞类型之间进行分析。
比如之前在生信技能树上的一篇推文【单细胞水平的拟时序分析看肿瘤进化关系】。当确定了NEPC是由 adenocarcinoma 演变过来的, 接下来就关心 adenocarcinoma-to-NEPC transition 这个过程,所以文献中仅仅是针对 adenocarcinoma和NEPC 进行拟时序(下图所示)。这样的分析更有生物学意义哦。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-03-04,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

