慕尼黑工业大学提出TwinBooster模型,结合自监督学习和大语言模型预测分子性质
慕尼黑工业大学提出TwinBooster模型,结合自监督学习和大语言模型预测分子性质

定量构效关系(QSAR)建模是加速药物发现和开发过程的关键因素。然而,药物发现中的数据集规模往往不足以开发准确可靠的QSAR预测因子。这是因为许多关键的分析既耗时又昂贵,使得收集足够的标记样本进行QSAR建模变得困难。此外,这些数据集可能缺乏化学多样性,降低了最终数据驱动模型的泛化能力,或者可能没有报告“负面”(例如,非活性)化合物,限制了可用于训练QSAR模型的信息量。
2024年6月5日,德国慕尼黑工业大学Maximilian G. Schuh等人在Journal of Chemical Information and Modeling上发表文章Synergizing Chemical Structures and Bioassay Descriptions for Enhanced Molecular Property Prediction in Drug Discovery。

作者提出了一种结合自监督学习(SSL)、大语言模型(LLM)和梯度增强机(GBM)的分子性质预测方法TwinBooster。TwinBooster基于自监督学习架构Barlow Twins,可以将文本和分子信息结合为定量构效关系(QSAR)建模的有用特征。实验证明了TwinBooster相比于现有方法更优秀的性能,可实现准确的分子性质预测。
TwinBooster将分子性质预测任务建模为文本分析与分子相匹配的过程,它接收分子结构和文本描述两种数据模式作为输入,并预测查询分子性质。这种方法的主要优点是,它允许使用公共数据库(例如PubChem)中存在的丰富语料预训练大语言模型,然后微调模型来生成对新分子和分析的预测。TwinBooster的训练过程可以概括为三个步骤,如图1所示。首先,对大语言模型进行微调,以提高药物发现领域文本嵌入的质量。然后,训练Barlow Twins架构,使得同种化合物的化学表示和文本表示相似。最后,训练GBM分类器,根据Barlow Twins架构学习的特征来预测分子性质。
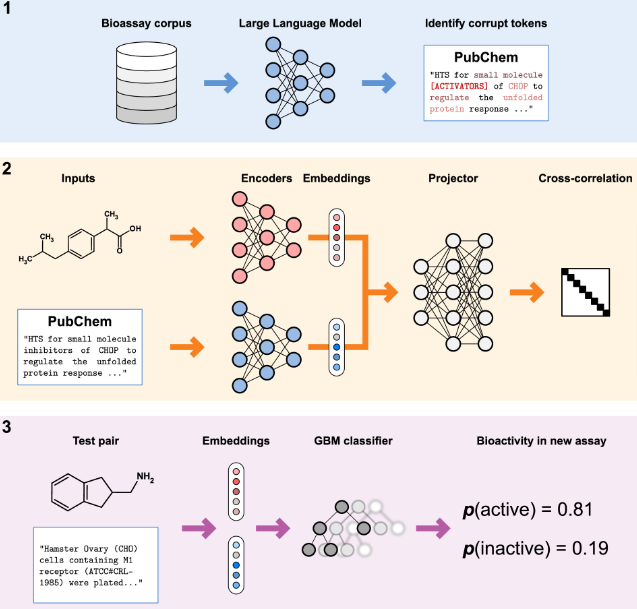
图1 TwinBooster结构图
TwinBooster由四个模块组成。(1)将化学结构转换为数值表示的分子特征化算法。在本研究中采用了扩展连接指纹(ECFP),因为其在QSAR建模中广泛流行,并且在分子性质预测研究中表现优异。(2)一个大语言模型,在本研究中使用了DeBERTA LLM架构对PubChem的大型生物测定(bioassay)语料库进行了表示学习以及微调。(3)自监督学习架构Barlow Twins,使得同种化合物的化学表示和文本表示相似。(4)一种分类算法,给定一对分子和文本特征,用于预测给定化合物是否具有生物活性。考虑到计算效率和QSAR建模的性能,本研究选择LightGBM(轻量化GBM)算法。
FS-Mol数据集是在小样本学习的情况下评估QSAR模型的基准,包括122项测定和27363种化合物。它可用于小样本或零样本场景中。前者测量模型在给定少量训练样本的情况下预测目标测定中生物活性的能力。后者评估算法预测训练期间从未观察到的测定的生物活性的能力。由于QSAR模型的性能通常随着用于训练的分析特定数据的增加而提高,因此零样本预测比小样本预测要困难得多。然而,在没有训练数据的情况下预测生物活性的能力在实践中更有用,因为它消除了事先进行任何测量的需要。
TwinBooster首先使用RDKit将FS-Mol数据集的所有分子转换为1024位,半径为2的ECFP。接着,基于生物测定的LLM文本嵌入获得标题,描述和协议作为每个分子和检测的附加表示,此分析文本信息是从PubChem中检索到的,映射到基于ChemBL的FS-Mol基准。对于TwinBooster,文本信息使用预训练的PubChemDeBERTa大语言模型在PubChem获得的综合生物测定语料库上微调。
与其他SSL技术相比,Barlow Twins的主要优势在于其新颖的目标函数,该函数测量处理不同输入表示的两个相同网络的输出之间的相互关联矩阵。在TwinBooster中,这两者分别对应于大语言模型的文本描述和查询化合物的化学信息。Barlow Twins可使不同的化合物具有与其文本描述相似的表示,同时最大限度地减少特征冗余。梯度增强机(GBM)使用Barlow Twins模型的学到的表示输入LightGBM进行训练,使用SMAC3优化LightGBM的超参数。
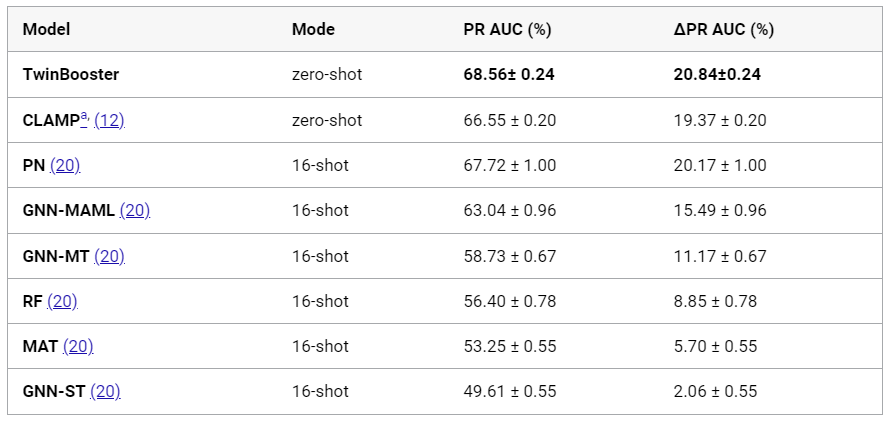
作者将TwinBooster与一些具有代表性的方法进行了比较。其中TwinBooster和CLAMP为零样本学习,其余样本为小样本学习,16-shot表示训练中有具有活性和不具有活性的样本各16个。如表1所示,将PR曲线下的面积(PR AUC)作为对比指标(越高越好),同时列出使用零样本(或小样本学习算法)所获得的PR AUC超过将所学习的表示进行随机猜测所获得的PR AUC的值,即△PR AUC。TwinBooster在零样本学习的情况下超过了其他方法在小样本学习的情况下的表现。
表1 与其他方法对比
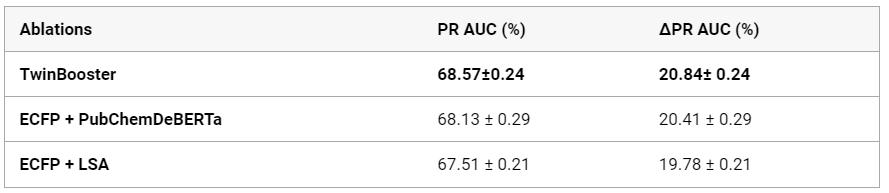
作者设计了消融实验来验证模型设计的有效性。首先使用ECFP+PubChemDeBERTa的模型,也就是去除Barlow Twins的自监督学习框架。接着使用ECFP+LSA(潜在语义分析)的模型,也就是去除Barlow Twins的自监督学习框架以及PubChemDeBERTa的大语言模型。结果如表2所示。这一结果表明,自监督学习和大语言模型有助于更好地协同文本和分子模式提供的信息,从而获得更好的分子性质预测性能。
表2 消融实验
作者还进行了案例分析。TwinBooster的一个关键应用是根据目标分析的描述和实验方案定制高通量筛选(HTS)库。作者在内质网(ER)应激背景下选择性激活C/EBP同源蛋白(CHOP)途径的化合物进行HTS分析。该案例不包括在FS-Mol的训练集中,这确保了本案例研究是对TwinBooster发现新的活性化合物的预期性能的公正评估,目的是在进行任何实际实验之前,仅依靠分析方案描述,评估TwinBooster能够在多大程度上识别出该活动中经过验证的HTS命中。TwinBooster的性能可以在图2a中直观地显示出来,也就是预测大多数HTS命中(特别是排名靠前的命中)很有可能是具有活性的。图2b通过绘制TwinBooster获得的召回曲线进一步量化其性能。在实践中,该分析描述了根据TwinBooster的预测,当选择更多的化合物进行测试时,检索到的总HTS命中的百分比。例如,在评估TwinBooster预测的前20%时,有可能识别出49%的活性化合物,进一步证明了该工具的良好优先级性能。
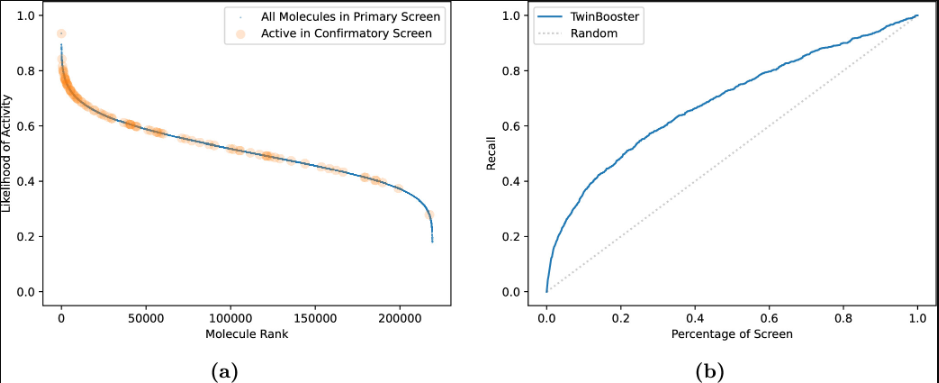
图2 案例分析
最后,作者研究了用于预训练的生物测定文本域是否对TwinBooster有“偏见”(bias)。这种“偏见”是指语言模型更可能关注到频繁命中的样本。在HTS中,频繁命中是指在多次试验中表现出活性的化合物,通常是由于非特异性相互作用而不是真正的生物学相关性,也就是由于自体荧光,氧化还原循环,胶体聚集等因素产生阳性,而不是由于期望的生化活性所产生。最后,不理想的多药理学也会使HTS数据中的靶点选择过程复杂化,例如泛激酶抑制剂的情况。
为了评估TwinBooster是否能预测频繁命中,所有分子都使用HitDexter 3进行处理,根据HitDexter 3在TwinBooster的预测和频繁击球概率之间进行相关性分析。平均Pearson相关系数为0.0359,表明没有相关性。具体而言,由于FS-Mol训练数据中最大的注释蛋白类由激酶组成,因此无论提供的生物测定描述如何,TwinBooster理论上都可以通过专注于识别泛激酶抑制剂,在该基准上获得良好的预测性能。为了TwinBooster的性能不是由于识别泛激酶抑制剂而得到,作者在训练数据中根据之前的研究筛选了73种潜在的泛激酶抑制剂。如果TwinBooster忽略了提供的文本模态,那么无论文本模态如何,这些分子都应该始终被预测为活动的。因此,作者研究了当用其他生物测定描述替换原始文本输入时,预测是否会发生变化。图3显示,TwinBooster的预测活性似然概率(likelihood of activity)反映了文本域的变化,因为泛激酶抑制剂在与非激酶试验配对时被归类为无活性,而在激酶相关试验中被预测为广泛活性。这表明TwinBooster的预测是可靠的,而不是由于对数据的“偏见”所产生。
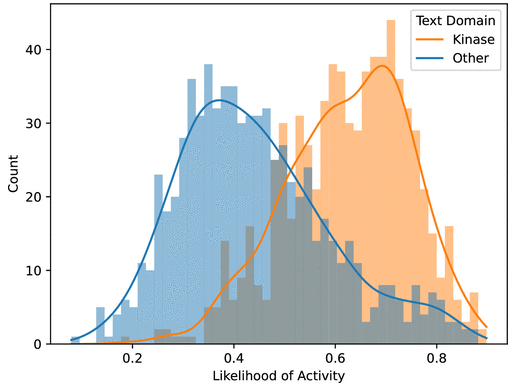
图3 预测活性似然概率的比较
本文提出了TwinBooster,通过集成大语言模型、Barlow Twins自监督学习架构和LightGBM,TwinBooster框架可以在数据稀缺的情况下进行分子特性预测。该方法在FS-Mol基准上取得了优异的性能,优于现有的QSAR模型,这一结果与消融研究相结合,证明了结合自监督学习和大语言模型对分子性质预测性能的增益。当TwinBooster被应用于高通量筛选的案例研究时,模型可以有效地识别活性化合物。TwinBooster可以帮助加速新型药物发现,使研究人员能够专注于筛选库中最相关的化合物,从而减少药物发现中与命中识别相关的时间和成本。
参考文献
Schuh et al. Synergizing Chemical Structures and Bioassay Descriptions for Enhanced Molecular Property Prediction in Drug Discovery. J Chem Inf Model. 2024
