基因的功能推断之大队列高低分组后差异分析然后功能富集
基因的功能推断之大队列高低分组后差异分析然后功能富集
前面我们强调了,基因功能推断的数据分析的重要性 ,接下来我们就演示一下常见的4种方法来进行基因的功能推断,首先是大队列高低分组后差异分析然后功能富集。
我们以2023的这个肾癌数据挖掘文章为例,标题所:《SLC17A9-PTHLH-EMT axis promotes proliferation and invasion of clear renal cell carcinoma》,研究者们首先很好的说明了SLC17A9这个基因在肾癌里面所一个非常典型的癌基因:
- 首先是在癌症样品里面相对于正常来说所高表达量的:SLC17A9 might serve as an unfa- vorable marker for ccRCC and be positively associated with poorer clinical prognosis.
- 然后 SLC17A9 was an unfavorable marker for ccRCC :patients in the group having high SLC17A9 expression levels were associated with poor OS and DFS
如下所示:
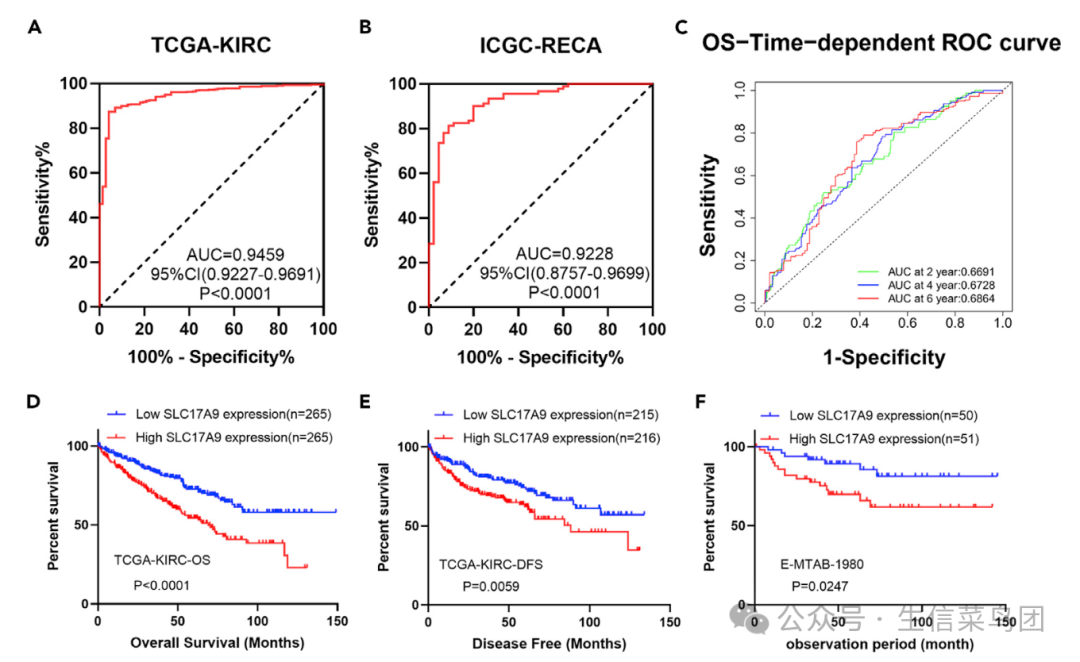
非常典型的癌基因
但是呢,Although SLC17A9 was identified as a biomarker for renal cancer, its role in tumor growth was unknown.
这个时候就可以使用tcga数据库资源啦, 里面的肾癌转录组表达量矩阵就是大队列样品,仅仅是肿瘤样品就有522个,很简单的下载counts矩阵然后就可以差异分析 ,作者给出来的方法学所:
All 522 samples of KIRC were initially divided into two groups according to the median expression of SLC17A9, and the ‘‘limma’’ package was then used to obtain the differential expressed genes (DEGs) related to SLC17A9, with adjusted p < 0.05 and log2|FC| > 1.
差异分析完成后,每个基因就有一个变化倍数,它就可以用来作为排序,然后就可以进行gsea分析,这个时候找到那些排名靠前的生物学功能就可以作为我们的目标基因的功能推断结果啦 :
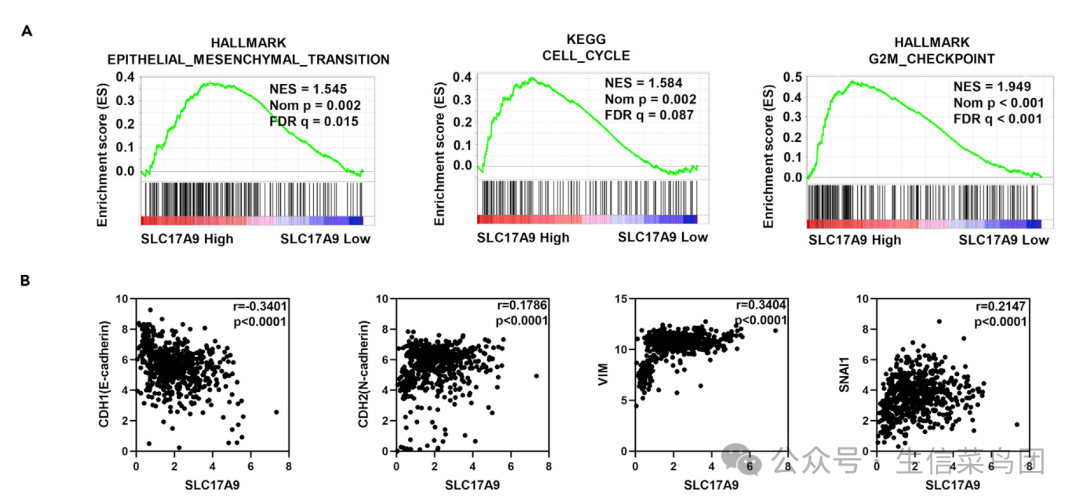
前人已经证明过 SLC17A9 could positively regulate cell proliferation ,所以可以侧面验证这个推断所合理的,然后作者又聚焦了EMT这个明星通路,所以看了看 SLC17A9 and four EMT markers (namely, CDH1, CDH2, SNAI1, and VIM) 的具体的表达量相关性散点图。
当然了,因为前面的差异分析有统计学阈值,所以也可以确定下 differentially expressed genes (DEGs) between the SLC17A9 high expression group and low expres- sions group,然后对上下调基因分别去做超几何分布检验看生物学功能情况。
下面是一个示例代码:
# exp 是一个表达量矩阵
# 然后this_gene 是 目标基因
Group = ifelse(exp[ this_gene ,] > median(exp[ this_gene ,]),'high','low')
table(Group)
# 需要把Group转换成因子,并设置参考水平,指定levels,对照组在前,处理组在后
Group = factor(Group,levels = c("low","high"))
Group
table(Group)
#需要表达矩阵和Group,不需要改
library(limma)
design=model.matrix(~Group)
fit=lmFit(exp,design)
fit=eBayes(fit)
deg=topTable(fit,coef=2,number = Inf)
head(deg)
#为deg数据框添加几列
#1.加probe_id列,把行名变成一列
library(dplyr)
deg <- mutate(deg,symbol=rownames(deg))
nrow(deg)
head(deg)
#3.加change列,标记上下调基因
logFC_t=1
P.Value_t = 0.05
k1 = (deg$P.Value < P.Value_t)&(deg$logFC < -logFC_t)
k2 = (deg$P.Value < P.Value_t)&(deg$logFC > logFC_t)
deg <- mutate(deg,change = ifelse(k1,"down",ifelse(k2,"up","stable")))
table(deg$change)
#4.加ENTREZID列,用于富集分析(symbol转entrezid,然后inner_join)
# 这里
library(clusterProfiler)
library(org.Hs.eg.db)
s2e <- bitr(deg$symbol,
fromType = "SYMBOL",
toType = "ENTREZID",
OrgDb = org.Hs.eg.db)#人类
# # #其他物种http://bioconductor.org/packages/release/BiocViews.html#___OrgDb
dim(deg)
deg <- inner_join(deg,s2e,by=c("symbol"="SYMBOL"))
dim(deg)
head(deg)
length(unique(deg$symbol))
save(Group,deg,logFC_t,P.Value_t,this_gene,
file = paste0(this_gene,"step4output.Rdata"))
