三篇Nature子刊谱写分子世界模型三部曲|ImageMol,VideoMol,SketchMol
三篇Nature子刊谱写分子世界模型三部曲|ImageMol,VideoMol,SketchMol

随着人工智能从大语言模型(LLM)向世界模型(world model)的进一步探索,基于分子图像的“分子世界模型”,正成为革新药物研发流程的关键引擎。近几年,湖南大学DrugAI实验室提出了分子图像系列世界模型ImageMol(NMI 2022),Video Mol(NC 2024),SketchMol(NMI 2025),以新颖的分子视觉思路为药物发现带来了革命性突破。
2022年11月17日发表的《Accurate prediction of molecular properties and drug targets using a self-supervised image representation learning framework》中,研究团队率先将分子图像应用于药物靶标与性质预测,提出了“ImageMol”技术。它突破传统的分子指纹或图结构方式,创新性地将分子结构转化为“图像”进行自监督深度学习。通过以像素方式捕捉分子局部与整体特征,ImageMol显著提升了溶解度、毒性、药代动力学等性质以及潜在药物靶点的预测准确度,并在大规模无标注数据上的预训练中展现出高效可行性。借助ImageMol,研究人员成功锁定多种抗病毒及中枢神经相关候选化合物,大幅缩短了从筛选到初步验证的研发周期。

2024年11月8日发表的《A molecular video-derived foundation model for scientific drug discovery》则引入了更具创新性的“VideoMol”理念,借助“三维动态分子视频”来捕捉分子的动态变化。与传统静态图像相比,VideoMol以每分子可达60帧以上的动态图像形式,将人工智能的分析能力从平面(2D)扩展至3D+时间维度(4D)。通过自监督视频表征学习策略,它显著提升了针对激酶、GPCR等蛋白靶点的预测准确度,并能更好地解析分子与蛋白之间的作用机理,为动态结构分析与精准成药提供了新思路。

在此基础上,2025年2月13日发表的《Image-based generation for molecule design with SketchMol》更进一步,将研究重点转向“分子生成”,以视觉绘制手段构建或修饰新的分子结构。通过基于扩散模型与强化学习的像素级重构,SketchMol能在保持分子合理性的同时定向修改分子骨架与官能团,对多重属性进行同步优化。既可从零开始“绘制”新结构,也可对已有先导化合物进行局部修饰与优化,在分子世界模型领域首次实现了从“理解”到“设计”的跨越。

分子表征和分子图像
现代药物研发和分子模拟中,如何表征分子的内部结构与外部性质是核心挑战之一。传统方法通常采用基于文本字符串的SMILES表示或者化学指纹、分子图结构等方式,来编码分子中的原子类型与键连接关系。近年来,随着计算机视觉与人工智能的融合,“分子图像”这一新兴表征形式开始受到广泛关注。它将分子结构“拍照”或“渲染”成二维或三维的图像,将原子及其空间布局以视觉方式直接呈现。相比纯文字或图结构表示,分子图像能够更直观地捕捉分子整体形态与局部微观特征,兼具可视化与可计算性。在此基础上,研究者们可采用深度学习尤其是自监督或生成模型来挖掘更丰富的化学与药理学信息,为分子性质预测、分子生成与药物靶点识别等环节带来新的可能性。
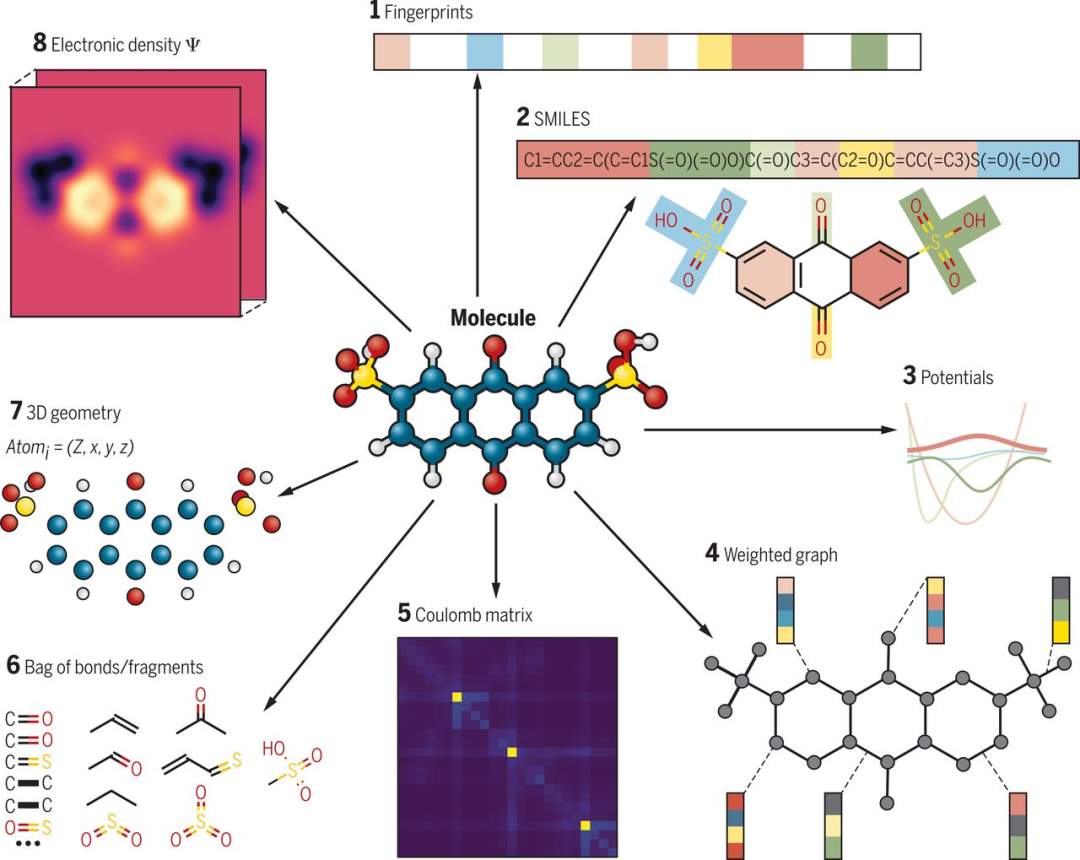
ImageMol:基于分子图像的自监督学习,提高药物性质与靶标预测精度
传统的药物性质和靶点预测,多依赖分子指纹或图结构等表征。2022年首次提出的“ImageMol”技术,摆脱了单纯依赖文本字符串或化学指纹的模式,创新性地将分子结构转化为“图像”进行表征,并结合自监督深度学习框架进行训练。
核心亮点
- 以像素方式捕捉分子局部与整体特征,避免过度依赖化学先验;
- 有效提高药物性质(溶解度、毒性、药代动力学等)与潜在靶点的预测准确度;
- 验证了分子图像在大规模无标注数据上进行预训练的可行性与高效性。
通过ImageMol,研究人员成功发现若干抗病毒及中枢神经相关候选化合物,大幅缩短了从筛选到初步验证的研发时间,展现了分子图像在药物发现中的实用潜力。
VideoMol:面向三维分子视频的基础模型,开启更深层药物机制探索
在分子图像应用获得成功后,学界进一步提出了“分子视频”概念,以捕捉分子的三维动态变化。由此诞生的“VideoMol”模型,将静态图像升级为动态视频,让人工智能可以“观看”到分子构象的时序变化。
核心亮点
- 针对分子构象采样,生成含有60帧甚至更多帧的三维动态视频;
- 利用自监督视频表征学习策略,提升对分子与靶点结合模式的理解;
- 取得了在激酶、GPCR等多种蛋白靶点预测上的高准确度,并能更好解释分子与蛋白之间的作用机理。
VideoMol的出现,为动态结构分析与精准成药提供了新思路。研究人员可基于分子视频挑选更符合真实生物环境的先导化合物,从而提高后续实验转化的成功率。
SketchMol:融合图像生成与化学智慧的分子设计新框架
在基于分子图像的研究中,先后出现了针对药物性质/靶点预测的ImageMol、针对三维动态结构分析的VideoMol等方法。它们的主要任务是“理解”和“鉴别”分子结构。而SketchMol的出现,则将分子图像从“识别”进一步拓展到“生成与设计”,为小分子的新颖结构创制和已有先导化合物的修饰带来突破性思路。
研究动机
目前的分子生成研究,多依赖序列(SMILES)或图结构(Graph)为基础的深度学习模型,常见方法如基于语言模型或图生成模型。这些方法在捕捉局部化学信息(如官能团、键类型等)方面有较好表现,但对于分子整体拓扑结构、环体系、分支等“全局布局”的把控相对有限。
方法框架
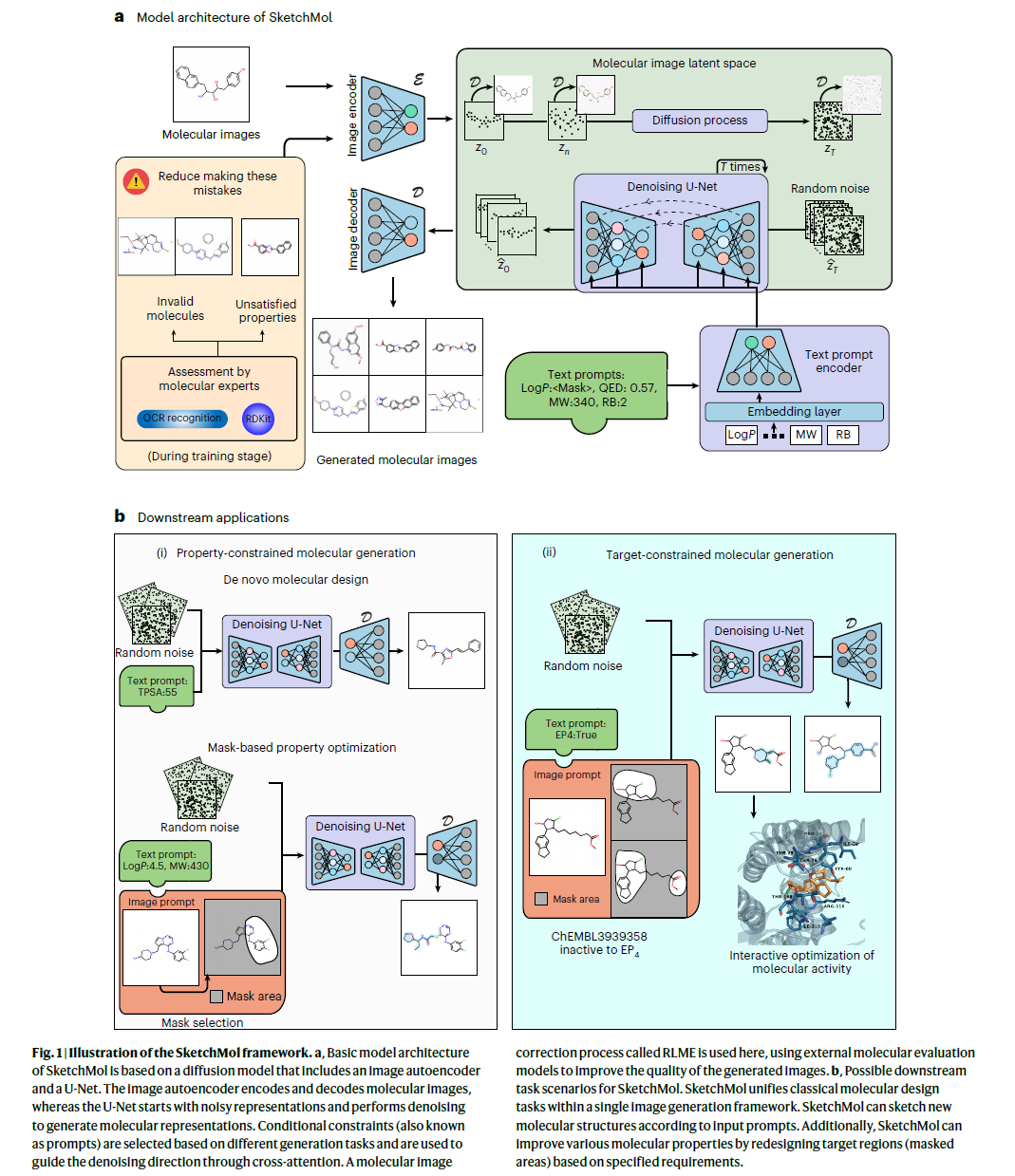
分子图像表示
与ImageMol和VideoMol类似,SketchMol也基于分子可视化渲染,将其转换成二维图像进行处理。
图像编码器与解码器
采用一个专门的图像自编码器(Autoencoder)或类似扩散/Transformer结构来学习分子的“潜在表示”,将像素级信息压缩至潜在空间并在解码端重建出分子图像。
扩散模型
扩散过程:在图像领域,扩散模型通过将初始图像逐步添加噪声并学习逆向去噪过程,最后得到清晰的图像。在SketchMol中,这一过程对应“从随机噪声起步生成结构合理的分子图像”。
强化学习校正:单纯依赖扩散模型可能会忽略化学合理性或属性约束。为此,SketchMol通过与外部的分子专业评估交互来对生成过程进行反馈式修正。例如,若模型生成的分子违反化学规则或无法达到既定物理化学属性,则调整模型参数进行校正,从而逐步逼近有效分子结构。
多属性约束与可控生成
可控条件输入:用户可指定多个物理化学指标(如LogP、分子量MW、极性表面积TPSA、氢键供体/受体数目等),SketchMol将其整合为“条件向量”或“文本提示”,指导扩散模型与解码器按要求生成满足这些指标的分子图像。
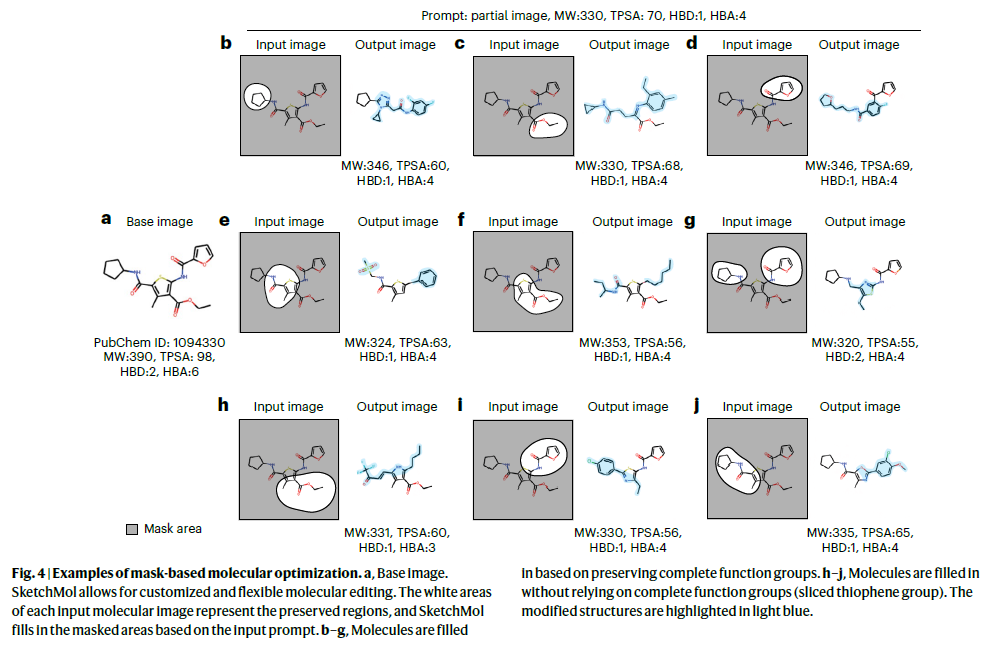
定向修改与inpainting:在已有分子骨架的图像上,可通过“Mask(遮盖)”局部区域,让模型自动填补/修饰该区域,并满足用户期望的属性变化。例如,针对先导化合物增加疏水基团、减少毒性官能团等。这样可以定向地只改变一部分结构,而不破坏其他功能位点。
主要实验结果
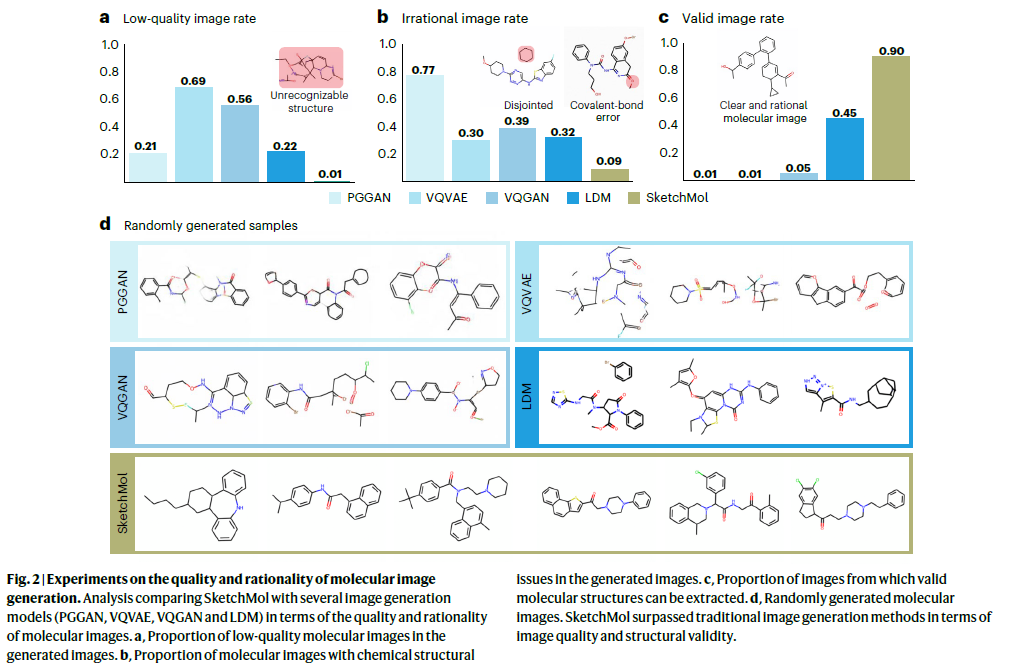
有效且合理的分子图像生成
经多轮强化校正后,SketchMol生成的分子中约90%以上可以被解析成有效、合理的化学结构(有效率远高于早期图像生成方法)。
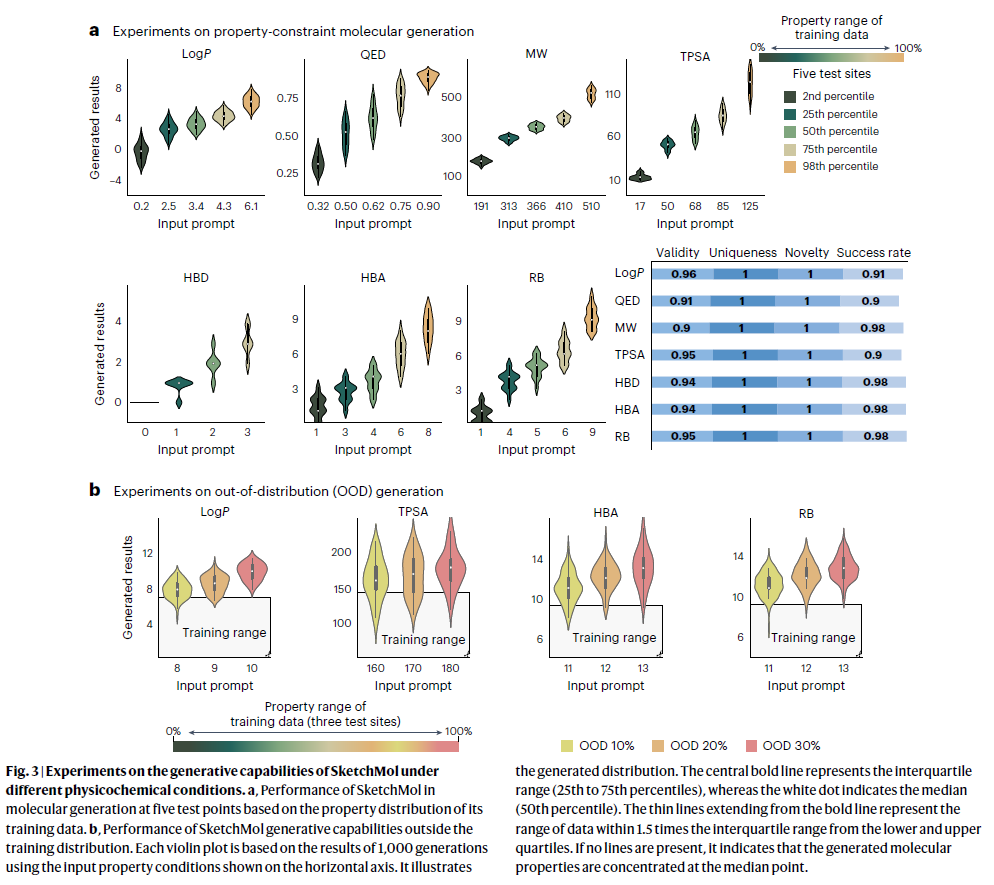
单属性与多属性的可控生成
- 当用户只给定一个约束(例如LogP=3.5)时,SketchMol能生成满足这一性质、且与训练集分子结构不同的全新分子;
- 当同时设定多达七个属性时(多属性联合约束),在一定范围内SketchMol也能保证高成功率,且有效保证结构多样性。
定向分子修饰与优化
面向已有先导分子,用户可在图像上标定希望修改的区域、指定属性提升或削弱的目标值。SketchMol在修饰完成后,常能得到更优的分子性质(如更高效、更低毒性等),并保持原有分子骨架的关键片段。
总结与展望
回顾自2022年的ImageMol到2024年的VideoMol,再到2025年的SketchMol,分子世界模型从静态表征到动态理解,再到可视化生成设计,正经历全面演进。如今,这一领域不仅在药物筛选与性质预测上成果斐然,更有望借助动态视频与可视化生成手段,为从头设计到精准研发带来颠覆式变革。随着技术的不断成熟与更多研究团队的加入,“看见分子、理解分子、设计分子”的未来蓝图将逐步照进现实,全面提升药物研发效率,助力精准医疗迈向崭新时代。分子世界模型的未来,值得我们共同期待!
参考资料
- Sanchez-Lengeling, Benjamin, and Alán Aspuru-Guzik. "Inverse molecular design using machine learning: Generative models for matter engineering." Science 361, no. 6400 (2018): 360-365.
- Zeng, Xiangxiang, Hongxin Xiang, Linhui Yu, Jianmin Wang, Kenli Li, Ruth Nussinov, and Feixiong Cheng. "Accurate prediction of molecular properties and drug targets using a self-supervised image representation learning framework." Nature Machine Intelligence 4, no. 11 (2022): 1004-1016.
- Xiang, Hongxin, Li Zeng, Linlin Hou, Kenli Li, Zhimin Fu, Yunguang Qiu, Ruth Nussinov et al. "A molecular video-derived foundation model for scientific drug discovery." Nature Communications 15, no. 1 (2024): 9696.
- Wang, Zixu, Yangyang Chen, Pengsen Ma, Zhou Yu, Jianmin Wang, Yuansheng Liu, Xiucai Ye, Tetsuya Sakurai, and Xiangxiang Zeng. "Image-based generation for molecule design with SketchMol." Nature Machine Intelligence (2025): 1-12.
--------- End ---------
