专题 | David Baker:蛋白质设计的先驱
专题 | David Baker:蛋白质设计的先驱

DrugOne
发布于 2025-11-17 20:35:40
发布于 2025-11-17 20:35:40
2024年10月9日,久负盛名的蛋白质设计领域巨擘David Baker教授同AlphaFold2开发者、DeepMind的Demis Hassabis和John M. Jumper共同获得了2024年诺贝尔化学奖。本专题旨在回顾David Baker教授的早期研究历程,以向广大读者介绍蛋白质结构预测与设计的原理和相关科学思想的形成。由于笔者水平有限,以下内容难免有疏漏错误之处,亦请广大读者批评指正。
蛋白质折叠的热力学假说和动力学控制证据
1972年诺贝尔化学奖得主Christian Anfinsen在获奖感言中描述了久负盛名的蛋白质折叠热力学假说(Thermodynamic Hypothesis):天然蛋白质在正常生理环境(溶剂、pH、离子强度、金属或其它辅因子、温度)下的三维结构是整个系统Gibbs自由能最低的结构,即给定环境下的蛋白天然构象完全由氨基酸序列决定[1]。核糖核酸酶P的变性和自主复性、小蛋白的折叠和展开反应达到表观平衡等实验事实有利地支持了热力学假说。然而,上个世纪90年代以来,一些实验数据仅表明天然状态是构象空间邻域内能量最低的状态,这里的邻域包括动力学可访问的状态。显然,几乎不可能通过实验手段访问该邻域之外的构象,因此热力学假说在某种意义上无法被证伪。
1968年,Cyrus Levinthal指出蛋白质正确折叠所需的时间仅为详尽构象搜索所需时间的极小一部分[2],不难想象在折叠过程中,实际上只能对多肽链所有可能构象中的一小部分进行采样,这些构象可视为动力学途径。考虑到构象空间近乎无穷大的规模,没有特别的理由认为天然状态的低能量构象即是全局的能量最小值:构象空间中完全可能存在动力学上无法访问、但热力学上更稳定的状态。如示意图1所示,倘若考虑一维的自由能截面,图1a展示了一种单一的全局最小值,可以从势能面的任何一点访问,即折叠反应的结果与起始构型无关,折叠过程处于热力学控制之下;相比之下,图1b的自由能截面则更加复杂,倘若多个能量极小值之间的势垒(barrier)足够高,则反应结果将在很大程度上取决于起点,实验测定的天然状态可能对应于局部或全局极小值,这种对初始条件的依赖性意味着折叠反应同样受动力学控制[3]。
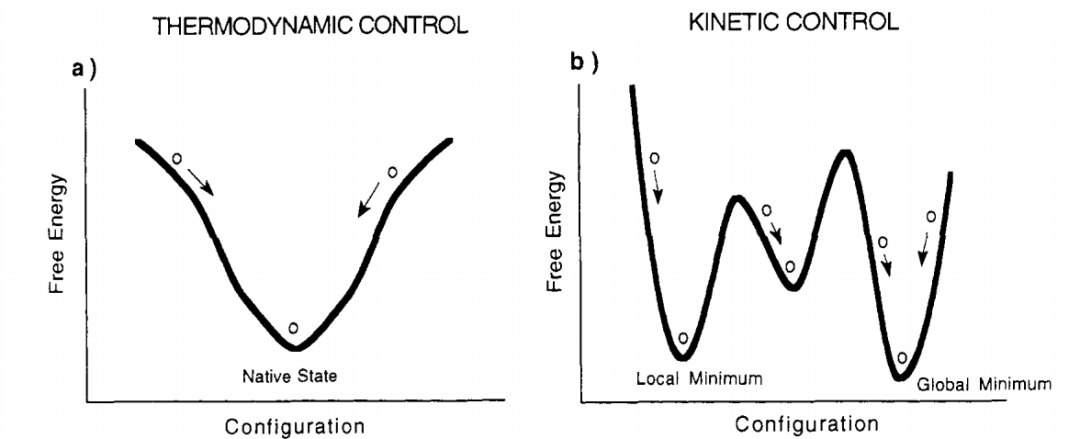
图1:简化的折叠势能曲线[3]
1992年,David Baker与UCSF的博士后合作导师David Agard研究了α-裂解蛋白酶(aLP)的折叠[4]。aLP携带一个166AAs的前体pro结构域,团队此前研究发现,在高浓度胍中变性的aLP蛋白能在透析去除胍之后恢复酶活性,且需要pro结构域的存在。考虑到pro区域能够强烈抑制天然酶的活性,作者认为pro区域应该在折叠途径的后期步骤中起作用。据此作者推断并验证了pro区域不存在时,变性剂的去除可以捕获折叠中间体。实验结果表明这种折叠中间体state I具有更大的半径和类似天然状态的二级结构,但几乎没有三级结构,能在缓冲液中稳定数月之久,且在加入pro区域后仍能迅速折叠并回复酶活性。由于pro区域能够与折叠产物(即天然状态)相互作用,这表明其促进折叠的机理是直接增加正向折叠反应的速率,而与分子伴侣降低聚集反应速率的机理不同。这项研究与同时期的其他几个案例共同对热力学假说的普遍性提出了质疑[3],表明动力学可及性也是蛋白质折叠的重要因素。
蛋白质折叠速率对序列变化具有稳健性
在华盛顿大学建立课题组后,David Baker希望继续深入研究折叠速率这一动力学问题。他首先推测对于给定的蛋白质折叠,很可能存在折叠速率范围非常广的不同序列。为了研究序列如何决定折叠速率和机制,David Baker重点关注小尺寸、自主折叠的蛋白结构域。他们首先开发了一种噬菌体展示技术[5],用于研究来源于大消化链球菌protein L上62AAs的IgG结合域,该域的正确折叠是其结合IgG所必需的。David Baker发现在所选择的14个诱变位点中,多达9个残基替换的变体仍能有效折叠[5-6]。随后这一策略被应用于另一种小型结构域SH3,惊讶地发现保留折叠能力的新序列甚至可以几乎完全由五种氨基酸(I、K、E、A、G)组成。尽管这些随机变体在热力学上往往比天然序列更不稳定,但它们的折叠速率有时比天然序列更高[6]。这些结果令David Baker意识到:氨基酸序列在进化过程中并未针对快速折叠进行广泛的优化,而稳定天然序列所形成的相互作用更可能引入了一种“漏斗”(Funnel)样的自由能景观,从而引导多肽链折叠至合适的结构[7]。
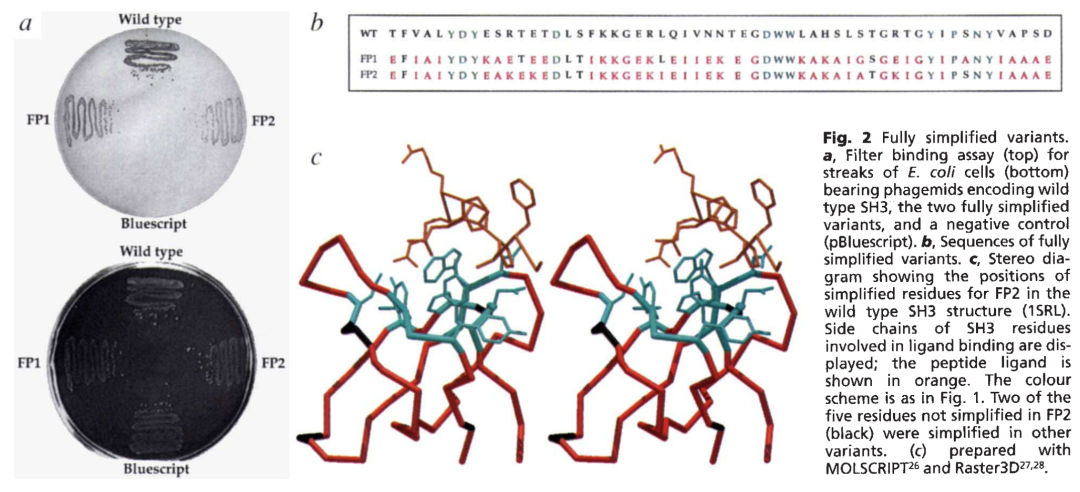
图2:通过简化的氨基酸字母表保留SH3结构域的折叠特性[6]
上述实验结果与David Baker的假设相悖,即蛋白质序列似乎不能决定其折叠动力学速率。因此,David Baker尝试了一种替代方案:考虑蛋白质折叠过程在热力学上是吸引性相互作用(attractive native interactions)形成和多肽链构象熵损失(chain configurational entropy)之间的权衡,那么折叠自由能能垒高度的决定因素是折叠早期吸引性相互作用能够补偿熵损失的程度。现在我们考虑序列上相距很远的残基,倘若这些残基形成有利相互作用,将会大幅减少中间肽段的可能构型,也即产生更大的熵损失;相反,序列上邻近残基的相互作用可以相对容易地形成,不需要极大地限制多肽链构象。因此David Baker推断:那些主要通过序列邻近残基形成相互作用的蛋白可能具有更快的折叠速率[8]。
尽管已有大量理论研究表明蛋白质的尺寸、稳定性和拓扑结构对其折叠速率有显著影响,然而在实验上严格证明可能非常困难。天然状态的尺寸和稳定性(dG)很容易测定,但拓扑结构的复杂性在数值上很难量化。为此,David Baker引入了relative contact order (相对接触序,CO)这一概念,作为拓扑复杂性的衡量标准,以反映序列上邻近的局部接触(local contact)和序列上远离的非局部接触(non-local contact)对蛋白天然结构的重要性[8]。contact order定义为所有接触残基对(residue pairs)之间的平均序列距离。
为了论证上述推测,David Baker对一小组非同源、单结构域蛋白的折叠动力学进行了统计。结果显示与过渡态势垒高度成正比的再折叠速率常数(refolding rate, k)的对数与CO密切相关,r = 0.81。相反,蛋白质的尺寸或稳定性与其折叠速率之间的相关性极弱或不存在。
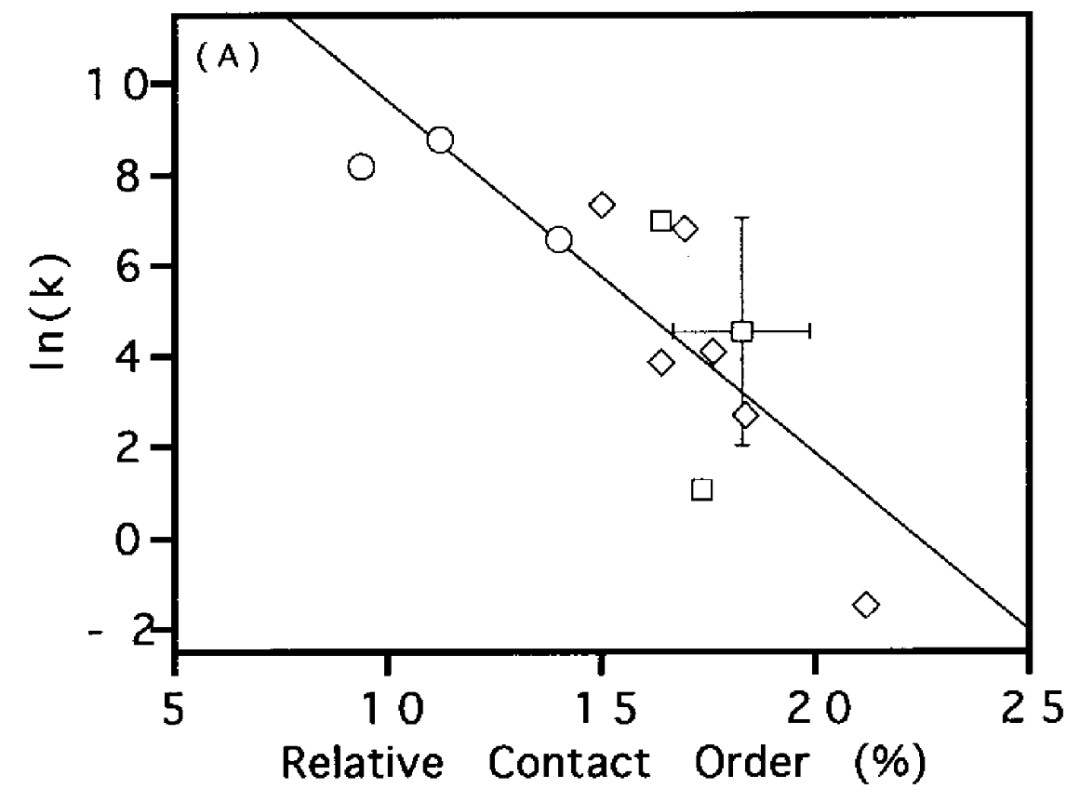
图3:contact order与ln(k)之间具有很好的线性关系[8]
CO概念的提出表明折叠过渡态的相对自由能对多肽链长度的依赖性远小于对拓扑结构的依赖性,这与成核-凝聚模型一致(nucleation-condensation model),认为折叠的限速步骤是形成一种近天然结构的区域,即“折叠核”(folding nucleus);随后折叠核起到模板样作用,快速凝聚并组织其周围的结构[9]。显然,这种机制极大地降低了折叠所需采样的构象数,在一定程度上解释了Levinthal悖论。
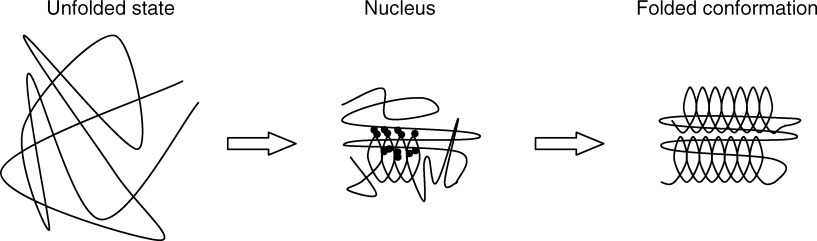
图4:nucleation-condensation model折叠机制示意图[9]
从实验现象推测折叠景观
在21世纪来临的前夕,David Baker课题组持续地通过实验手段研究小蛋白的折叠机理。除contact order的概念以外,他们还进行了一些额外的考察,这些观察结论决定了David Baker后来的研究走向。首先,通过蛋白主链酰胺键的氢-氘交换(hydrogen-deuterium exchange)实验,David Baker发现前述蛋白L上截取的62AAs IgG结构域并不像完整蛋白一样具有持久稳定的结构,而是明显分立为两个群体,稳定折叠群体的疏水核心质子受到保护而不发生氢-氘交换,而不稳定群体的所有质子都可以被交换为氘核[10]。
其次,当时的一些实验和理论研究表明,折叠过渡态是天然状态的延展版本,大多数相互作用已经部分形成。然而David Baker对蛋白L IgG结合结构域和src SH3结构域折叠动力学的研究与这一观点相左。在IgG结合结构域中,螺旋形成残基E32/A35/T39的三重甘氨酸突变导致了明显的不稳定效应,然而动力学测定显示三重突变导致解折叠速率提高了56倍,而折叠速率仅下降了不到3倍,即稳定性的损失几乎完全归因于解折叠速率的提高。这表明在折叠的限速步骤中,螺旋结构尚没有完全形成[11]。在src SH3结构域中,保守氨基酸Glu 30、Ser 47和Thr 50所形成的non-local氢键网络对折叠速率非常重要,据此推断折叠过渡态下远程氢键网络已经形成,而多肽链的其余部分在很大程度上是非结构化的[12]。
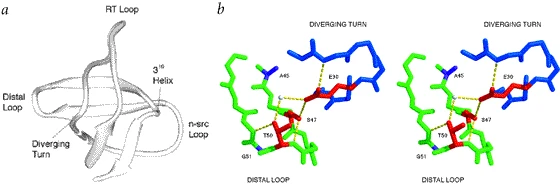
图5:通过广泛的突变研究关键残基对src SH3结构域折叠过程的重要性[12]
根据当时课题组内外的研究结果,David Baker推测蛋白质的折叠图景是多肽链的每个片段(segment)都采样了一系列满足其局部氨基酸序列的局部构象,而非局部相互作用可以优先稳定这些原本是瞬态的局部结构的近天然排列,从而使得这些片段同时采样到正确的结构和取向,并推动疏水氨基酸埋藏在核心中时,折叠过程随之发生。
Rosetta的诞生:将折叠机理知识运用于结构预测
1994年,CASP(Critical Assessment of Protein Structure Prediction)比赛首次举办,旨在通过社区竞赛推动蛋白质结构预测领域的发展。自David Baker了解到CASP后,他一直对结构预测问题非常感兴趣。在研究折叠动力学的过程中,他们还进行了一些统计上的研究,例如发现某些局部序列在跨蛋白质家族外被重复观察到[13],且局部序列与局部结构之间存在明显的映射关系[14]。
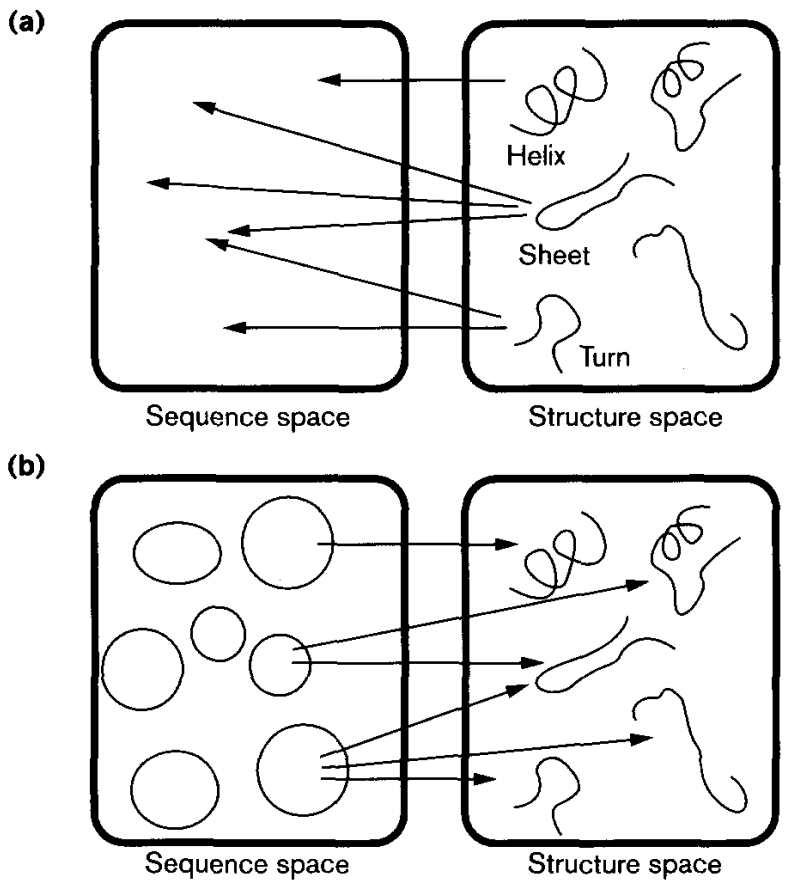
图6:序列-结构关系的两种研究模式[15]
基于这些铺垫,David Baker开始进军结构预测领域,初步的假设即是“短序列片段的结构应该类似于已知结构数据库中最相关序列所采用的局部结构”。为了在计算上易于处理,最早的研究将每个侧链都简化为一个球体,即质心(centroid)模型。考虑到9-mer残基片段具有最高的局部序列-结构相关性[15],对于待折叠序列的每个9-mer残基片段,使用近邻序列的已知结构来构建主链扭转角,随后通过模拟退火(Simulated Annealing)以不断优化结构打分,打分函数的推导详见引文[16]。David Baker团队发现,上述方法能够快速有效地将小蛋白折叠为具有疏水核心的紧凑三维结构,这种将片段近似、简化侧链模型和蒙特卡洛模拟退火组合而成的结构预测方法最早被David Baker的研究生Kim Simons称为“Rosetta”。

图7:使用Rosetta进行从头结构预测[16]
基于对若干小蛋白计算机折叠景观的分析,David Baker团队发现Rosetta的不同折叠轨迹最终将得到不同的构象,而天然结构通常接近最大的聚类簇[17]。这一现象表明天然结构并非一个孤立的能量最低点,而是坐落在一个宽阔、由大量结构相似的低能构象组成的盆地中。这一发现带来了新的预测范式,即相比于依赖远非完美的能量函数,不如通过结构聚类来识别最“拥挤”的低能量区域,聚类中心可能是更接近天然模型的结构。此外,这一结果还表明蛋白质可能已经进化出这种具有宽阔能量盆地的天然结构,以保证折叠过程的高效性和对氨基酸序列变化的稳健性。
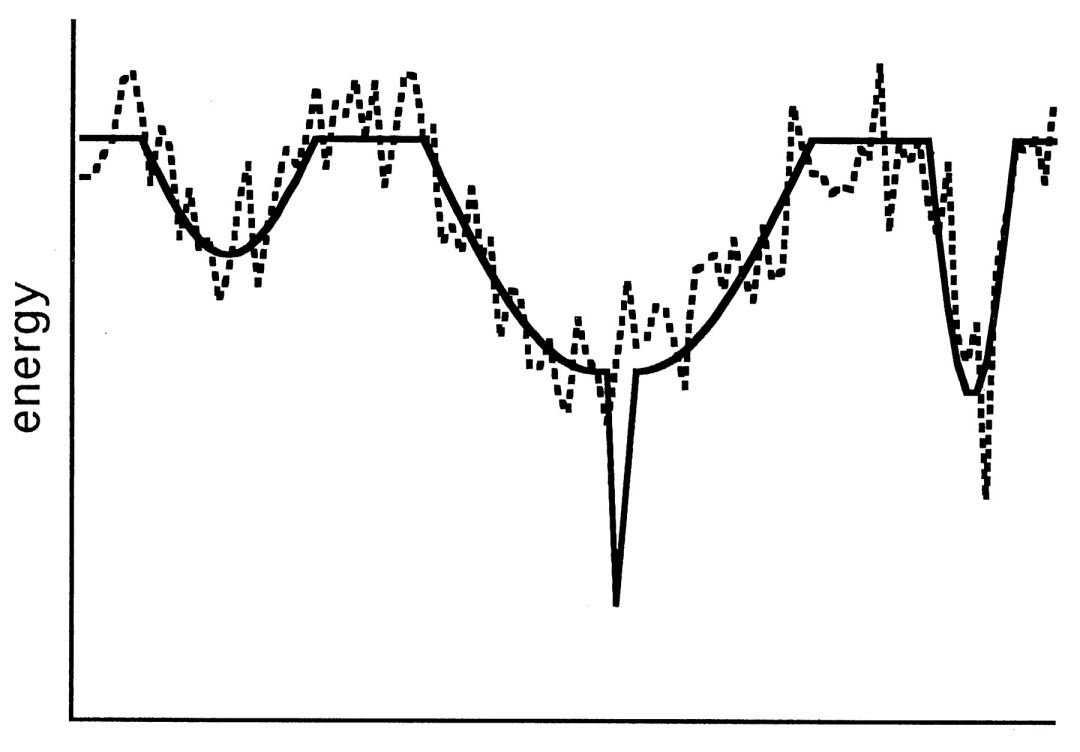
图8:蛋白质可能进化出了漏斗样能量景观[17]
为了更准确地建模并区分不同的极小值,Rosetta随后扩展了所有侧链原子,以同时最小化侧链和主链自由度的能量。值得注意的是,质心模型不仅仅是一种计算上的捷径,同时也是一种抽象,有效地模糊化了能量景观上的微小细节和崎岖之处,使得基于蒙特卡洛的退火方法能够高效地找到正确折叠所处的大致区域。
当使用改进的全原子模型进行评估时,David Baker团队发现天然结构在能量上几乎总是低于Rosetta预测的替代拓扑结构。然而,这种能量降低仅发生在天然结构2-3 Å RMSD的范围内。为了进行更全面的构象搜索,David Baker团队当时的计算资源是远远不够的;因此,他们创建了名为Rosetta@home的分布式计算项目(https://boinc.bakerlab.org/),旨在利用公众志愿者的闲置计算资源来执行Rosetta预测。Rosetta@home随后为David Baker实验室的许多研究做出了极为宝贵的贡献。

图9:使用Rosetta@home帮助解决生物学问题
Rosetta在单体蛋白质的结构预测中取得发展之后,David Baker实验室将类似的思想运用于蛋白质-蛋白质对接[18]、膜蛋白结构预测[19]、对称寡聚体组装[20]和RNA折叠[21]中。在所有这些案例中,David Baker团队发现与单体可溶蛋白质的情况相似,与Rosetta产生的结构相比,天然结构在能量上明显更优。倘若我们的采样范围足够接近天然结构,那么准确的结构预测是可能的。这种普遍现象实际上是生物大分子自由能景观的一个基本特征,例如对多肽链而言,其能够达到的非天然状态的构象数目极大,折叠时必然产生巨大的熵成本。为了弥补熵的损失,天然状态必须在能量上比非天然状态低得多。
或许更为读者熟知的是,尽管有了Rosetta@home,蛋白质结构预测的成功率依然不高、进展较为缓慢,直到AlphaFold2时代才取得令人瞩目的成就。然而,David Baker团队发现倘若能够结合稀疏的实验数据来指导构象搜索时,Rosetta结构预测方法突然变得很有效。这是因为在搜索庞大的构象空间时,即便是非常稀疏的信息量也将大幅加快定位最小值的位置。利用NMR数据的CS-Rosetta[22]和利用XRD数据的MR-Rosetta[23]在很长一段时间内作为结构解析的常规方法。
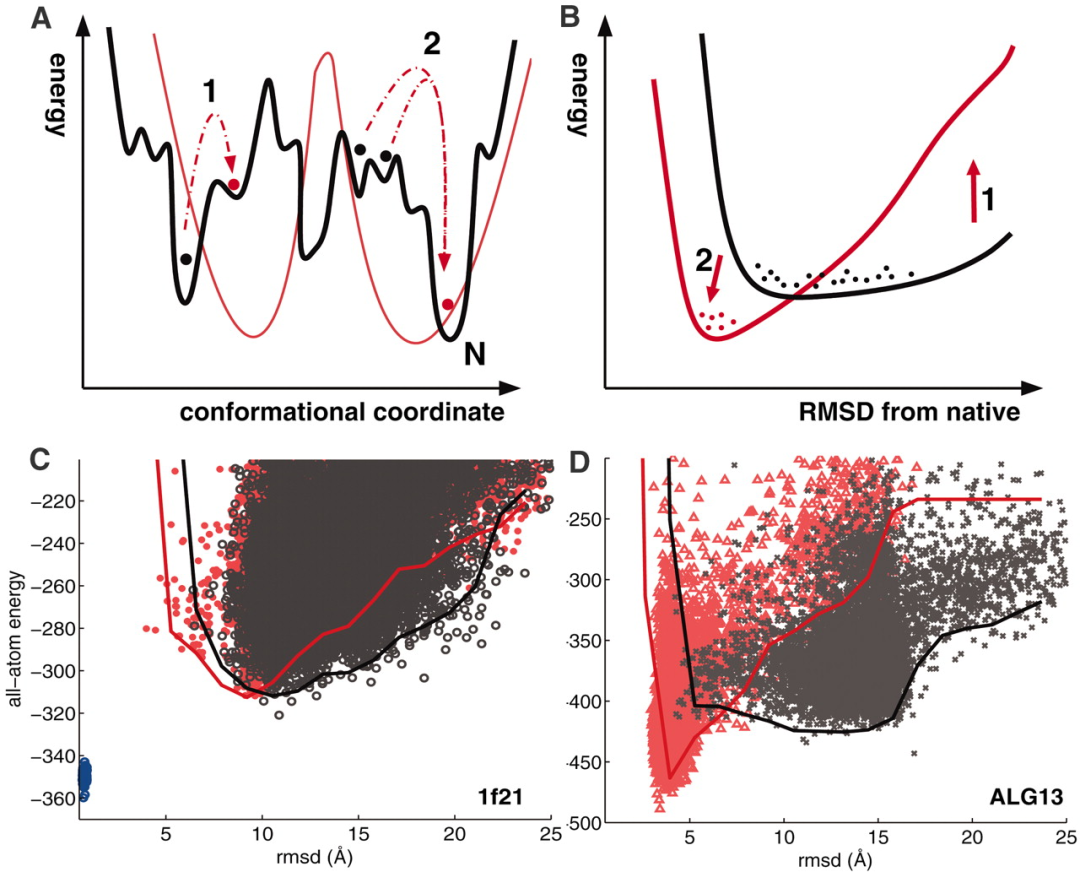
图10:稀疏实验数据显著改善Rosetta结构预测[22]
理解-超越:从预测结构到设计结构
上文提到,为了更准确地预测精细结构,全原子模型被引入到Rosetta中。David Baker敏锐地意识到,可以利用Rosetta解决一类全新的问题:如果Rosetta可以搜索给定序列的最低能量结构,那它应该也可以用来搜索给定结构的最低能量序列,这就是蛋白质设计问题。课题组的Brian Kuhlman开发了有效的设计方法,通过旋转异构体的替换结合蒙特卡洛搜索来优化序列,使用Rosetta预测优化序列的骨架构象,并在序列设计和骨架优化之间反复循环。利用这种策略,他们设计了球状蛋白Top7,它具有PDB数据库中完全不存在的全新α/β混合拓扑结构,且设计模型与解析的晶体结构之间仅存在1.17Å主链原子RMSD[24]。这项突破性的研究有效验证了当时所使用能量函数的准确性,且表明设计全新的蛋白质不仅在物理上是可能的,而且可以具有非凡的稳定性。
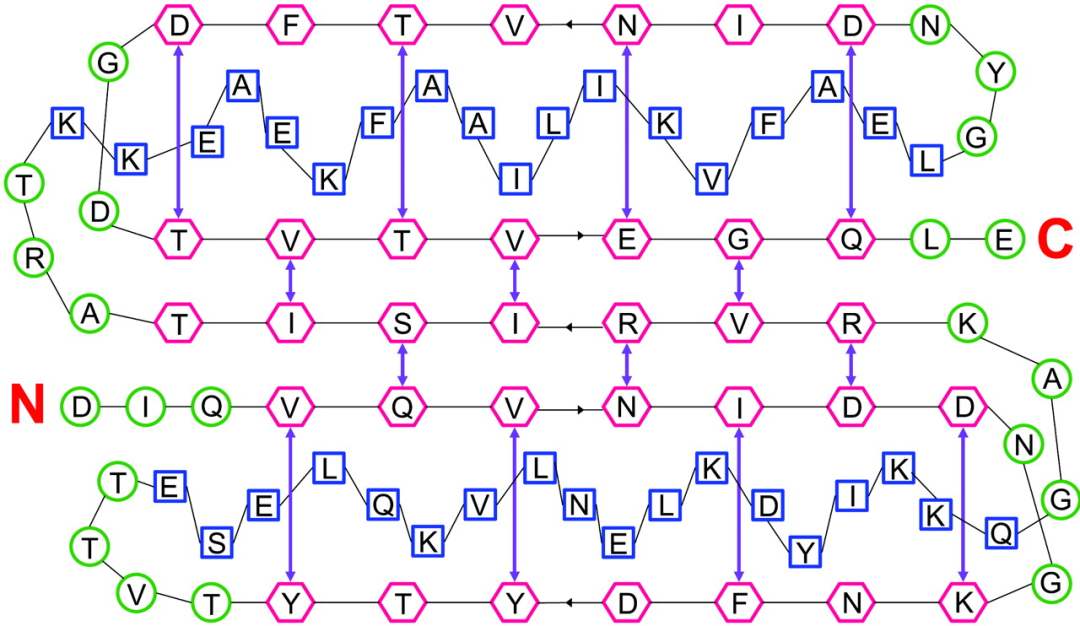
图11:从头设计具有全新拓扑结构的球状蛋白Top7[24]
与结构预测类似,当可以设计单体蛋白质后,David Baker团队迅速将这种方法应用于设计可以相互作用的蛋白质正交对和结构域-结构域界面等[25]。
蛋白质最神奇的功能之一就是高效催化化学反应,相比于高稳定性球状蛋白,设计具有功能活性的酶极具挑战性。David Baker团队考虑可以首先设计一个理想的活性位点,即包含催化残基的理论酶(theozyme)过渡态,然后通过高效的几何哈希算法来尝试设计纳入这些活性位点的蛋白质。通过这种被称为Rosetta Match的方法,实现了催化Kemp消除酶和逆醛缩酶的从头设计[26-27]。尽管设计得到的酶活性远低于天然存在的酶,但可以通过定向进化得到显著改善,被认为是酶设计领域的里程碑工作。
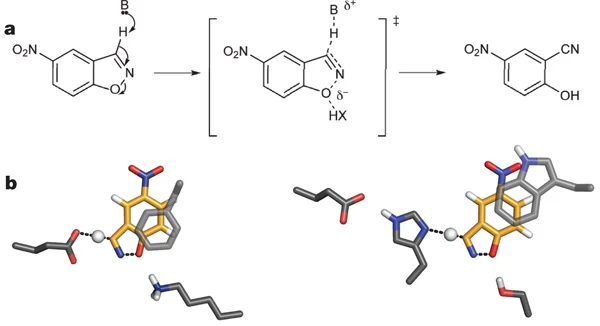
图12:通过嫁接理论酶过渡态从头设计酶[26]
蛋白质设计一个极具吸引力的应用领域是创造用于治疗和诊断的新型结合蛋白,针对这一问题,David Baker团队也开发了通用的蛋白设计方法:首先将单个游离氨基酸残基对接至目标蛋白表面,以识别有利的相互作用模式;随后,搜索蛋白质支架结构库,找到那些能够支持热点残基且与靶标蛋白具有良好形状互补性的支架,将这些支架对接至靶标蛋白,并通过RosettaDesign重新设计支架表面的其它残基。利用这种计算策略并结合亲和力成熟等实验手段,David Baker团队成功针对多种靶标蛋白(如流感病毒血凝素)设计得到了极高亲和力的结合蛋白[28],这些小蛋白有作为诊断和治疗试剂应用的潜力。

图13:通过对接鉴定热点残基结合高形状互补度支架从头设计结合蛋白[28]
有趣的是,Rosetta@home通过贡献闲置算力帮助David Baker团队完成了无数的预测和设计任务,该程序在运行时会显示屏幕保护程序,展现正在进行的计算过程。然而,有几名志愿者表示他们在观看屏保后认为Rosetta在某些情况下效率低下,应该有更好的方法指导蛋白质折叠。为了实现这一目标,David Baker团队与华盛顿大学计算机科学系合作,开发了名为FoldIt的在线多人游戏(https://fold.it/),它包装了Rosetta的优化算法和能量函数,并提供了一个交互式游戏界面,让玩家自行调用各种方法寻找能量更优的结构,以帮助解决蛋白质结构预测和设计问题。FoldIt玩家协助解决了逆转录病毒蛋白酶的结构、开发了寻找低能蛋白质构象的新算法等等。FoldIt至今仍在积极更新并为解决生命科学问题做出独特贡献。
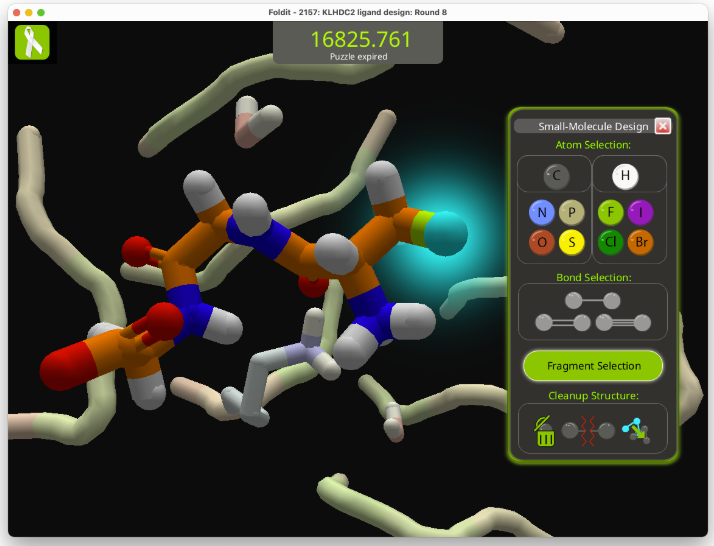
图14:使用FoldIt帮助设计小分子药物
结语
David Baker教授被媒体誉为“上帝之手”,即便是在AI时代来临之前,其团队在蛋白质结构预测和设计领域的成果也远远超过这篇短文所能涵盖的范围。而在AI技术为生物学带来惊天变革之后,David Baker教授仍在不断推陈出新,领导着整个领域继续蓬勃发展。本文着重回顾了David Baker教授的早期研究脉络,我们不难看出,他的科研之路是一条逻辑严密、环环相扣又层层递进的探索之旅。从对经典热力学假说的审慎质疑出发,通过精巧的实验和计算分析一步步揭示了“动力学可及性”在蛋白质折叠中的关键作用。也正是基于对折叠机理、能量景观等概念的深刻理解,Rosetta才能应运而生,而后又被反向应用于蛋白质设计,开启了理性设计蛋白质的宏伟纪元。
David Baker教授的早期研究历程完美地诠释了“天才”科学家是如何通过持续的思想更新和迭代来一步步揭开分子世界神秘面纱下的壮丽图景,从“理解规则”、“应用规则”到“扩展规则”的完整历程。
本文作者:WFZ
参考文献
[1] Anfinsen, C. B. Principles that Govern the Folding of Protein Chains. Science181, 223–230 (1973).
[2] Levinthal, C. Are there pathways for protein folding? J. Chim. Phys.65, 44–45 (1968).
[3] Baker, D. & Agard, D. A. Kinetics versus Thermodynamics in Protein Folding. Biochemistry33, 7505–7509 (1994).
[4] Baker, D., Sohl, J. L. & Agard, D. A. A protein-folding reaction under kinetic control. Nature356, 263–265 (1992).
[5] Gu, H. et al. A phage display system for studying the sequence determinants of protein folding. Protein Science4, 1108–1117 (1995).
[6] Riddle, D. S. et al. Functional rapidly folding proteins from simplified amino acid sequences. Nat Struct Mol Biol4, 805–809 (1997).
[7] Kim, D. E., Gu, H. & Baker, D. The sequences of small proteins are not extensively optimized for rapid folding by natural selection. Proceedings of the National Academy of Sciences95, 4982–4986 (1998).
[8] Kevin, W. P. et al. Contact order, transition state placement and the refolding rates of single domain proteins. Journal of Molecular Biology277, 985–994 (1998).
[9] Nölting, B. & Agard, D. A. How general is the nucleation–condensation mechanism? Proteins: Structure, Function, and Bioinformatics73, 754–764 (2008).
[10] Yi, Q. & Baker, D. Direct evidence for a two-state protein unfolding transition from hydrogen-deuterium exchange, mass spectrometry, and NMR. Protein Science5, 1060–1066 (1996).
[11] Kim, D. E. et al. The single helix in protein L is largely disrupted at the rate-limiting step in folding. Journal of Molecular Biology284, 807–815 (1998).
[12] Grantcharova, V. P., Riddle, D. S., Santiago, J. V. & Baker, D. Important role of hydrogen bonds in the structurally polarized transition state for folding of the src SH3 domain. Nat Struct Mol Biol5, 714–720 (1998).
[13] Han, K. F. & Baker, D. Recurring Local Sequence Motifs in Proteins. Journal of Molecular Biology251, 176–187 (1995).
[14] Han, K. F. & Baker, D. Global properties of the mapping between local amino acid sequence and local structure in proteins. Proceedings of the National Academy of Sciences93, 5814–5818 (1996).
[15] Bystroff. C. et al. Local sequence-structure correlations in proteins. Current Opinion in Biotechnology7, 417–421 (1996).
[16] Simons, K. T., Kooperberg, C., Huang, E. & Baker, D. Assembly of protein tertiary structures from fragments with similar local sequences using simulated annealing and bayesian scoring functions. Journal of Molecular Biology268, 209–225 (1997).
[17] D. Shortle, K.T. Simons, & D. Baker, Clustering of low-energy conformations near the native structures of small proteins, Proc. Natl. Acad. Sci. U.S.A. 95 (19) 11158-11162 (1998).
[18] Gray, J.J., Moughon, S., Wang, C., Schueler-Furman, O., Kuhlman, B., Rohl, C.A. and Baker, D. (2003) Protein–protein docking with simultaneous optimization of rigid-body displacement and side-chain conformations. J. Mol. Biol. 331, 281–299.
[19] Barth, P., Schonbrun, J. and Baker, D. (2007) Toward high-resolution prediction and design of transmembrane helical protein structures. Proc. Natl. Acad. Sci. U.S.A. 104, 15682–15687
[20] Andre, I., Bradley, P., Wang, C. and Baker, D. (2007) Prediction of the structure of symmetrical protein assemblies. Proc. Natl. Acad. Sci. U.S.A. 104, 17656–17661
[21] Das, R. and Baker, D. (2007) Automated de novo prediction of native-like RNA tertiary structures. Proc. Natl. Acad. Sci. U.S.A. 104, 14664–14669
[22] Raman, S., Lange, O.F., Rossi, P., Tyka, M., Wang, X., Aramini, J., Liu, G., Ramelot, T., Eletsky, A., Szyperski, T. et al. (2010) NMR structure determination for larger proteins using backbone-only data. Science 327, 1014–1018
[23] DiMaio, F., Terwilliger, T.C., Read, R.J., Wlodawer, A., Oberdorfer, G., Wagner, U., Valkov, E., Alon, A., Fas, D., Axelrod, H.L. et al. (2011) Improved molecular replacement by density- and energy-guided protein structure optimization. Nature 473, 540–543
[24] Kuhlman, B., Dantas, G., Ireton, G.C., Varani, G., Stoddard, B.L. and Baker, D. (2003) Design of a novel globular protein fold with atomic-level accuracy. Science 302, 1364–1368
[25] Kortemme, T., Joachimiak, L.A., Bullock, A.N., Schuler, A.D., Stoddard, B.L. and Baker, D. (2004) Computational redesign of protein–protein interaction specificity. Nat. Struct. Mol. Biol. 11, 371–379
[26] Rothlisberger, D., Khersonsky, O., Wollacott, A.M., Jiang, L., DeChancie, J., Betker, J., Gallaher, J.L., Althoff, E.A., Zanghellini, A., Dym, O. et al. (2008) Kemp elimination catalysts by computational enzyme design. Nature 453, 190–195
[27] Jiang, L., Althoff, E.A., Clemente, F.R., Doyle, L., R ¨othlisberger, D., Zanghellini, A., Gallaher, J.L., Betker, J.L., Tanaka, F., Barbas, 3rd, C.F. et al. (2008) De novo computational design of retro-aldol enzymes. Science 319, 1387–1391
[28] Fleishman, S.J., Whitehead, T.A., Ekiert, D.C., Dreyfus, C., Corn, J.E., Strauch, E.-M., Wilson, I.A. and Baker, D. (2011) Computational design of proteins targeting the conserved stem region of influenza hemagglutinin. Science 332, 816–821
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-15,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

