Nat. Methods | CytoTRACE 2:重塑单细胞发育潜能的精准重建新一代算法
Nat. Methods | CytoTRACE 2:重塑单细胞发育潜能的精准重建新一代算法

DrugOne
发布于 2025-11-17 20:50:37
发布于 2025-11-17 20:50:37
DRUGONE
单细胞 RNA 测序(scRNA-seq)为解析细胞发育轨迹和细胞命运决策提供了强大工具,但如何准确推断细胞发育潜能仍然是一项挑战。研究人员开发了 CytoTRACE 2 —— 一种基于机器学习的改进版发育潜能重建算法,它在原始 CytoTRACE 框架基础上大幅提升了精度、稳定性与跨平台泛化能力。
CytoTRACE 2 结合转录组复杂度(transcriptional diversity)与数据驱动特征选择,利用超过 2000 个高质量单细胞数据集进行训练与验证。与前一代算法相比,该模型在干细胞分化轨迹重建、发育状态排序及跨组织预测任务中均表现出显著优势,尤其在多物种与多测序技术背景下的泛化性更强。研究人员还引入 基因重要性评分(gene importance scoring) 模块,使模型能识别影响发育潜能的关键基因,从而在机制研究中具有解释性价值。

单细胞转录组技术的快速发展使研究人员能够从全基因组水平解析细胞的状态多样性及发育过程。然而,如何量化和重建细胞发育潜能仍是一项复杂任务。传统方法(如 Monocle、Slingshot 及原版 CytoTRACE)依赖伪时间推断和基因表达趋势拟合,虽然在部分体系中有效,但普遍受到噪声、批次效应和数据来源差异的影响。特别是原版 CytoTRACE 虽通过基因表达复杂度估计实现了零假设建模,但在低质量数据、稀疏转录组或跨物种应用时性能下降明显。
为突破这些限制,研究人员基于多层次数据统计特征与机器学习优化策略,提出了 CytoTRACE 2。新模型能够在保持无监督特性的同时自动学习不同特征对发育潜能的贡献权重,从而在多数据集环境下保持一致性与可解释性。
方法
CytoTRACE 2 的设计核心是引入特征学习和非线性加权机制,以提升发育潜能的预测精度。整体流程包括以下四个模块:
- 特征构建
从单细胞表达矩阵中提取细胞层面的表达复杂度指标(基因检测数量、表达熵、稀疏度等),并结合高变基因子集构成多维特征空间。
- 特征加权与建模
利用随机森林与神经网络结合的回归框架,自适应学习各特征与发育潜能之间的非线性关系,从而确定最具贡献的信号模式。
- 发育潜能得分计算
将学习到的权重映射至单细胞表达特征,生成平滑的潜能分布(Developmental Potential Score),该分布在细胞群体中形成由干性到分化的连续梯度。
- 基因重要性识别
通过特征贡献分析(SHAP)评估单个基因对潜能变化的影响,并生成可解释的基因调控图谱。
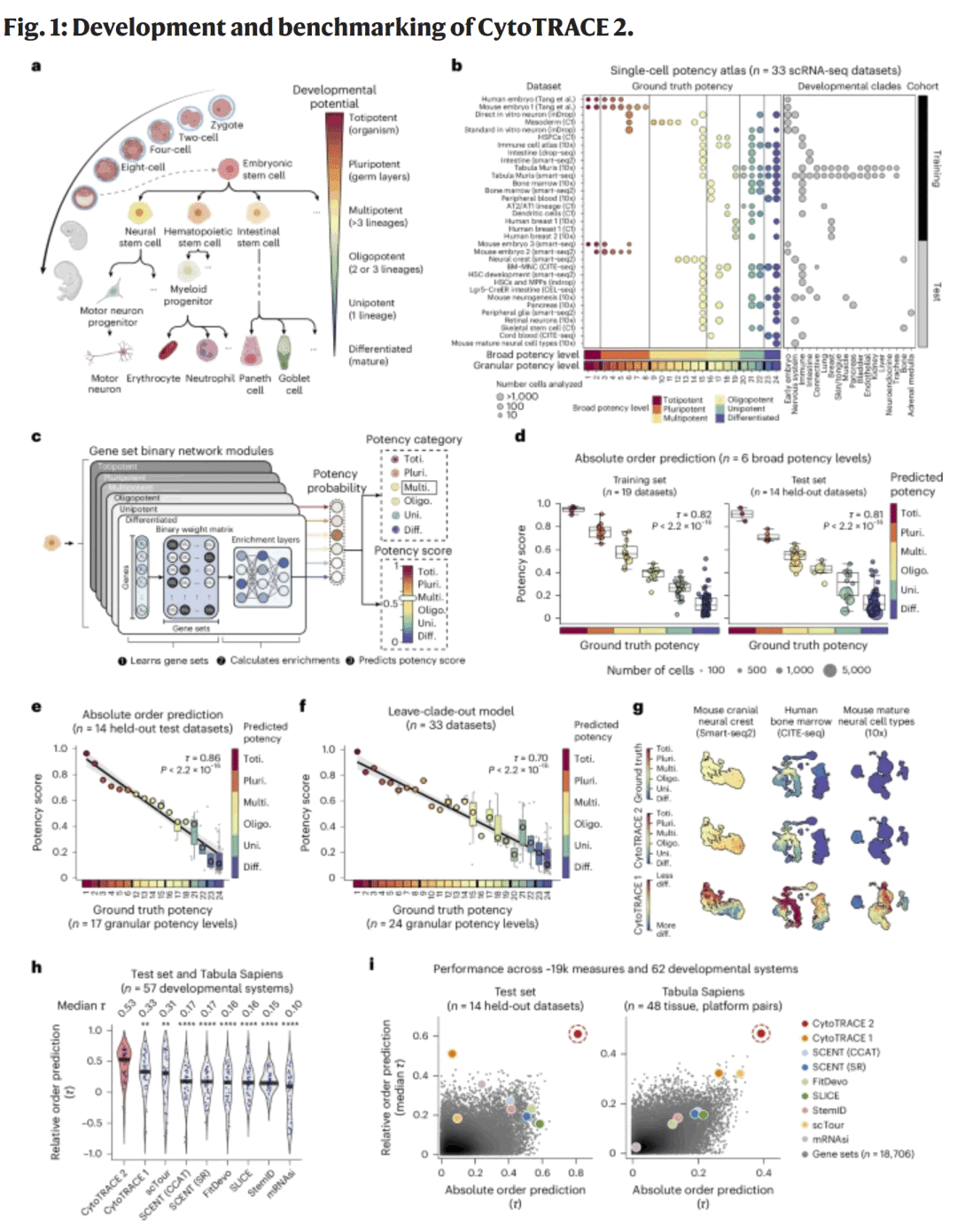
结果
研究人员在多个单细胞数据集中评估了 CytoTRACE 2 的性能,包括人类胚胎发育、小鼠造血系统和神经干细胞谱系。结果显示,CytoTRACE 2 相较原版 CytoTRACE 显著提升发育潜能预测的相关性与分辨率。
在跨平台分析中(如 10× Genomics、Smart-seq2、Microwell-seq),CytoTRACE 2 保持一致的排序结果,表明该算法对技术噪声具有强鲁棒性。
在模拟数据与真实生物系统中,模型能准确识别干细胞群体,并在连续的分化路径上呈现符合生物逻辑的潜能递减趋势。
此外,CytoTRACE 2 自动识别出的高贡献基因与实验验证的发育调控因子高度重叠,例如在人类造血系统中,模型优先选出了 GATA2、RUNX1、SPI1 等关键转录因子;在神经发育数据中,识别到 SOX2、PAX6、NEUROD1 等调控因子。
这些结果证明,CytoTRACE 2 不仅能够从噪声较高的单细胞数据中提取稳定的发育信号,还能提供可解释的分子机制视角。
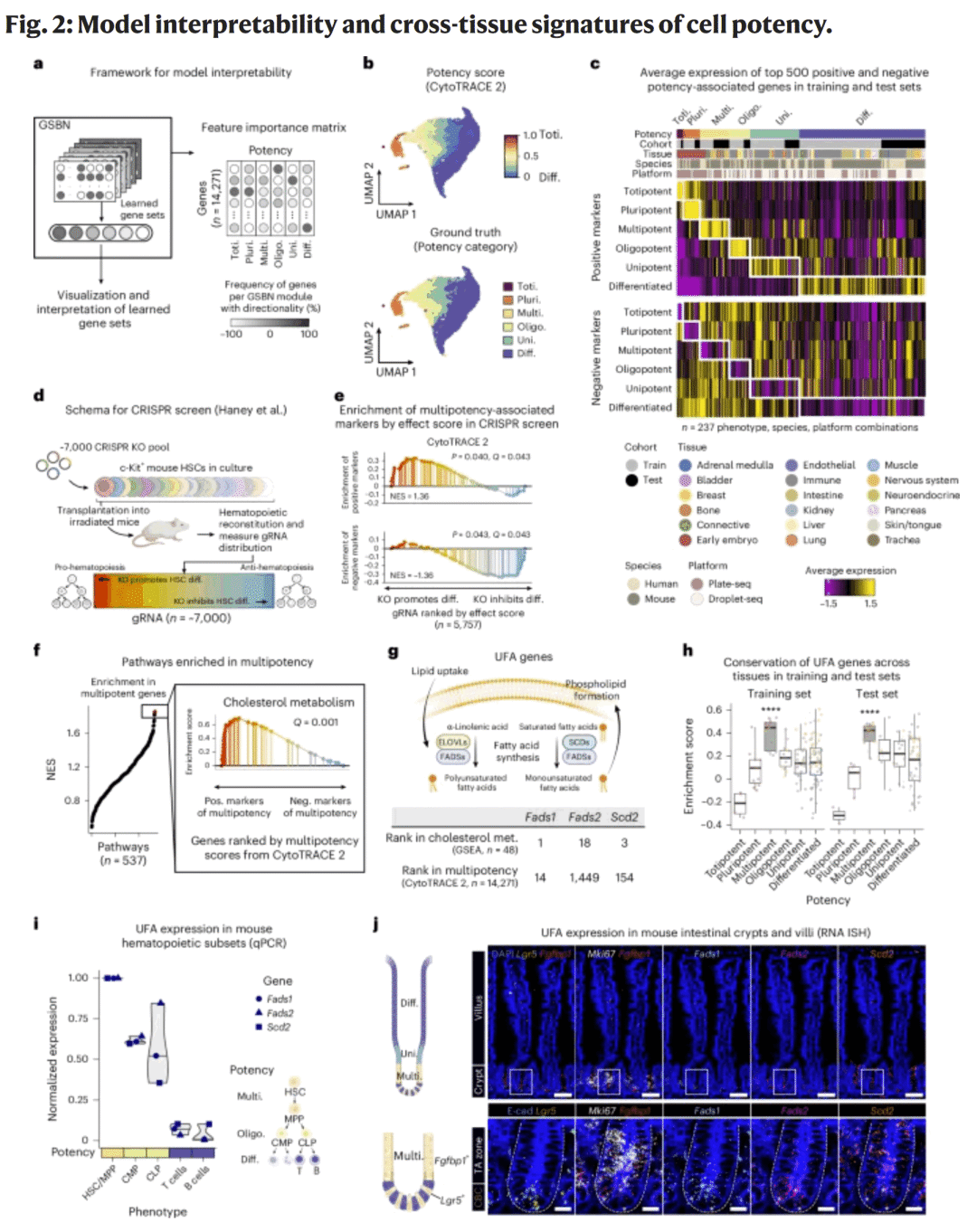
讨论
CytoTRACE 2 的提出标志着从统计型伪时间推断向学习型潜能建模的过渡。该算法通过整合转录复杂度、特征加权和非线性学习,在无监督条件下实现了对细胞发育潜能的稳定重建。
其主要优势体现在以下方面:
- 跨数据集泛化性 —— 模型在不同平台与物种间表现一致,无需重新训练;
- 生物学可解释性 —— 能识别对发育潜能具有显著影响的基因并揭示调控机制;
- 鲁棒性提升 —— 对稀疏、批次效应或低测序深度数据表现稳定;
- 通用性 —— 可扩展至癌症重编程、干细胞分化及组织再生等多类研究场景。
研究人员指出,未来工作将结合空间转录组与单细胞多组学数据,以进一步提升发育潜能建模的分辨率与多维解析能力。
整理 | DrugOne团队
参考资料
Kang, M., Gulati, G.S., Brown, E.L. et al. Improved reconstruction of single-cell developmental potential with CytoTRACE 2. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02857-2
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-28,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

