Nat. Mach. Intell. | 用神经微分方程学习细胞动力学
Nat. Mach. Intell. | 用神经微分方程学习细胞动力学

DrugOne
发布于 2025-12-25 15:03:31
发布于 2025-12-25 15:03:31
DRUGONE
单细胞测序为重建生物系统的动态过程提供了关键线索,但细胞命运决定同时受到确定性调控与随机波动的共同影响。现有方法多聚焦于确定性动力学,对随机性建模不足。研究人员提出 scDiffEq,一种基于神经随机微分方程(neural SDE)的生成式框架,用于同时学习细胞状态演化中的漂移(确定性)与扩散(随机性)。在带有谱系追踪的造血单细胞数据上,scDiffEq 在细胞命运预测、时间插值及体内外扰动模拟方面均优于现有方法,并可推广至仅含单时间点的单细胞数据,为解析发育与疾病中的细胞动力学提供了统一工具。

细胞分化、发育及疾病进程本质上是动态系统问题。单细胞 RNA 测序通过捕获大量细胞的瞬时分子状态,为推断细胞过去与未来状态提供了可能。传统轨迹推断和 RNA velocity 方法在一定程度上刻画了细胞转变趋势,但往往假设随机噪声在不同细胞状态间一致,难以反映真实的状态依赖性随机性。要深入理解细胞命运决策的调控逻辑,亟需同时刻画确定性调控与状态依赖随机性的动力学模型。
方法
scDiffEq 在低维嵌入空间中,用神经网络参数化漂移项与扩散项,构建神经随机微分方程以描述细胞状态随潜在时间的演化。模型通过最小化模拟细胞群体与真实观测群体在不同时间点上的分布差异进行训练,从而学习细胞特异性的动力学规律。该框架无需显式时间序列,可应用于多时间点、少时间点甚至单时间点的单细胞数据。
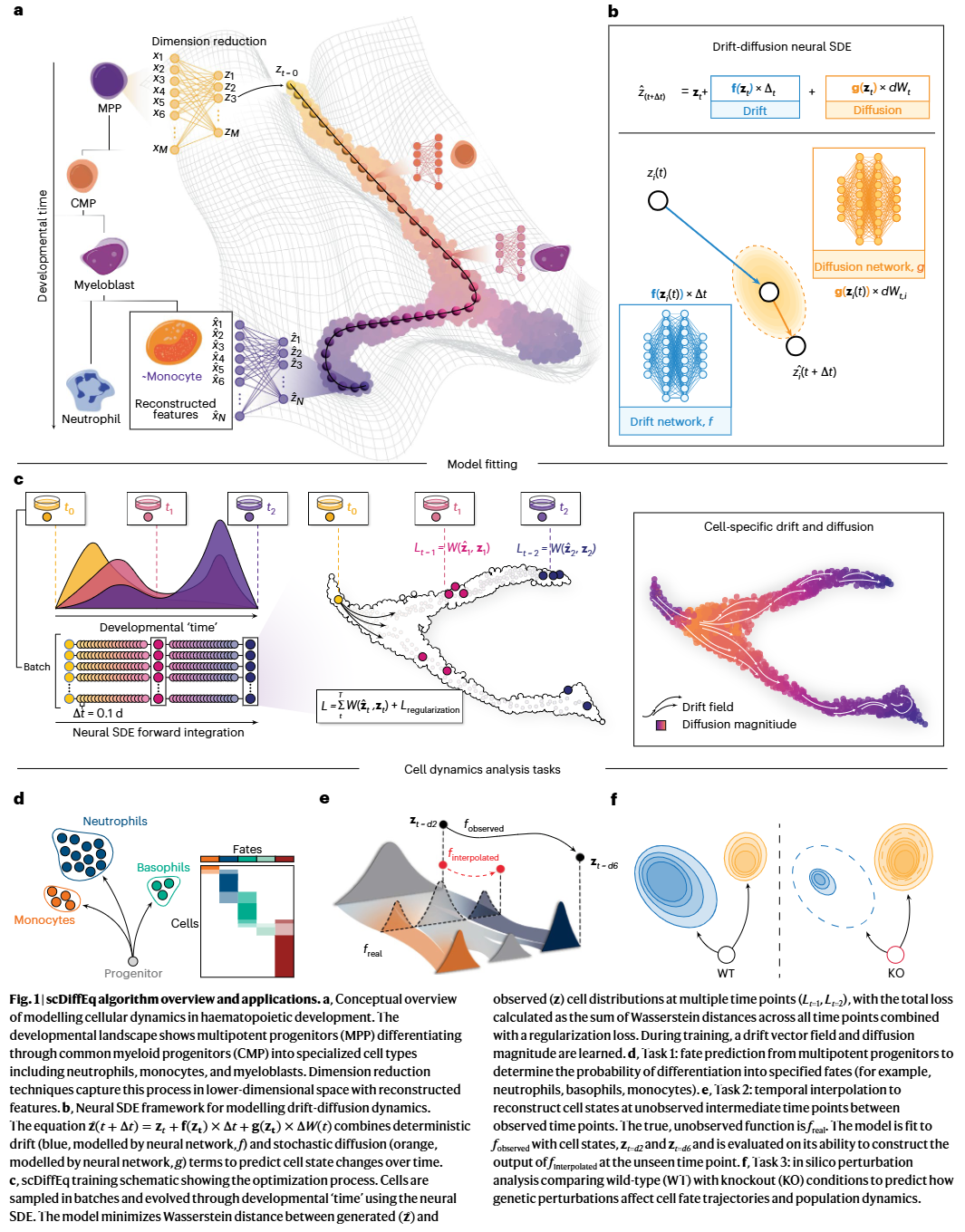
图1| scDiffEq 框架与应用。
结果
细胞命运预测性能提升
在带谱系追踪的造血数据集中,scDiffEq 在细胞命运预测任务中显著优于现有单细胞动力学方法,尤其在稀有命运与多命运分支的预测上表现更稳健。
未观测时间点的动力学重建
scDiffEq 能准确插值未观测的中间时间点细胞分布,重建连续发育轨迹,表明其学习到的动力学不仅是静态拟合,而是真实反映了时间演化过程。
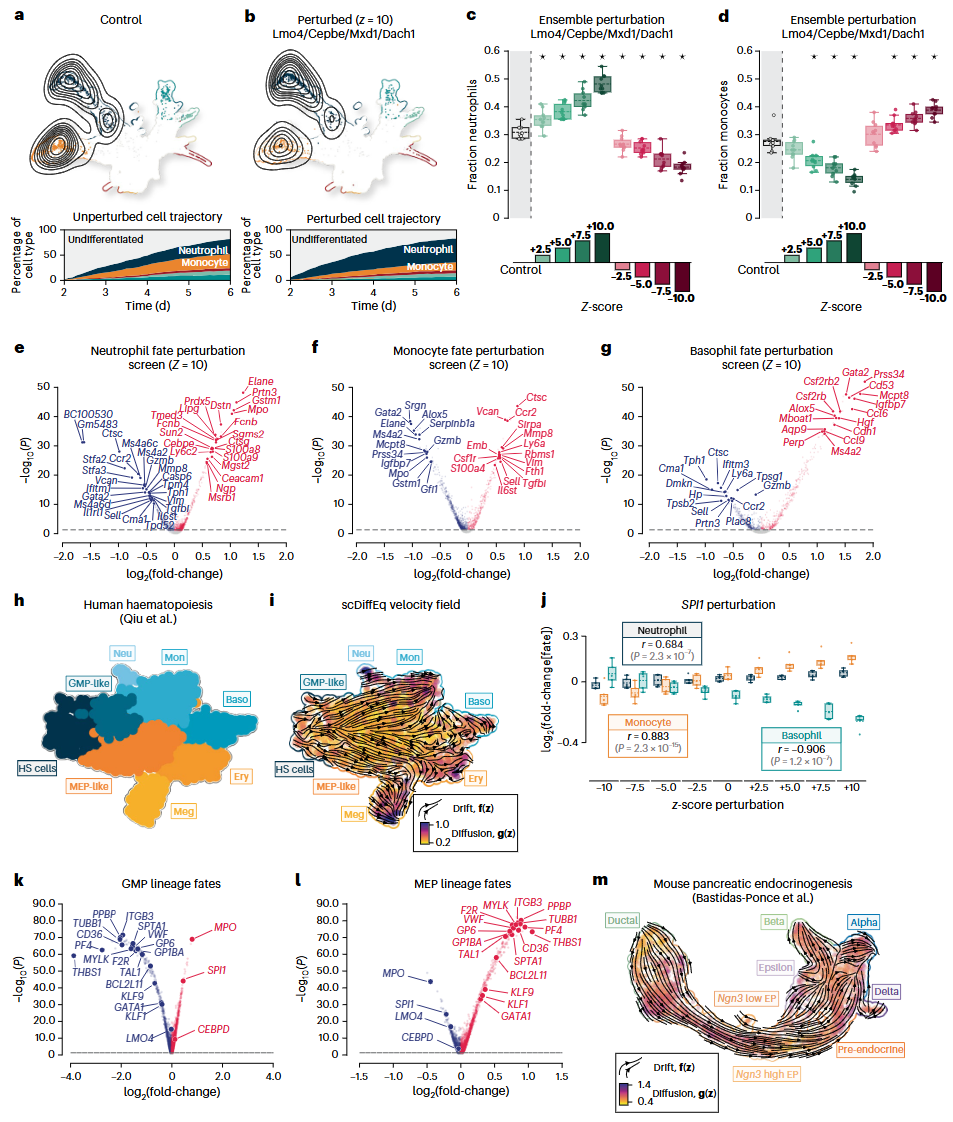
图 2|基于谱系追踪造血发育数据的 scDiffEq 性能评估。
体内外扰动的动力学模拟
通过对关键转录因子进行in silico 扰动,scDiffEq 能预测剂量依赖的命运变化趋势,成功重现已知的造血调控规律,显示其在功能基因筛选与机制假设生成中的潜力。

图3| 基因扰动下的命运变化模拟。
漂移与扩散的生物学解释
分析显示,细胞可塑性越高,其漂移与扩散幅度越大。scDiffEq 揭示了不同发育分支中确定性与随机性成分的动态变化,并能关联到关键调控因子的时序表达模式。
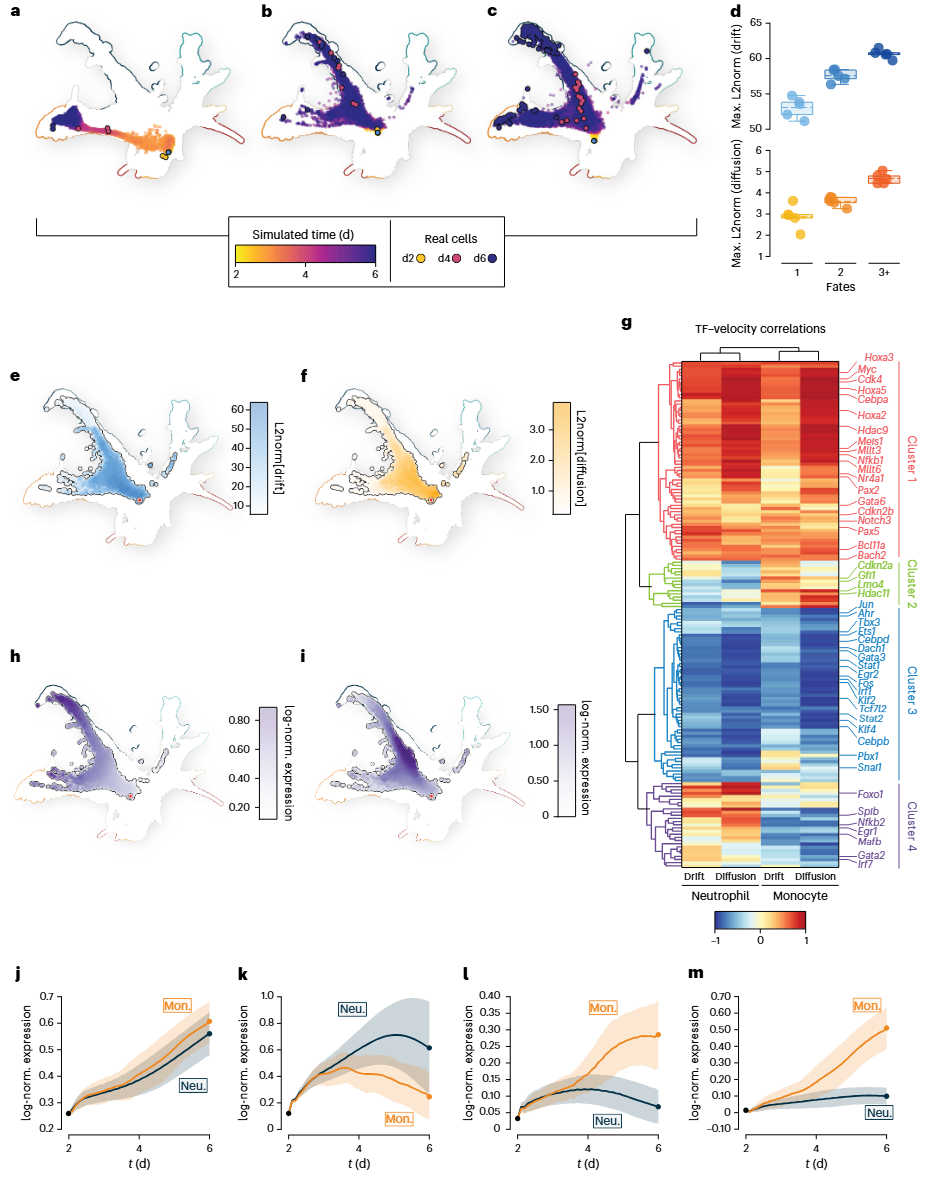
图4| 漂移–扩散分解与调控因子动态。
讨论
scDiffEq 提供了一种统一的生成式框架,用于从单细胞数据中学习细胞动力学的确定性与随机性成分。通过显式建模状态依赖的扩散过程,该方法能更真实地刻画多命运决策与稀有分支。尽管当前依赖低维嵌入表示,可能弱化部分基因层级相互作用,但其良好的可扩展性与跨数据集泛化能力,使其成为研究发育、生物稳态与疾病动力学的重要工具。未来结合多模态数据与更精细的表示学习,有望进一步提升其生物解释力。
整理 | DrugOne团队
参考资料
Vinyard, M.E., Rasmussen, A.W., Li, R. et al. Learning cell dynamics with neural differential equations. Nat Mach Intell (2025).
https://doi.org/10.1038/s42256-025-01150-3
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-19,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

