Cell | 微软推出GigaTIME: 多模态 AI 生成虚拟群体赋能肿瘤微环境建模
Cell | 微软推出GigaTIME: 多模态 AI 生成虚拟群体赋能肿瘤微环境建模

DrugAI
发布于 2026-01-06 14:07:04
发布于 2026-01-06 14:07:04
DRUGONE
肿瘤免疫微环境(TIME)在肿瘤进展和免疫治疗响应中起关键作用。多重免疫荧光(mIF)能够在单一组织切片上同时解析多种蛋白的空间分布,但其高成本和低通量限制了大规模应用。研究人员提出 GigaTIME,一种多模态人工智能框架,通过学习细胞形态与功能状态之间的跨模态映射,将常规 H&E 病理切片转换为虚拟 mIF 图像。该方法基于约 4,000 万个成对 H&E–mIF 细胞数据训练,并应用于超过 14,000 名患者,构建了大规模、多癌种的虚拟肿瘤微环境群体。该虚拟群体揭示了大量此前难以发现的蛋白–生物标志物关联,并在独立队列中得到验证,为群体尺度的肿瘤免疫研究提供了新路径。

肿瘤免疫微环境由肿瘤细胞、免疫细胞、成纤维细胞和血管相关细胞等构成,其空间组织方式深刻影响免疫逃逸和治疗反应。传统免疫组化一次只能检测单一蛋白,难以刻画复杂的细胞互作网络。mIF 技术虽然能够同时检测多种蛋白,但在真实临床环境中难以大规模部署。
相比之下,H&E 切片在临床中广泛、低成本获取。尽管 H&E 不直接反映蛋白活性,但其中隐含的细胞形态与空间结构可能蕴含免疫状态信息。随着多模态与基础模型的发展,研究人员开始探索利用人工智能从 H&E 中推断空间蛋白表达,从而突破 mIF 数据稀缺的限制。
方法
GigaTIME 是一个跨模态生成框架,核心任务是从 H&E 图像生成虚拟 mIF。研究人员基于成对的 H&E 与 mIF 切片数据,构建了以编码器–解码器为核心的深度学习模型,对 21 个肿瘤免疫相关蛋白通道进行像素级预测。模型首先在约 4,000 万个细胞级样本上进行训练,随后应用于大规模真实世界 H&E 队列,生成完整的虚拟 mIF 全切片图像,并进一步用于空间统计与群体层面的临床分析。
结果
虚拟 mIF 群体的构建
GigaTIME 被应用于来自多家医院的大规模 H&E 数据,生成数十万张虚拟 mIF 切片,覆盖多种癌种和亚型。由此形成的虚拟群体同时包含空间蛋白激活信息和临床变量,为群体级分析提供基础。
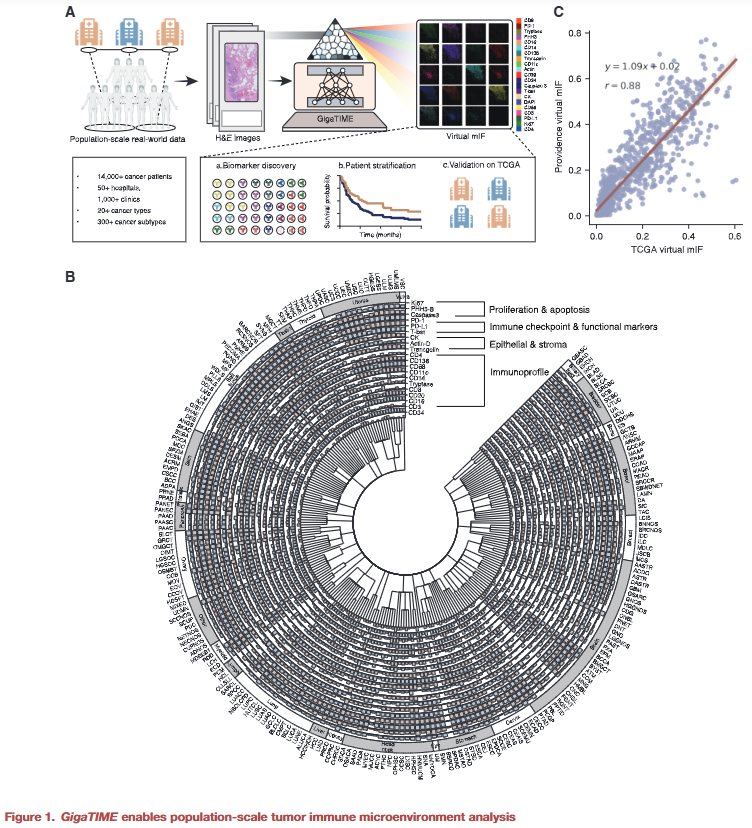
图1 |GigaTIME 框架与虚拟群体构建
H&E → mIF 跨模态翻译性能
在像素级、细胞级和切片级评估中,GigaTIME 在大多数蛋白通道上显著优于传统图像翻译模型,能够准确复现核标记、免疫检查点蛋白及免疫细胞相关信号,显示其确实捕捉到了形态与功能之间的非平凡关系。
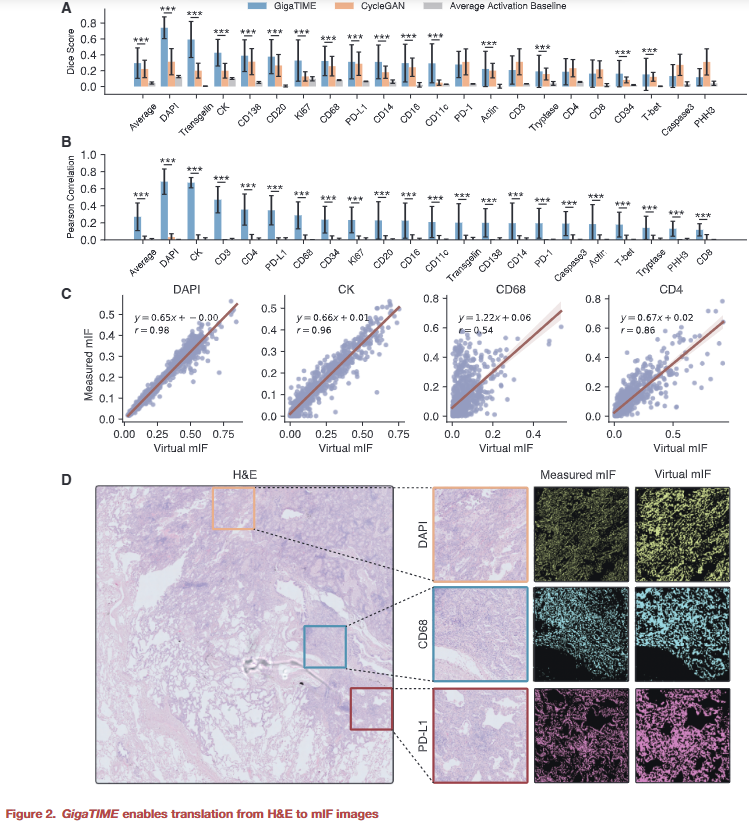
图2|虚拟 mIF 与真实 mIF 的定量与定性对比
蛋白–生物标志物关联的群体发现
基于虚拟群体,研究人员系统分析了蛋白激活与基因突变、肿瘤突变负荷、免疫检查点状态等临床指标之间的关系,识别出上千个显著关联,涵盖泛癌种、特定癌种及亚型层面。
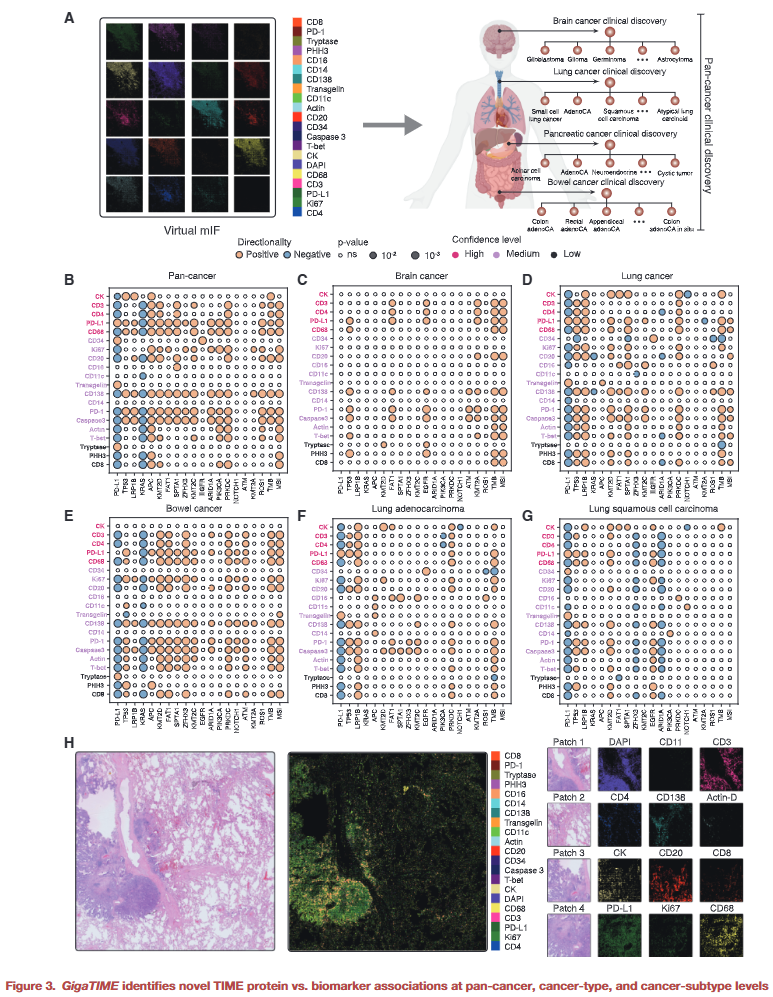
图3|蛋白–生物标志物关联全景图
分期与生存预测中的患者分层
虚拟 mIF 蛋白激活模式可用于患者分层,区分不同病理分期及生存结局。将多个蛋白通道整合为综合特征后,其预测能力明显优于单一蛋白,凸显多重信号协同的重要性。
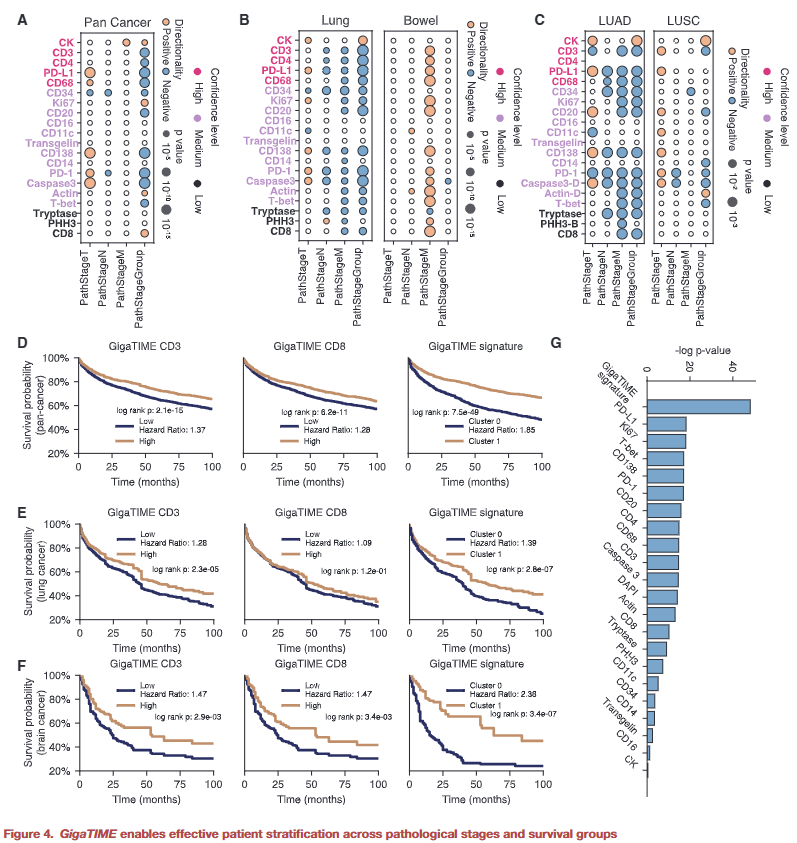
图4 |基于虚拟 mIF 的分期与生存分析
独立队列的泛化验证
研究人员将 GigaTIME 应用于独立癌症队列,发现不同数据来源中虚拟蛋白激活模式高度一致,且关键蛋白–生物标志物关联具有显著重叠,验证了模型的稳健性与可推广性。
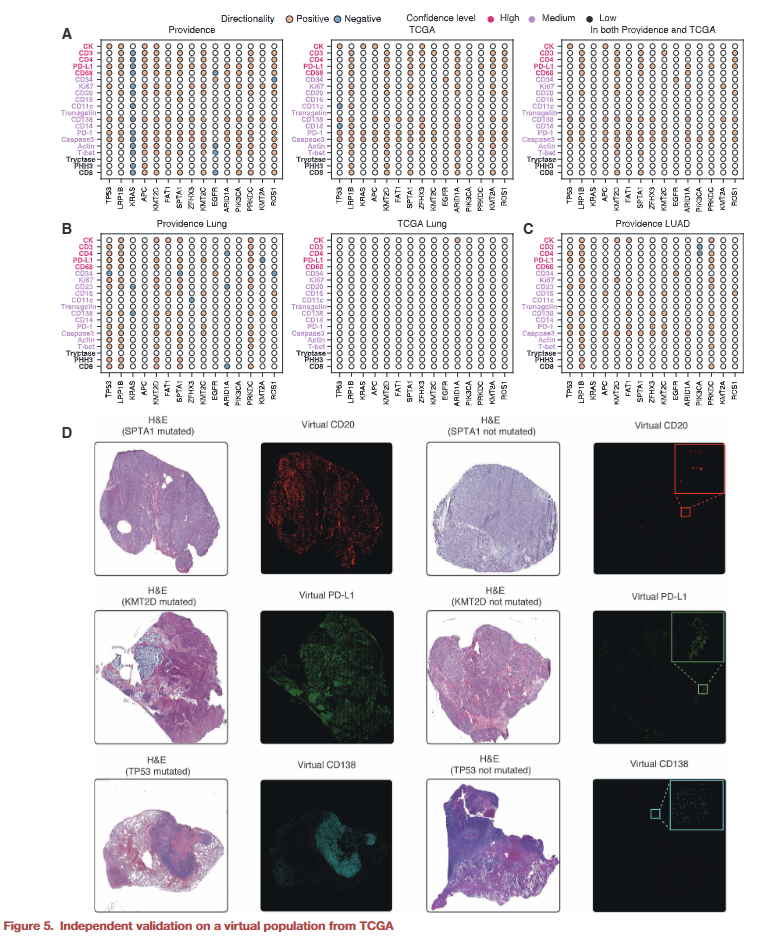
图5|独立队列中的一致性验证
空间与组合蛋白模式分析
除激活密度外,研究人员进一步分析了熵、清晰度等空间指标,并探索蛋白组合信号。结果显示,某些蛋白组合在揭示免疫状态和临床关联方面明显优于单一蛋白,反映了 TIME 中协同作用的生物学本质。
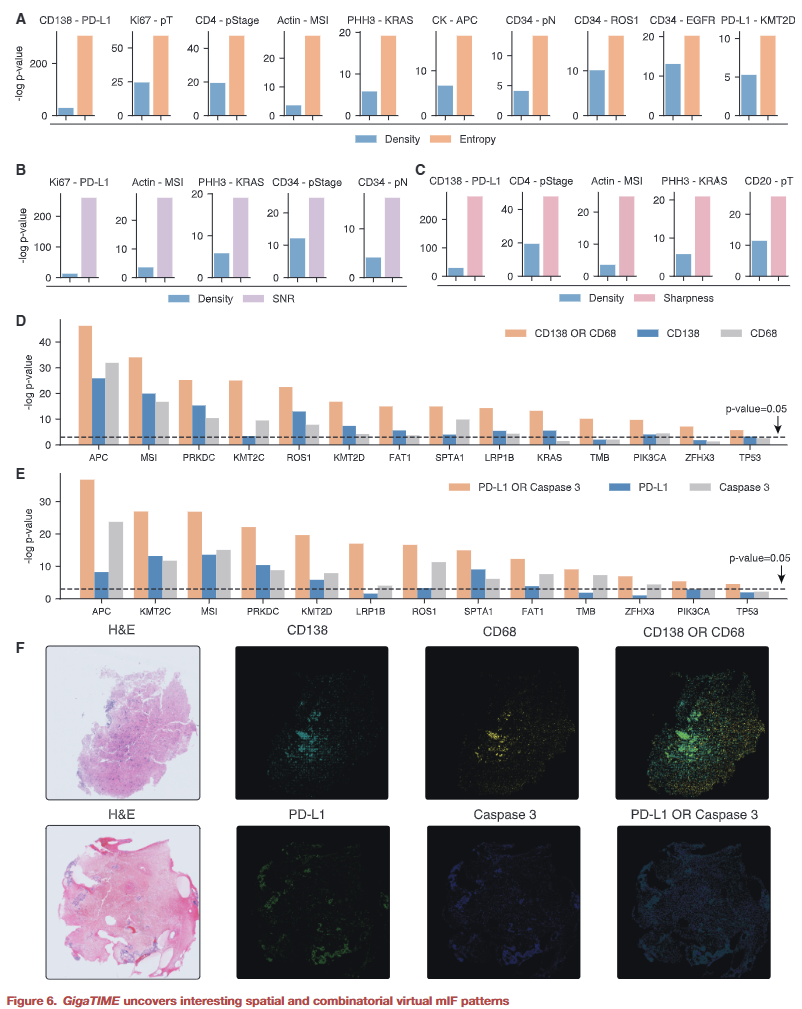
图6|空间指标与蛋白组合模式
讨论
GigaTIME 展示了一种通过多模态人工智能扩展肿瘤微环境研究规模的可行路径。该方法将低成本、广泛可得的 H&E 切片转化为高信息量的虚拟空间蛋白图谱,使得原本受限于 mIF 数据稀缺的大规模分析成为可能。研究结果表明,肿瘤免疫反应往往表现为多种免疫细胞和蛋白信号的协同变化,而非单一标志物的独立作用。通过构建虚拟群体,研究人员能够系统刻画这种协同结构,并为免疫治疗靶点发现、患者分层和精准肿瘤学研究提供新的工具。
整理 | DrugOne团队
参考资料
Valanarasu et al., Multimodal AI generates virtual population for tumor microenvironment modeling, Cell(2026)
https://doi.org/10.1016/j.cell.2025.11.016
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-12,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

