Nat. Mach. Intell. | 自监督图 Transformer 解码空间单细胞互作: 从细胞状态到生态位关联
Nat. Mach. Intell. | 自监督图 Transformer 解码空间单细胞互作: 从细胞状态到生态位关联

DrugAI
发布于 2026-01-06 14:29:35
发布于 2026-01-06 14:29:35
DRUGONE
细胞—细胞相互作用(CCI)是组织发育与器官功能维持的核心机制。尽管成像型空间转录组技术使单细胞分辨率的 CCI 研究成为可能,但现有分析方法仍受限于配体–受体覆盖不足、空间信息利用不充分以及模型可解释性弱等问题。研究人员提出 GITIII,一种轻量、可解释的自监督图 Transformer模型,将细胞视为“词”,其空间邻域视为“上下文”,通过解析细胞状态与其生态位(niche)之间的相关性来推断空间单细胞层面的相互作用。GITIII 能量化邻近细胞对受体细胞基因表达的影响,实现空间 CCI 可视化、基于 CCI 的细胞亚群划分以及 CCI 网络构建。该方法在多物种、多组织、多平台的四个空间转录组数据集中成功揭示了大脑与肿瘤微环境中的关键相互作用模式。

CCI 既可通过直接接触介导,也可经由旁分泌/自分泌信号影响基因表达与细胞行为,其强度受空间距离、局部微环境、信号分子表达等多因素共同调控。当前多数计算方法依赖已知配体–受体对,往往忽略同一细胞类型内部的状态异质性,并且难以刻画距离对相互作用强度的连续影响。图神经网络虽引入了空间结构,但多层架构常导致可解释性不足。鉴于此,研究人员提出从**“邻域如何塑造细胞状态”**这一角度出发,直接建模邻近细胞对受体细胞转录状态的影响,以获得更具生物学可解释性的 CCI 推断。
方法
GITIII 以成像型空间转录组数据为输入,包括基因表达、空间坐标和细胞类型注释。模型首先将每个细胞的表达分解为细胞类型表达与细胞状态表达;随后基于空间邻近关系为每个细胞构建局部子图,并通过单层图 Transformer学习中心细胞与邻居细胞之间的相互作用。模型在自监督框架下预测中心细胞的状态表达,同时生成影响张量,用于量化不同邻居细胞对各基因表达的贡献。基于该影响张量,研究人员开展单细胞层面的空间可视化、同类型细胞内的 CCI 驱动聚类、细胞类型层面的 CCI 网络构建以及不同条件间的相互作用强度比较。
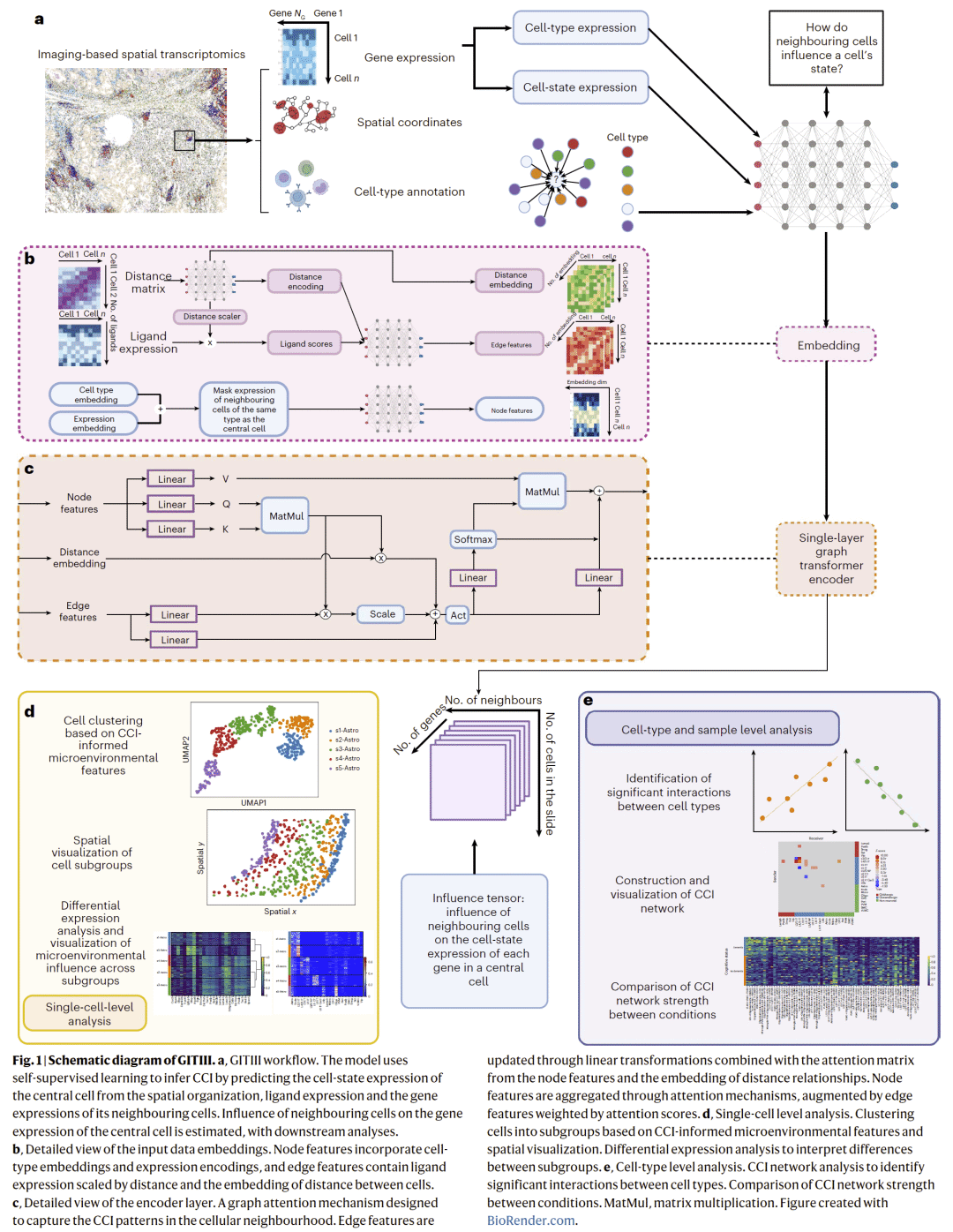
图1 | GITIII 的整体架构。
结果
GITIII 总体框架与空间相互作用建模
GITIII 能够在单细胞层面捕捉相互作用强度随距离衰减的总体趋势,同时识别出在一定距离范围内由信号表达差异驱动的强相互作用,说明空间接近性并非唯一决定因素。
小鼠大脑皮层中的空间 CCI 模式
在小鼠初级运动皮层数据中,GITIII 区分了不同细胞类型对之间的相互作用模式,并在星形胶质细胞和 L2/3 IT 神经元中识别出具有明确层特异分布的亚群。这些亚群的转录差异与其所处空间邻域高度一致,反映了微环境驱动的状态异质性。
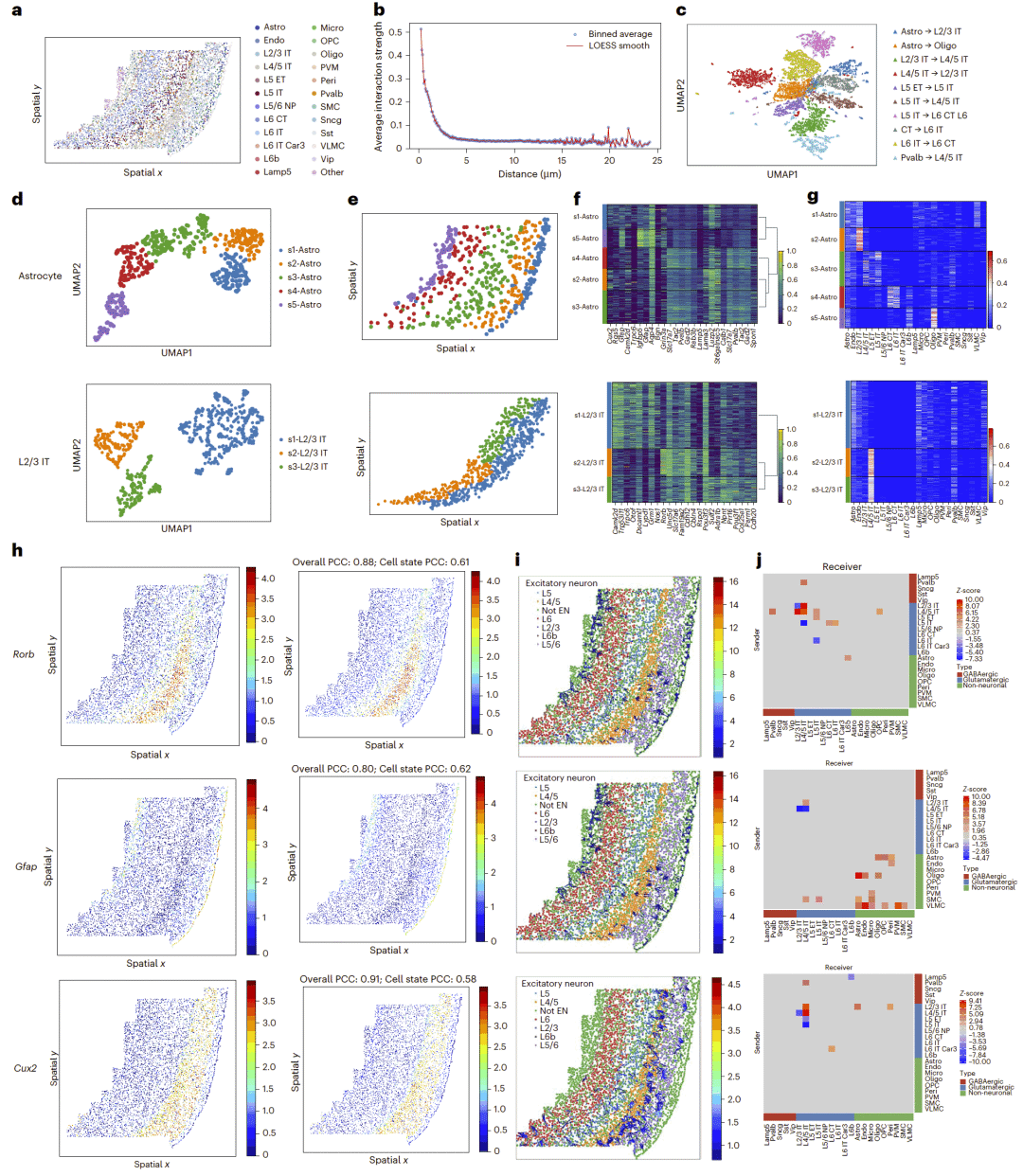
图2 | 小鼠大脑皮层中 CCI 模式及 CCI 驱动的细胞亚群划分。
与基因表达直接关联的 CCI 网络
通过构建基因层面的 CCI 网络,研究人员揭示了特定细胞类型对之间的相互作用如何上调或下调关键基因的表达。例如,神经元—神经元或胶质细胞—胶质细胞之间的相互作用与层特异基因表达变化高度一致。
阿尔茨海默病脑组织中的 CCI 重塑
在阿尔茨海默病患者脑组织中,GITIII 识别出小胶质细胞与神经元之间的异常相互作用模式,并发现与疾病风险基因相关的 CCI 网络在痴呆与非痴呆样本间存在系统性差异,提示免疫相关细胞在疾病进程中的重要作用。
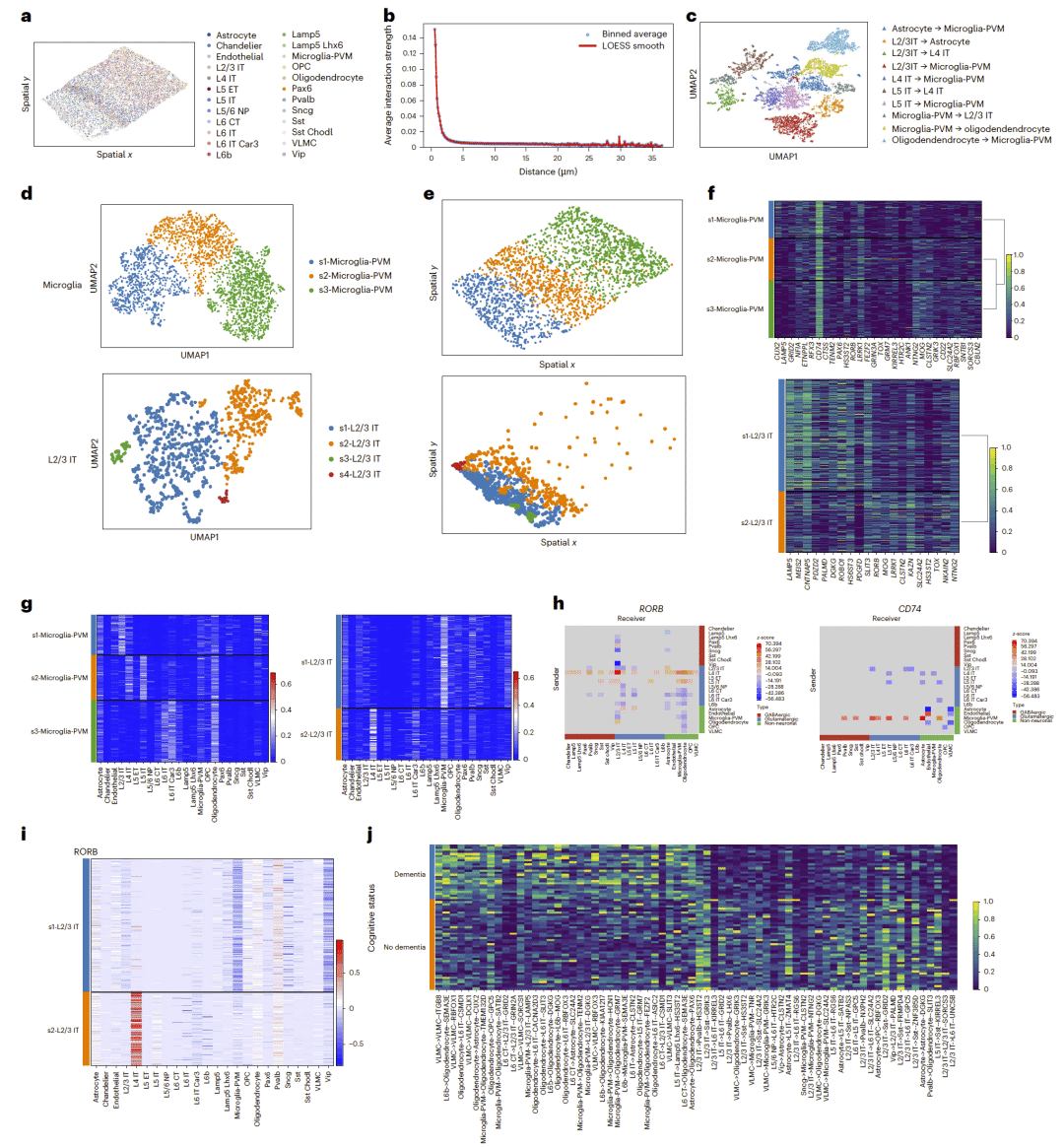
图3 | 阿尔茨海默病数据集中 CCI 网络差异及其与认知状态的关联。
肿瘤微环境中的空间互作解析
在非小细胞肺癌和乳腺癌数据中,GITIII 揭示了肿瘤细胞、免疫细胞和结构细胞之间复杂的双向调控关系。部分巨噬细胞和内皮细胞亚群显示出明显受肿瘤细胞影响的转录特征,反映了肿瘤驱动的微环境重塑。
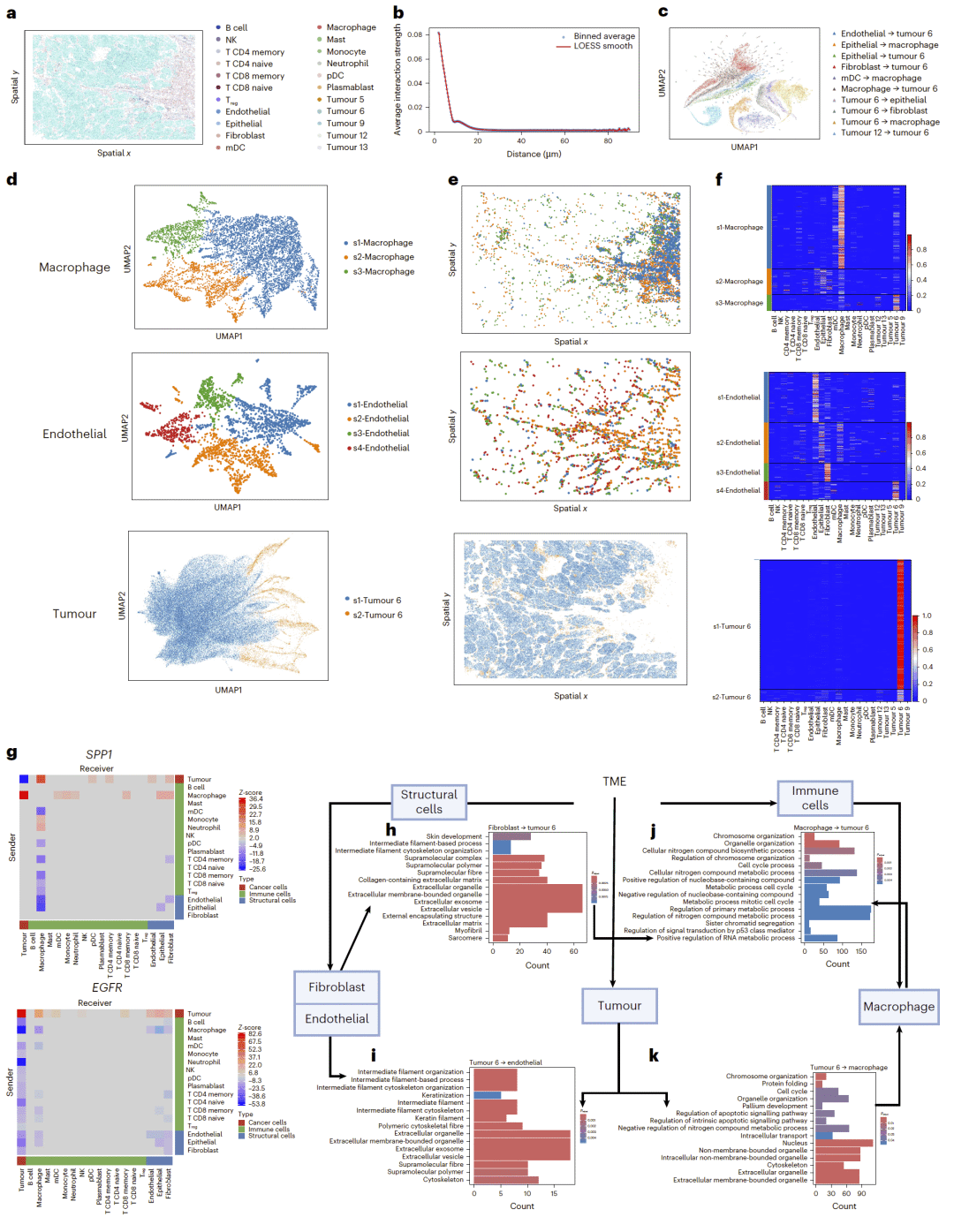
图4 | 肿瘤微环境中不同细胞亚群的 CCI 模式。
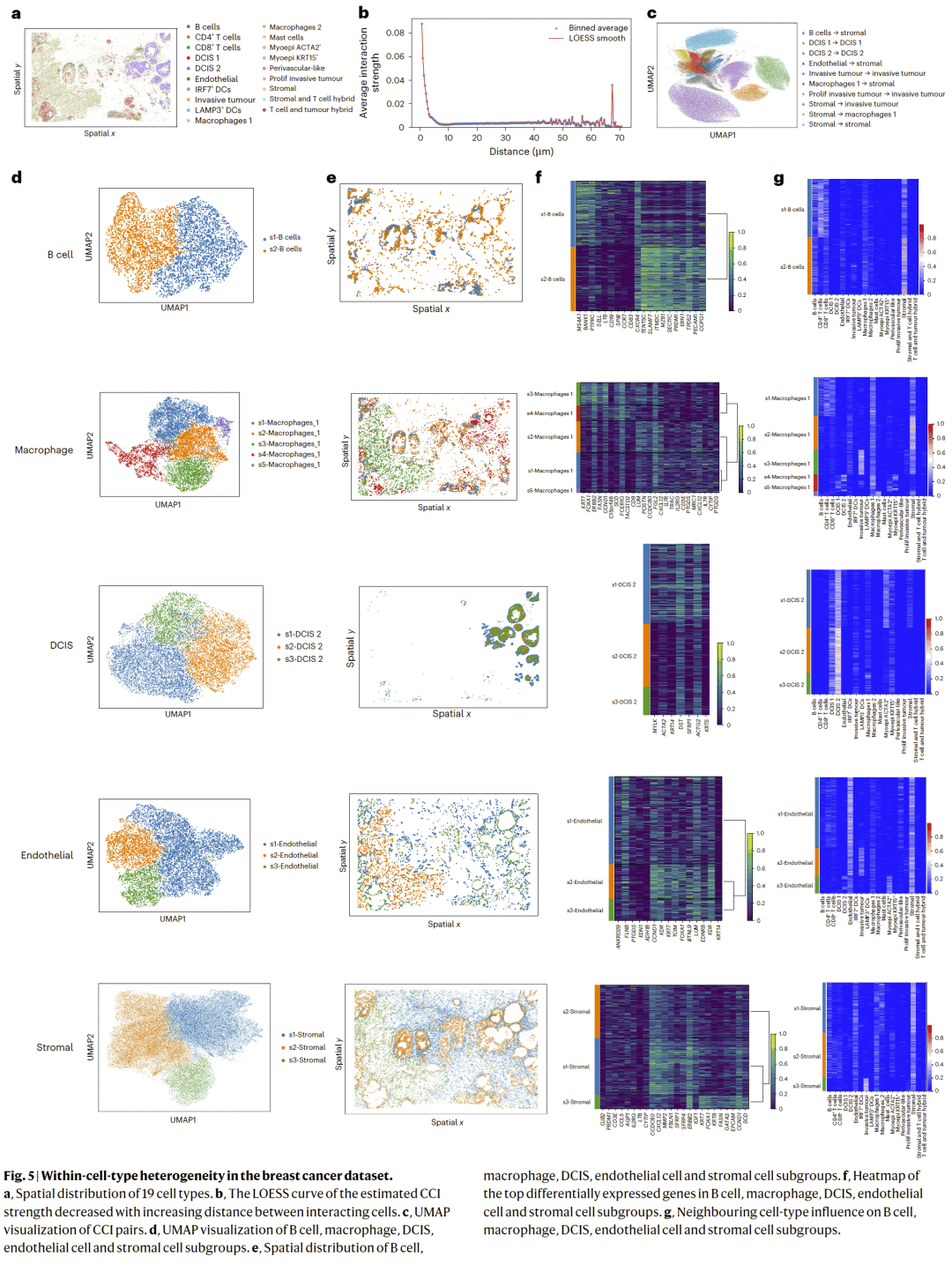
图5 | 乳腺癌数据集中的细胞类型内异质性。
跨数据集一致性与方法比较
跨物种、跨平台分析表明,GITIII 在不同数据集中能够一致地识别相似的 CCI 规律。与多种现有方法相比,GITIII 在单细胞层面和细胞类型层面均表现出更强的空间一致性与解释能力。
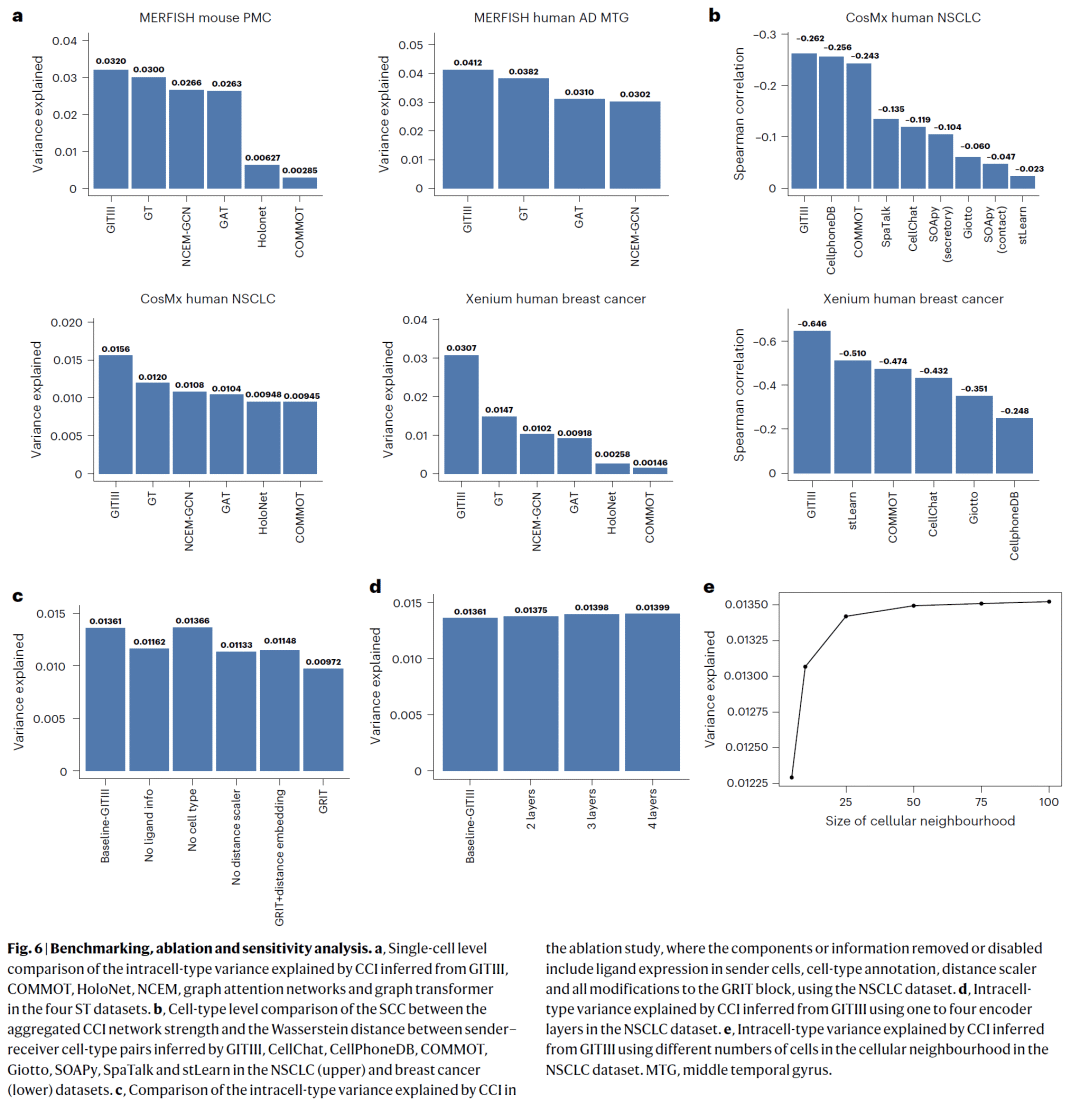
图6 | 跨数据集一致性分析与方法性能比较。
讨论
GITIII 提供了一种从“细胞状态—生态位相关性”角度理解空间 CCI 的新范式,避免了对配体–受体数据库的强依赖,并显著提升了模型的可解释性。该方法能够系统性揭示微环境如何塑造细胞状态,为理解脑功能、神经退行性疾病以及肿瘤微环境中的细胞互作提供了新的计算工具。未来,将该框架与多模态数据或时间维度结合,有望进一步拓展其在发育生物学和疾病研究中的应用。
整理 | DrugOne团队
参考资料
Xiao, X., Zhang, L., Zhao, H. et al. Inferring spatial single-cell-level interactions through interpreting cell state and niche correlations learned by self-supervised graph transformer. Nat Mach Intell (2025).
https://doi.org/10.1038/s42256-025-01161-0
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-04,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

