Nat. Biomed. Eng. | 华中科大团队推出深度学习与大语言模型融合的组学数据解读工作流
Nat. Biomed. Eng. | 华中科大团队推出深度学习与大语言模型融合的组学数据解读工作流

DrugOne
发布于 2026-01-13 15:41:22
发布于 2026-01-13 15:41:22
DRUGONE
大规模组学数据能够描绘细胞内分子调控的整体图景,但其生物学机制解释仍高度依赖人工经验与实验验证。为解决这一瓶颈,研究人员提出 LyMOI,一种将深度学习与大语言模型(LLM)推理相结合的组学解读混合工作流。LyMOI 通过图神经网络在跨物种蛋白互作知识图谱与多组学数据上进行分层训练,用于预测情境特异性的关键分子;随后,借助大语言模型生成类似研究人员思维链的机制解释,对候选分子的调控作用进行推理。以自噬为核心案例,LyMOI 系统性解析了约 1.3 TB 的转录组、蛋白组与磷酸化组数据,显著扩展了已知自噬调控网络,并在酵母与哺乳动物体系中实验验证了多个新型调控因子。该研究展示了一种将“数据驱动预测”与“知识驱动解释”融合的通用组学解读范式。

高通量测序和质谱技术的发展,使转录组、蛋白组与磷酸化组等多组学数据成为解析生命系统的基础工具。传统的组学解读方法主要依赖差异分析、功能富集和网络建模,其结果往往停留在统计相关性层面,难以直接提供机制层面的解释。
与此同时,自然语言处理领域的大语言模型在整合与推理生物医学知识方面展现出潜力,但其直接用于组学分析容易受到知识更新滞后和“幻觉”问题的限制。
研究人员认为,将深度学习的结构化预测能力与大语言模型的知识推理能力进行有机整合,有望在大规模组学数据中实现更接近研究人员思维方式的系统性解读。
方法
LyMOI 由两大核心模块构成:图学习预测模块与语言模型推理模块。
首先,研究人员构建了一个覆盖 562 个真核物种、约百万蛋白节点的跨物种蛋白互作知识图谱,并利用图卷积网络进行监督式预训练。在此基础上,通过教师–学生分层结构,引入多组学数据对模型进行情境特异性微调,用于预测潜在的关键分子。
随后,大语言模型在精心设计的提示策略下,对预测结果进行逐步推理,生成分子功能、调控关系及潜在机制的“机器思维链”,从而实现从数据到生物学解释的闭环。
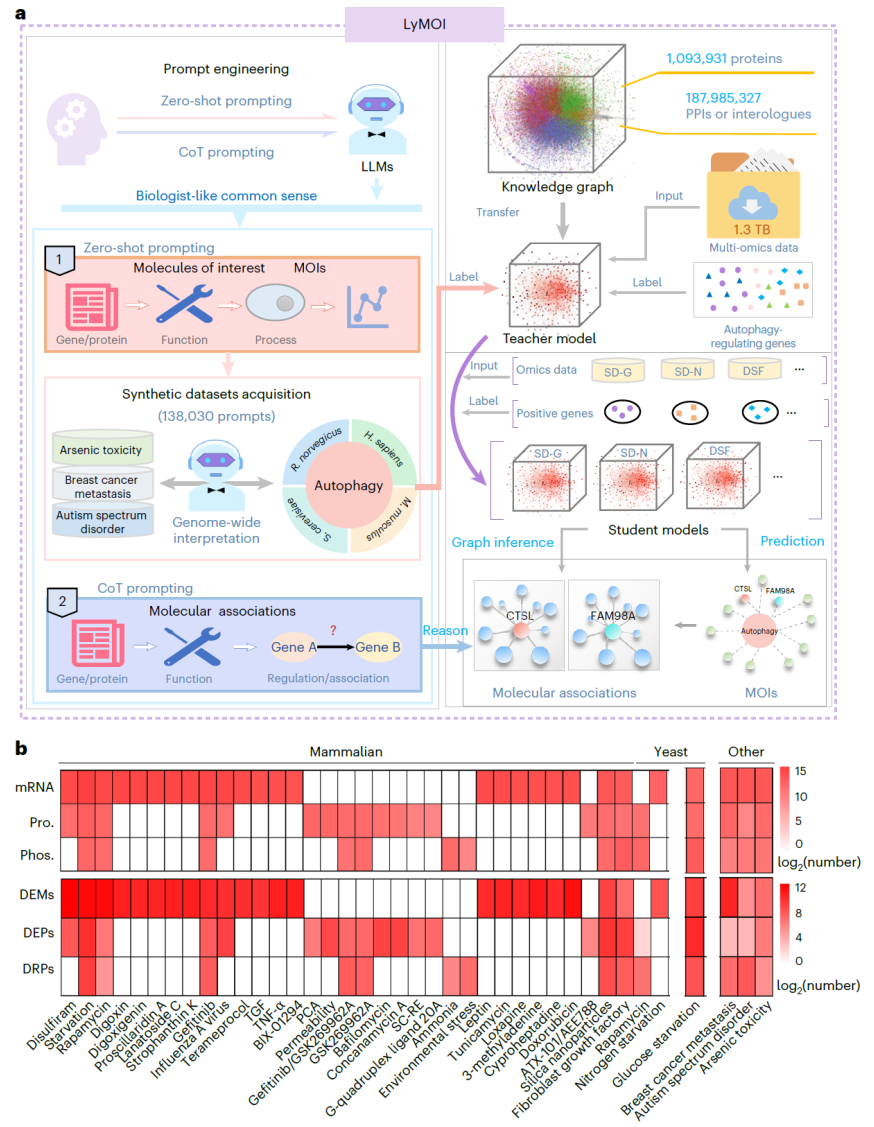
图 1:混合框架的整体工作流程。
结果
LyMOI 的整体框架与性能
LyMOI 能够在保持预测准确性的同时,大幅提升组学数据挖掘的生物学相关性。与传统差异分析相比,LyMOI 在多个场景中优先识别出更多已知调控因子。
大语言模型驱动的全基因组功能解读
通过零样本与思维链提示,大语言模型能够在全基因组尺度上对潜在调控因子进行功能判断,并在严格提示设计下有效降低不可靠推理。
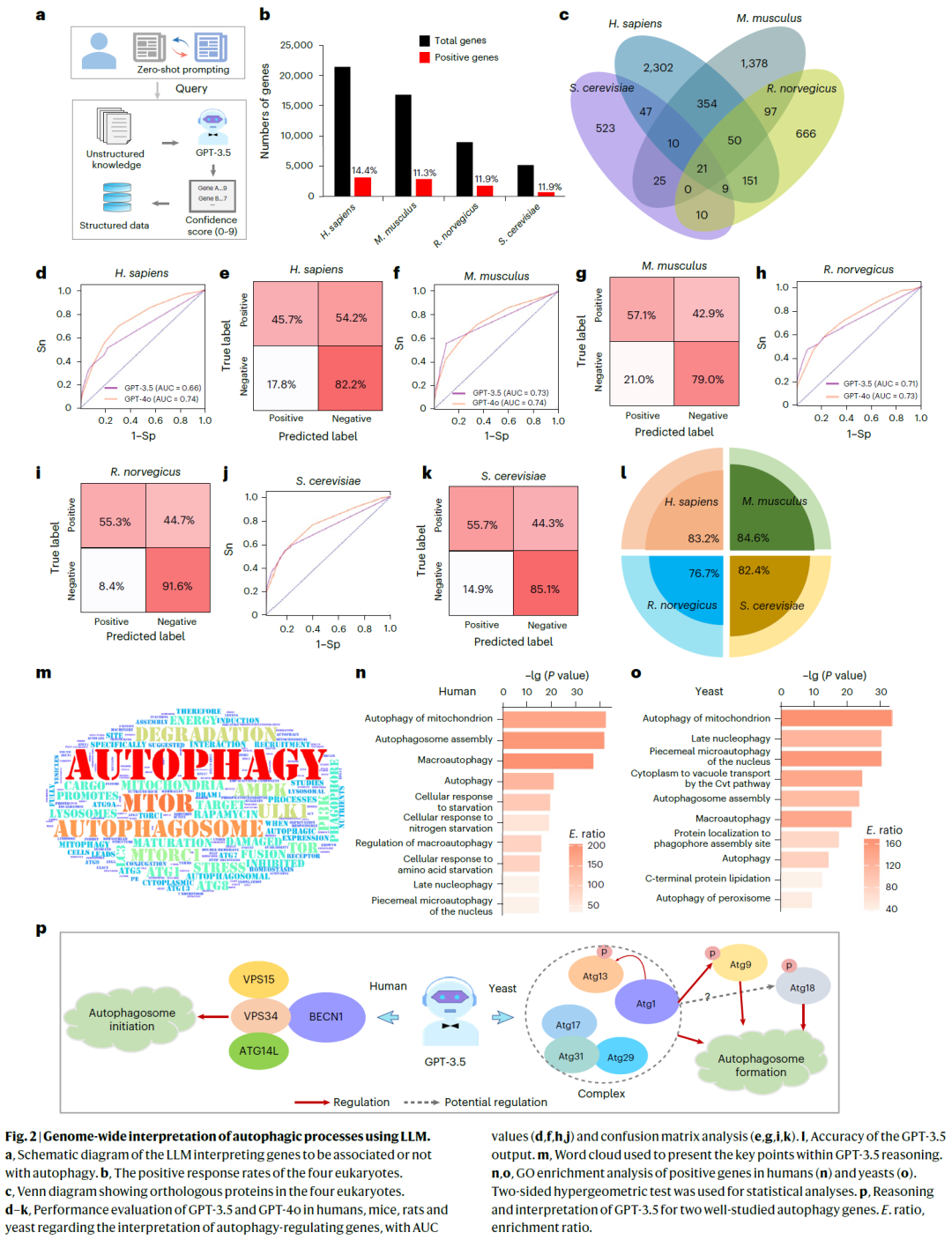
图 2:基于大语言模型的自噬过程全基因组尺度解读。
图学习显著扩展自噬调控因子
结合跨物种知识图谱与多组学数据,LyMOI 在多种刺激条件下显著扩展了情境特异性的自噬调控因子数量,优于多种传统机器学习方法。
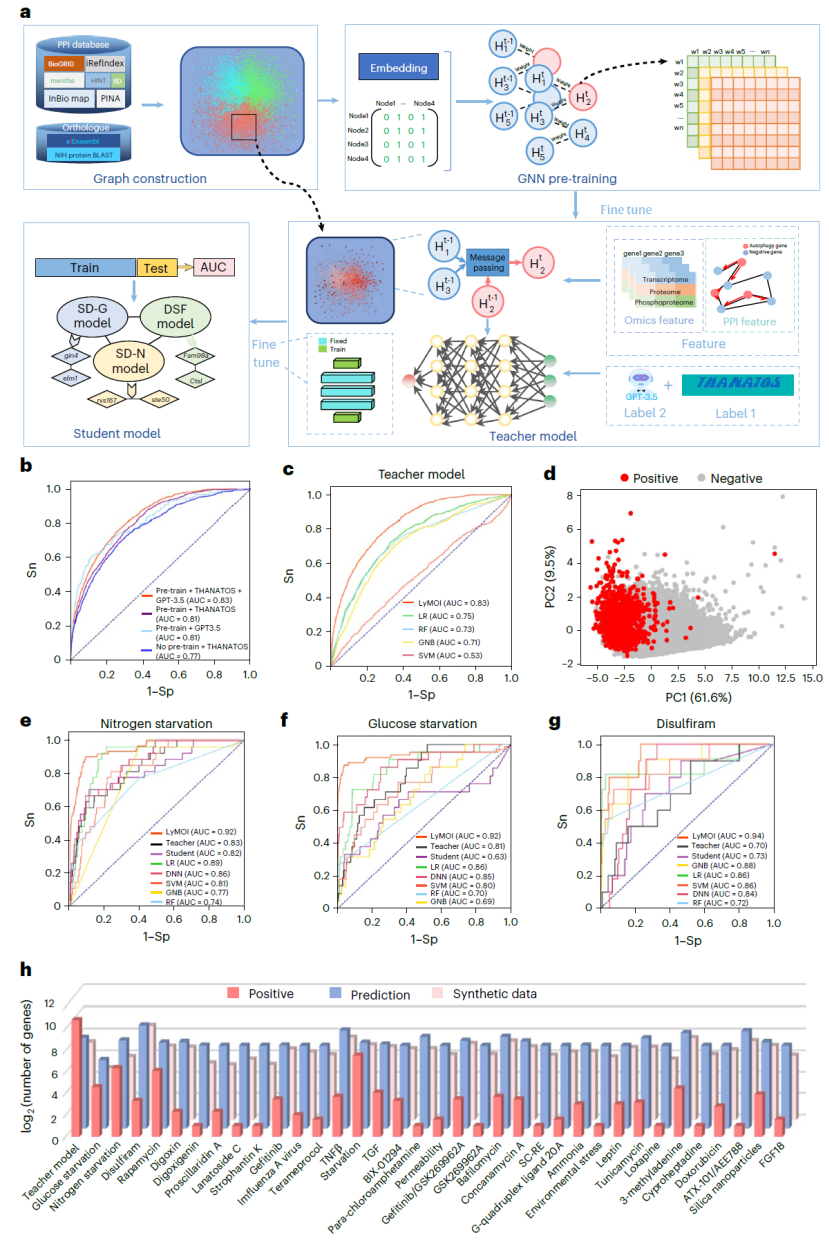
图 3:LyMOI 框架概览及其性能评估。
酵母体系中新型自噬调控因子的实验验证
在葡萄糖饥饿和氮饥饿条件下,LyMOI 成功预测并实验验证了多个此前未被充分表征的自噬调控因子,且其预测结果在功能富集层面表现出更高的生物学一致性。
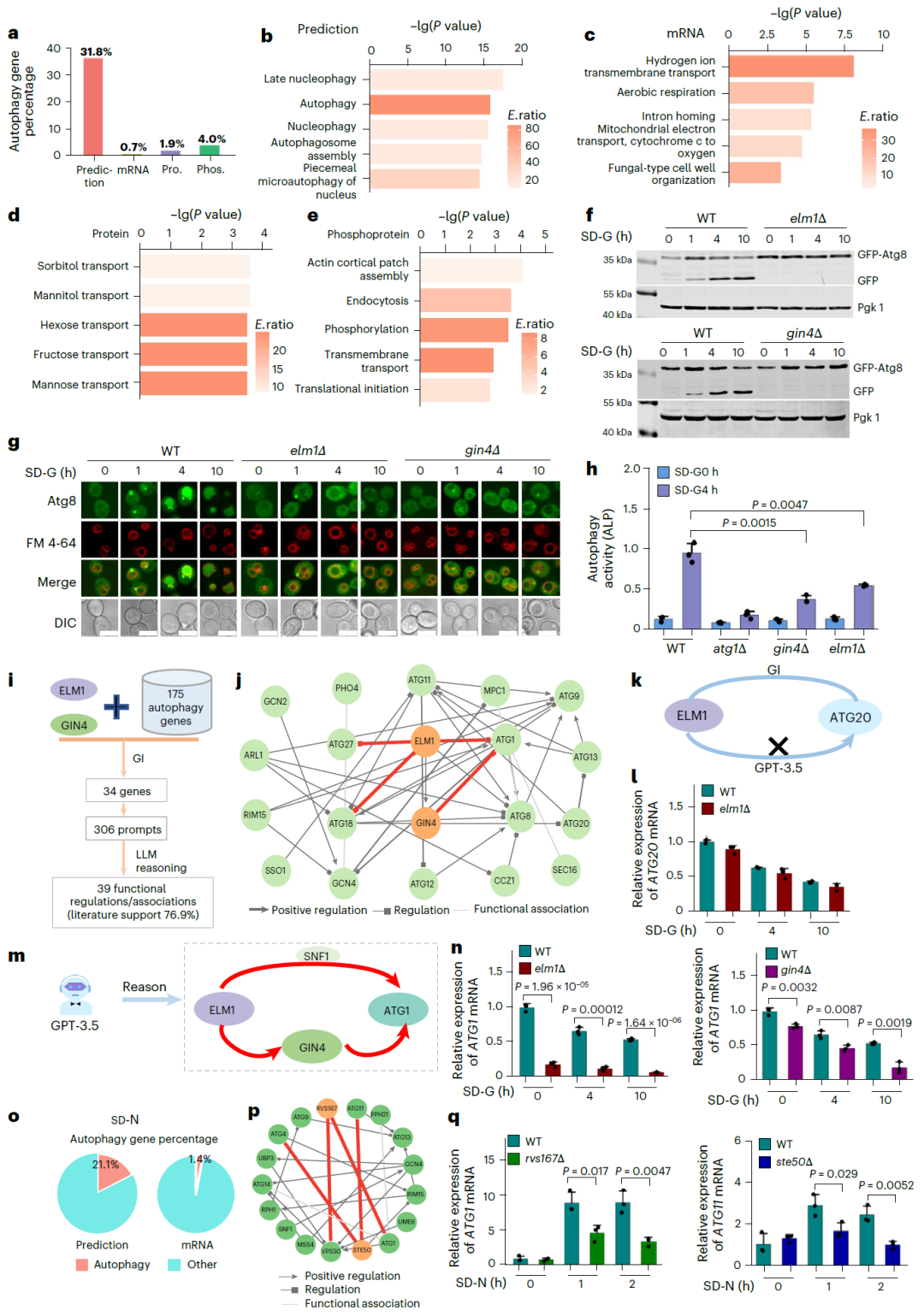
图 4:酵母自噬中新型关键调控因子的鉴定。
大语言模型辅助的分子机制推理
通过将图推断结果与语言模型推理相结合,LyMOI 构建了以关键调控因子为中心的分子调控网络,并给出机制层面的假设,为后续实验提供直接线索。
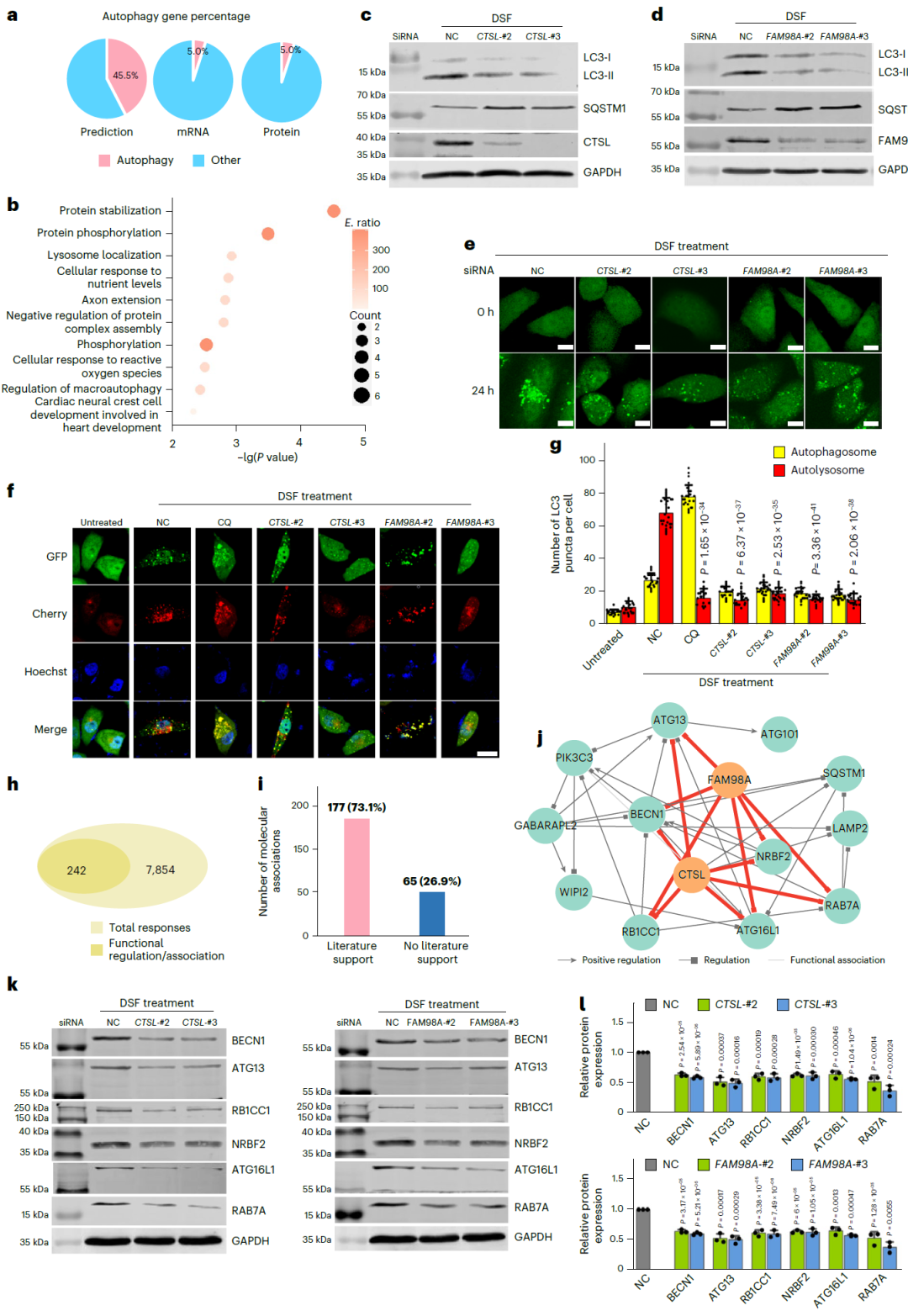
图 5:FAM98A 与 CTSL 在二硫仑(DSF)诱导的自噬激活中发挥关键作用。
哺乳动物体系中的应用:药物诱导自噬
在抗肿瘤药物处理模型中,LyMOI 揭示了新的自噬相关关键分子,并通过体内外实验验证其在自噬激活与肿瘤抑制中的作用。
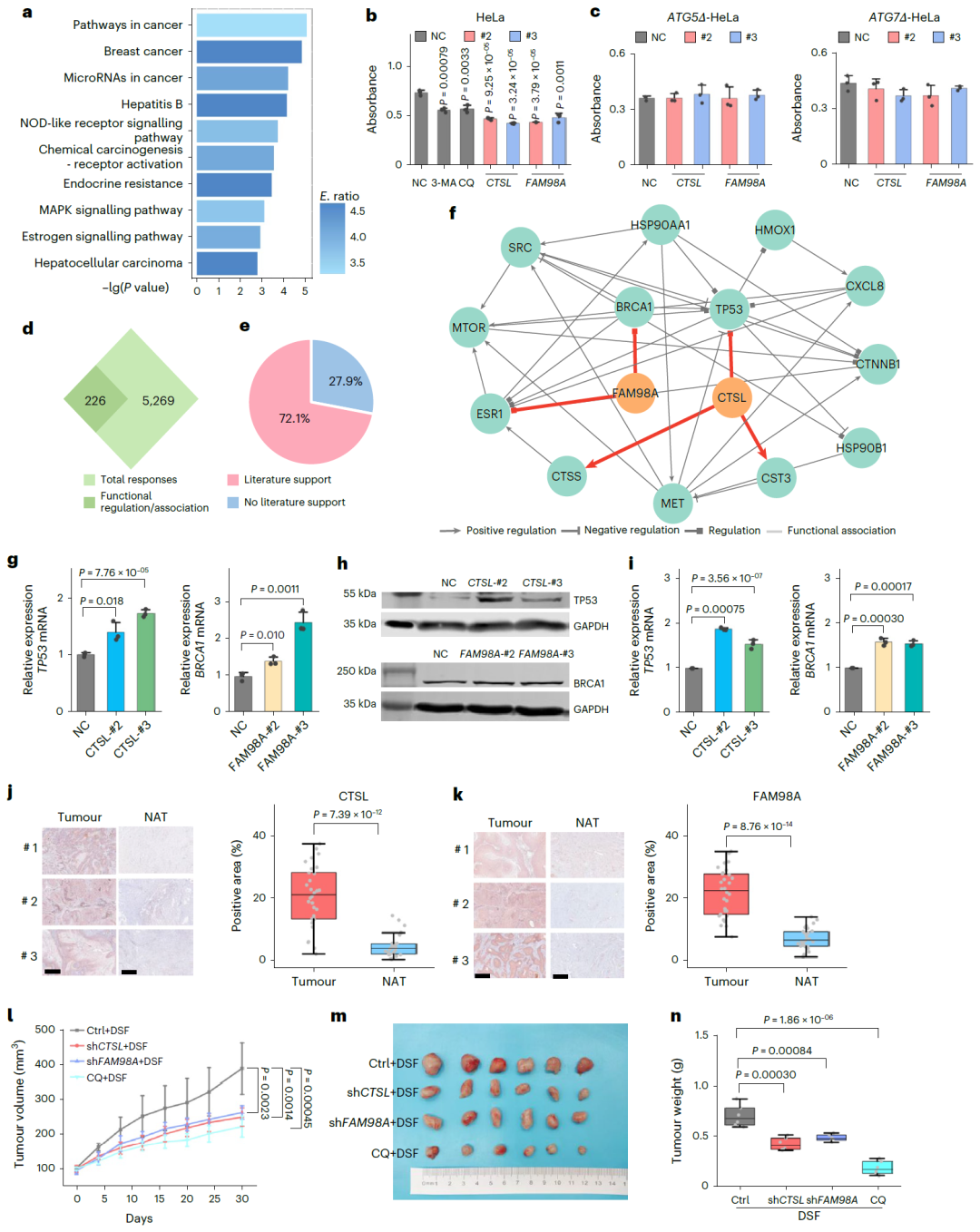
图 6:CTSL 与 FAM98A 通过自噬通路促进肿瘤细胞存活。
跨系统的可扩展性
LyMOI 被进一步应用于多种生物系统的组学数据解读,显示出良好的通用性与可扩展潜力。
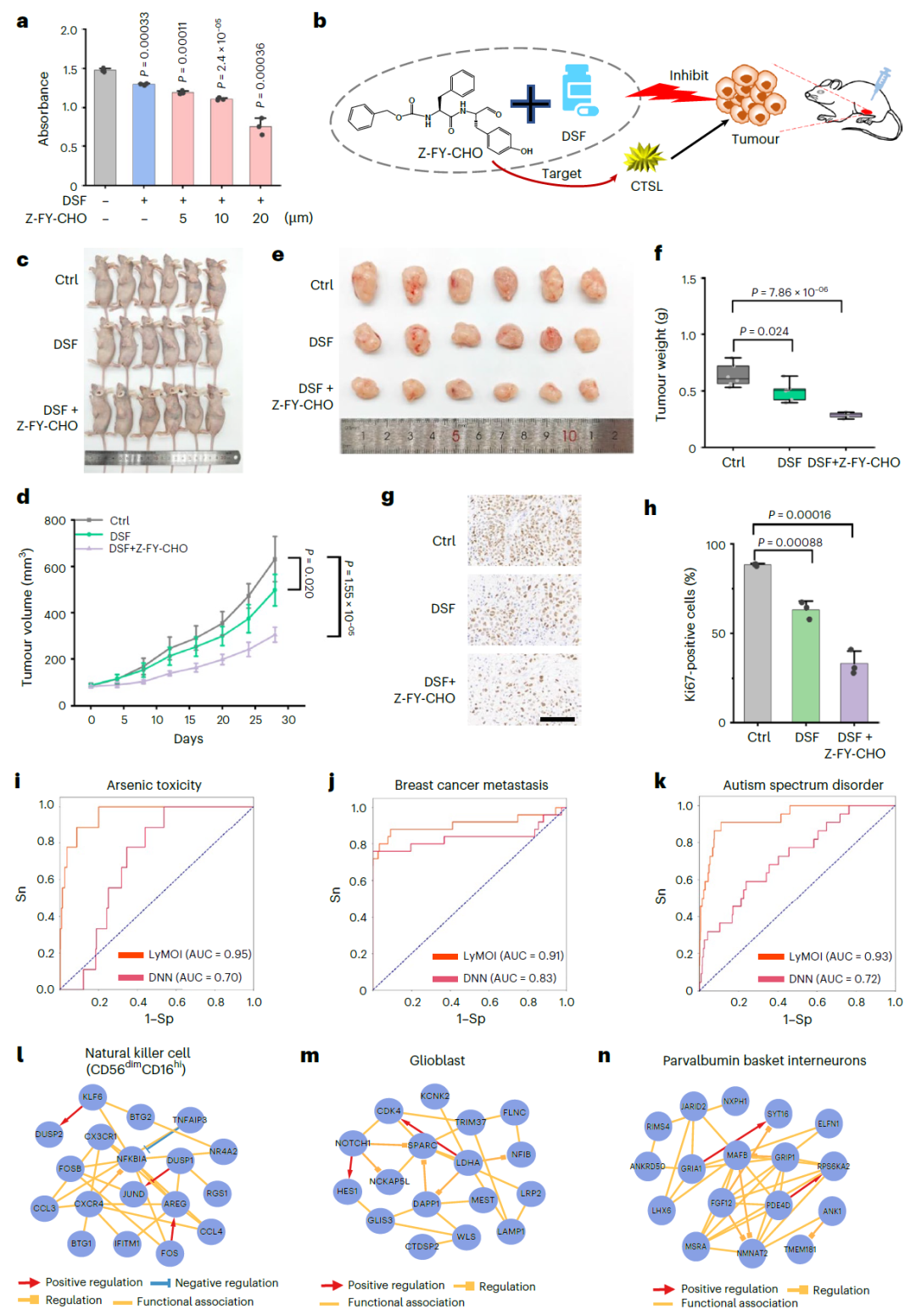
图 7:组合治疗策略及 LyMOI 的扩展应用。
讨论
该研究提出了一种将深度学习预测与大语言模型推理深度融合的组学解读框架,为大规模组学数据提供了更具机制导向的解释路径。LyMOI 不仅能够提升候选分子的生物学相关性,还通过类似研究人员思维的推理过程,为实验设计和机制假设提供直接支持。
研究人员也指出,大语言模型的推理结果仍需结合实验验证,且模型性能依赖于底层知识图谱和组学数据质量。未来,随着知识库扩展与提示策略优化,此类“深度学习 × 大语言模型”的混合范式有望成为系统生物学与精准医学中重要的通用工具。
整理 | DrugOne团队
参考资料
Tang, D., Zhang, C., Zhang, W. et al. A deep learning and large language hybrid workflow for omics interpretation. Nat. Biomed. Eng (2026).
https://doi.org/10.1038/s41551-025-01576-5
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-09,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

