ICLR2025 | CBGBench: 在蛋白-分子的复合物3D空间做完形填空
ICLR2025 | CBGBench: 在蛋白-分子的复合物3D空间做完形填空

基于结构的药物设计(Structure-Based Drug Design, SBDD)旨在生成能够与目标蛋白结合的潜在药物分子,并在生成式 AI 技术的推动下得到了极大加速。然而,由于多样化的实验设置、复杂的实现、难以复现的结果以及单一任务的局限性,这一领域仍缺乏系统性的理解。
首先,缺乏标准化会导致不同方法之间难以进行公平的比较,影响研究结论的可靠性。为了解决这一问题,作者提出 CBGBench,一个针对 SBDD 任务的综合基准,它将 SBDD 统一为生成式图补全(Generative Graph Completion)任务,类似于 3D 复合物结合图的“完形填空”问题(fill-in-the-blank)。CBGBench 通过对现有方法进行属性分类,构建了模块化、可扩展的框架,支持最新的生成方法,并确保公平的比较和评价。
其次,单一的 de novo 分子生成任务难以全面评估模型能力。为拓展 SBDD 的研究范畴,本文将生成模型适配到一系列核心任务,并将其视为图填充任务的子任务,包括:
- 从头分子生成
- 连接原子设计
- 分子片段补全
- 分子骨架生成
- 分子侧链优化
所有任务均以蛋白结合位点口袋结构为条件,确保任务的生物学相关性。 在评估方面,本文采用公平且全面的评价体系,从多个维度评估生成模型的表现,包括:
- 蛋白-配体相互作用分析
- 化学合理性
- 几何真实性
- 子结构有效性
此外,本文通过全面的实验分析提供了对模型表现的深入见解,并揭示了现有 SBDD 方法的局限性和改进空间。研究表明,
- 基于密度图的卷积神经网络设计方法仍然十分有效,这也许是图网络方法存在一定瓶颈。优化图神经网络架构是一个重要的改进方向
- 基于Diffusion的方法在整体性能上超过其他生成式建模,是目前主流的研究方向。
- 自回归方法需在训练时考虑到原子间键的几何关系,因为键的几何模式更容易被捕获,从而生成化学上有效的分子;否则效果都不佳。
- 先验知识仍然没有很好地被融入模型,有效整合物理和化学领域知识,融入模型设计仍是挑战,留有大量未来研究的空间。
- 大部分方法在先导化合物优化的任务上都可以直接应用,不过差距不明显,关于相关任务还有很大的提升空间。
最后,为降低入门门槛并促进该领域的发展,作者提供了统一代码库,涵盖数据预处理、模型训练、采样和评估,以支持研究人员和工程师更高效地开发和测试 SBDD 相关模型。
CBGBench 旨在建立一个标准化、可扩展的基准,促进结构基药物设计中更系统、公平和可复现的研究,为未来的 SBDD 研究奠定坚实基础。
引言
结构基药物设计(SBDD)广泛应用于新药发现,其目标是生成能够与目标蛋白结合的化学分子。近年来,生成式 AI 在 SBDD 任务中的应用日益增多,特别是基于深度学习的分子生成模型,得益于 Graph Neural Networks(GNNs) 和 Diffusion Models的发展,极大提升了分子设计的效率。然而,SBDD 领域仍存在如下挑战:
- 研究方法不统一:不同研究采用不同的任务定义和实验设置,导致难以进行公平的比较。
- 实验实现复杂且复现困难:许多方法代码封闭或依赖特定环境,缺乏标准化的评测体系。
- 任务设定单一:大多数研究仅关注 de novo 分子生成,而忽略了其他关键的药物设计任务,如Linker 设计、片段补全、骨架生成等。
为解决这些问题,作者提出 CBGBench,它提供:
- 统一任务定义:将 SBDD 归纳为生成式图补全(Graph Completion)问题,使不同方法可以在相同框架下进行比较。
- 模块化与可扩展性:支持多种 SBDD 任务,涵盖分子生成、Linker 设计、片段补全等关键任务。
- 系统性评估体系:采用多维度评估指标,确保模型在相互作用、化学性质、几何真实性和子结构有效性方面的表现。
CBGBench 评估模型以及任务定义
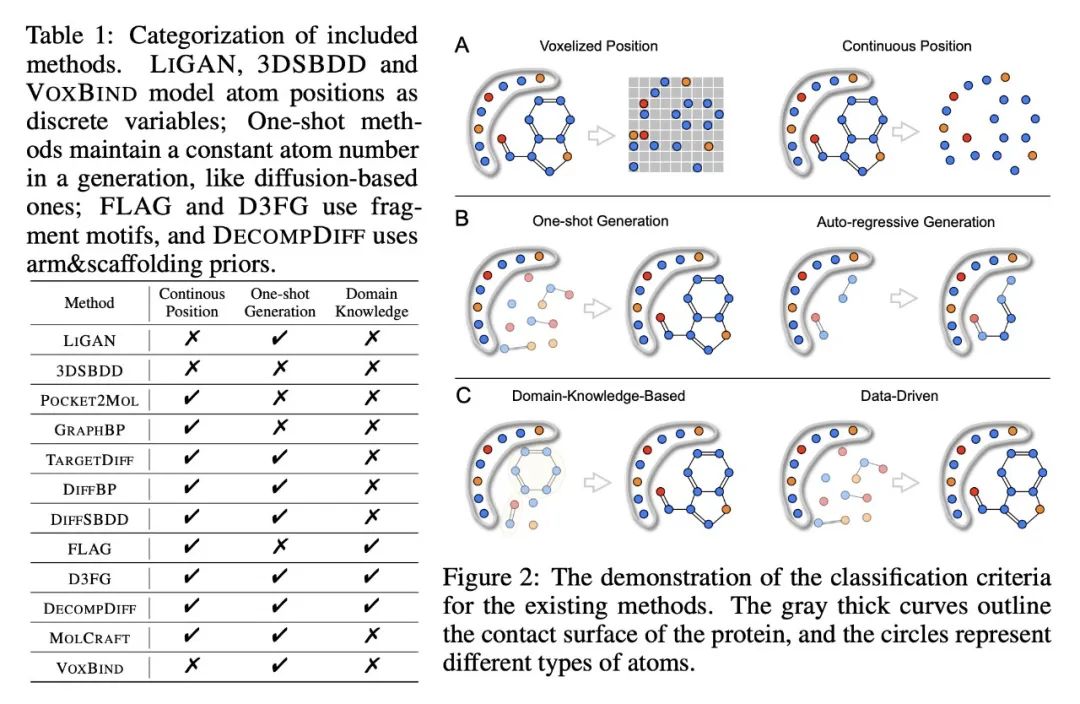
作者首先将现有的方法进行分类,根据以下三个标准对这些方法进行分类:
- 原子位置是否在连续的三维空间中生成,还是在体素化网格中生成。
- 生成过程是自回归的(auto-regressive)还是一次性生成的(one-shot)。
- 是否引入了领域知识,将额外的先验信息集成到模型中。
此外,作者将 SBDD 任务建模为3D 复合物结合图的“填空”任务(fill-in-the-blank),即在给定蛋白结合位点(Pocket)结构的情况下,生成符合特定条件的化合物。具体而言,作者定义了以下五个关键任务:
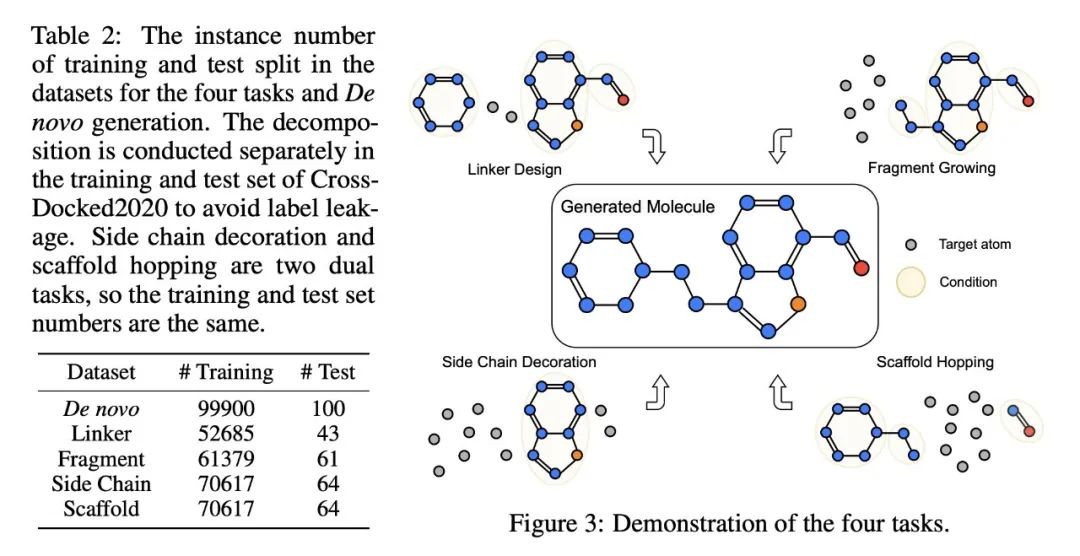
De novo 分子生成 (De novo Generation)
- 输入:蛋白结合口袋
- 目标:生成完整的新分子,与蛋白结合位点形成稳定的相互作用。
连接体设计 (Linker Design)
- 输入:蛋白结合口袋 + 片段 A 和片段 B
- 目标:生成化学连接体(Linker),使两个片段在 3D 结构上连接,同时维持生物活性。
片段生长(Fragment Growing)
- 输入:蛋白结合口袋 + 部分分子片段
- 目标:预测并补全缺失的分子部分。
骨架生成(Scaffold Hopping)
- 输入:蛋白结合口袋 + 侧链
- 目标:生成新的骨架(Scaffold),在保持配体关键特征的同时优化结合能力。
侧链优化(Sidechain Decoration)
- 输入:蛋白结合口袋 + 骨架
- 目标:优化分子的侧链,使其与蛋白结合更紧密,同时满足药物开发要求。
评估体系
作者提出了一套全面的评估体系,涵盖四大核心维度:
蛋白-配体相互作用质量
- 基于结合能(Binding Affinity)的评估
- 统计氢键、疏水作用等关键相互作用
化学性质
- 计算分子可合成性(Synthetic Accessibility)
- 评估分子药物相似性(Drug-likeness)
- 计算 LogP、Lipinski 规则等指标
几何真实性
- 评估 3D 结构合理性(如键长、键角等)
- 分子-蛋白复合体构象的稳定性(是否存在clash等)
子结构有效性
- 验证生成的分子是否包含关键药效团
结果分析
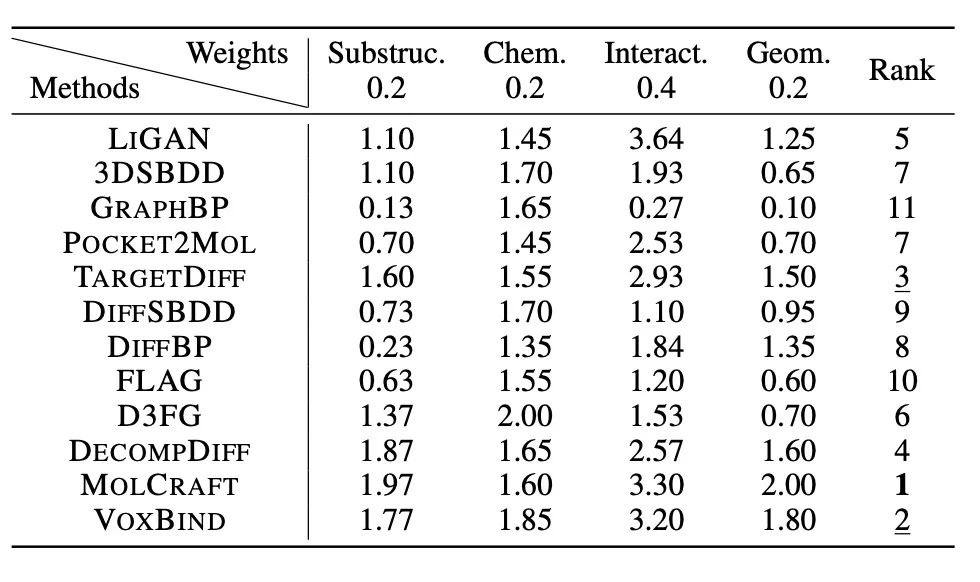
作者的实验表明:
- 基于 CNN 的方法(如 LIGAN 和 VOXBIND)具有高度竞争力,特别是在相互作用方面,这也解释了为什么这些方法在药物设计和分子生成领域近年来依然占据主导地位。这在一定程度上归因于 CNN 在利用单个滤波器感知多体模式方面相比 GNN 更具优势。因此,这也激励了进一步研究 GNN 在 3D 点云中的应用,开发能够与 CNN 相媲美的架构。
- 在基于 GNN 的方法中,MOLCRAFT 实现了最佳的整体性能,而 TARGETDIFF 紧随其后。与之相比,作为 TARGETDIFF 的变种,集成了领域知识的 DECOMPDIFF 和 D3FG 的性能有所下降。这表明当前引入的物理化学先验知识很难提升生成分子的质量。有效地将领域知识整合到模型中以引导生成结构合理的分子仍然是一个挑战。例如,生成的分子中原子冲突(atom clashes)是常见的问题。尽管 DECOMPDIFF 采用了先验指导来解决这一问题,但仍未完全解决。将领域知识集成到训练过程中可能有助于缓解这一问题。
- 只有 POCKET2MOL 作为自回归方法取得了具有竞争力的结果,作者归因于以下原因:首先,它利用化学键约束原子沿化学键有序生长,而不是像 DIFFBP 那样基于与口袋的距离进行生长。其次,它同时预测键类型,并通过对比学习,采样原子位置的正负实例,将真实和虚假的键作为正负样本,这一点 FLAG 没有充分考虑,从而增强了模型感知化学键模式的能力。因此,作者认为使自回归方法能够成功捕捉化学键的模式是至关重要的。
- 骨架生成在子任务中(Scaffold Hopping)是最具挑战性的任务,因为生成分子相对于参考分子的改进非常有限;而连接体设计(Linker Design)相对来说是最容易的任务。
- 直接应用这些方法在优化任务中,虽然效果理想,但是仍有很大的改进空间,这是由于在大多数情况下,生成的分子并没有被优化(binding affinity相关指标没有提升)。一些前沿技术,如 DPO 和 ITA ,可以用来增强优化后的分子在模型中的监督占比。
代码开源与未来展望
为降低入门门槛,作者提供 统一代码库,包括:
- 预处理数据集
- 训练和评估脚本
- SBDD 相关的生成模型
未来,作者计划进一步扩展任务类型、集成更多基于 AI 的 SBDD 方法、提供更高效的计算优化策略。作者希望 CBGBench 能够成为 SBDD 研究领域的标准基准,推动AI辅助的基于结构药物设计的发展。
本文作者为西湖大学Haitao Lin,Yufei Huang, 深势科技Guojiang Zhao,Zhifeng Gao,及华盛顿大学Odin Zhang等;通讯作者为Stan Z. Li. 被ICLR2025接受为spotlight paper。
参考资料
Lin, H., Zhao, G., Zhang, O., Huang, Y., Wu, L., Liu, Z., Li, S., Tan, C., Gao, Z. and Li, S.Z., 2024. CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph. CoRR.
https://openreview.net/forum?id=mOpNrrV2zH
代码
https://github.com/EDAPINENUT/CBGBench
