Nat. Commun. | 深度学习赋能RNA研究,NuFold实现精确RNA结构预测
Nat. Commun. | 深度学习赋能RNA研究,NuFold实现精确RNA结构预测

DRUGAI
今天为大家介绍的是来自美国普渡大学Daisuke Kihara团队的一篇论文。RNA不仅作为信使RNA在基因表达过程中发挥信息传递的重要作用,还作为非编码RNA参与各种生物学功能。理解其功能的机械机制需要三级结构信息;然而,实验测定RNA三维结构既昂贵又耗时,导致RNA序列和结构数据之间存在巨大差距。为了应对这一挑战,作者开发了NuFold,这是一种新型计算方法,利用最先进的深度学习架构来准确预测RNA三级结构。NuFold是一个端到端训练的深度神经网络,可以从输入序列预测输出结构。NuFold采用核碱基中心表示法,使核糖环构象更加灵活。基准研究表明,NuFold明显优于基于能量的方法,并且与现有最先进的基于深度学习的方法相比表现相当。NuFold在构建RNA的正确局部几何结构方面具有特殊优势。对NuFold流程中各个组件的分析表明,通过利用宏基因组序列进行多序列比对并增加recycling次数,可以提高其性能。NuFold还能够通过连接输入序列来预测RNA的多聚体复合物结构。

核糖核酸(RNA)是生物体的基础分子。除了作为转录中使用的信使RNA的核心角色外,RNA分子还以非编码RNA(ncRNA)的形式执行各种生物功能。ncRNA包括核糖体RNA(rRNA)和转运RNA(tRNA),这些可能是最广为人知的ncRNA,以及小核RNA(snRNA)、小核仁RNA(snoRNA)、环状RNA(circRNA)和核酶,它们参与基因调控和修饰等功能。RNACentral数据库目前包含超过三千万条ncRNA序列。ncRNA在药物设计中也受到关注,因为新药可能被设计用来抑制或模仿功能性RNA的活性。
要理解ncRNA的功能机制,获取三级结构信息至关重要。然而,由于实验确定的RNA结构稀缺,作者对RNA结构的了解仍然有限。蛋白质数据库(PDB)包含大约6000个条目,其中包括RNA分子,这仅占整个PDB条目的约3%。在Rfam数据库(v14.8)列出的4094个RNA家族中,仅有124个(3.0%)在PDB中有一个或多个对应结构。在这里,作者介绍了NuFold,一种使用端到端深度网络架构的从头开始RNA结构预测方法。该方法接收目标RNA序列并通过一个全面训练的单一网络生成三级结构模型。
模型架构
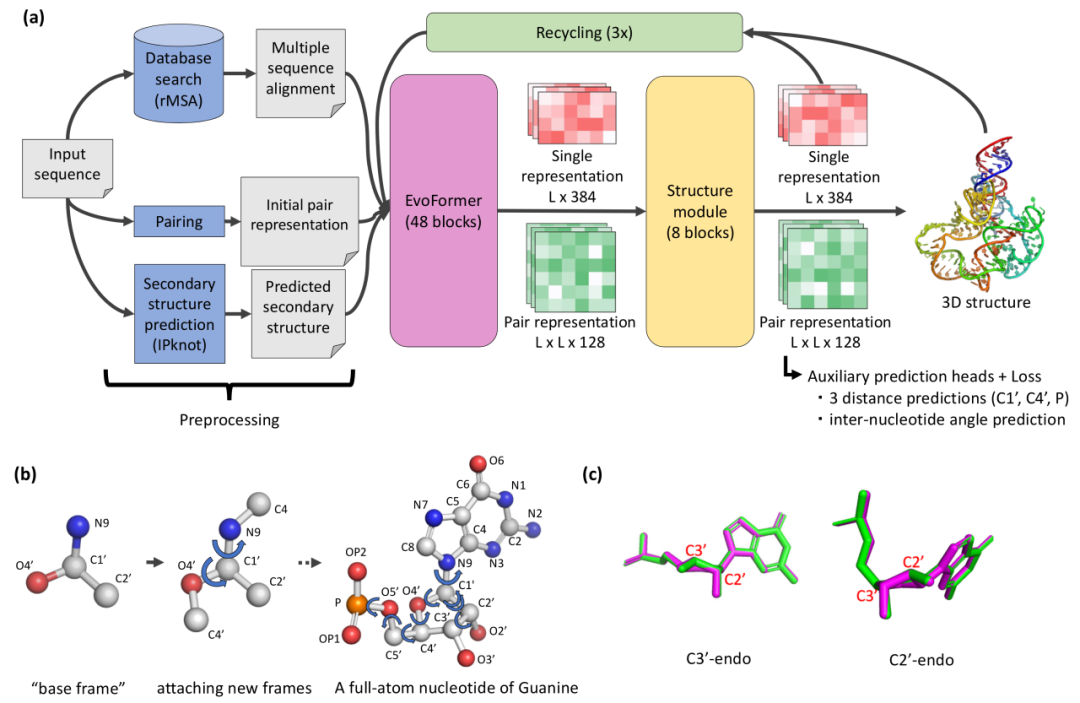
图 1
图1a描述了NuFold的架构。NuFold是一个端到端网络,可以从RNA序列预测其全原子三级结构。NuFold与AF2共享深度学习架构的基本框架,该框架由三个主要模块组成:第一个模块处理输入数据,第二个模块是Evoformer块,用于生成为目标序列收集的多序列比对(MSA)和残基对信息的嵌入,最后一个模块是结构模块,用于构建目标RNA的三维(3D)结构模型。对于输入的RNA序列,使用rMSA生成MSA,并使用IPknot生成预测的二级结构。结构模块利用图1b所示的灵活核苷酸碱基中心表示。
在NuFold的结构模块中,作者用以下四个原子定义碱基框架:O4'、C1'、C2'和碱基的第一个氮原子(C和U的N1,G和A的N9)。所有其他原子被分为十个框架,然后使用预测的框架之间键上的扭转角度作为指导原则进行迭代连接(图1b)。与AF2对氨基酸所做的一样,这些定义在RNA中被硬编码。这种RNA核苷酸的表示可以重现核苷酸构象的完整动态。
在图1c中,作者展示了核苷酸的两种不同构象,C3'-endo和C2'-endo糖构象。这两种构象在已知RNA结构中分别占约82%和10%的核苷酸,它们的结构差异在均方根偏差(RMSD)约0.3至0.5Å。使用NuFold结构表示,这两种构象被精确构建,C3'-endo和C2'-endo构象的RMSD分别为0.04Å和0.03Å。
NuFold在2860条RNA链上进行了训练,在训练期间对48条链进行了验证,并主要在36个测试RNA上进行了测试。使用常用的80%核苷酸序列同一性截断值,训练集、验证集和测试集中的条目是非冗余的。这些条目来源于截至2022年2月28日从蛋白质数据库(PDB)下载的RNA条目。作者还使用了来自bpRNA-1m数据集的11101个条目作为自蒸馏数据集,该数据集在训练期间与训练数据集合并。更多细节可在方法部分找到。
结构预测性能
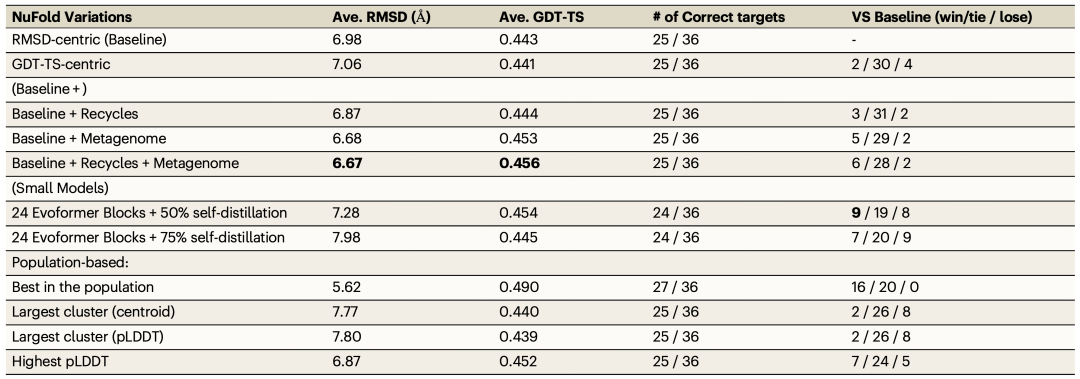
表 1
表1总结了不同NuFold模型对36个测试目标的结构预测准确性。前两个结果,以RMSD为中心和以GDT-TS为中心,代表了两个网络模型的结果。前者选自在验证数据集上表现出与天然结构最小平均均方根偏差(RMSD)的训练步骤,而后者则选自在验证数据集上表现出最高平均全局距离测试-总分(GDT-TS)的训练步骤。RMSD是针对核苷酸中的C1'原子计算的。GDT-TS测量预测结构与天然结构之间的整体结构相似性,范围从0到1,1表示与天然结构完全一致。
这两个网络模型在测试集上表现相似,将25个RNA目标折叠在6Å的RMSD范围内,并且在平均RMSD和GDT-TS值上表现相当。在这些模型生成的结构直接比较中,以RMSD为中心的模型为四个目标生成了更低RMSD(超过0.5Å)的结构,而以GDT-TS为中心的模型为两个目标生成了更低RMSD的结构。对于剩余的30个目标,两个模型生成的结构在0.5 Å RMSD以内,在表1中标记为平局(tie)。作者将以RMSD为中心的模型指定为基准线,因为与以GDT-TS为中心的模型相比,它表现更好,并用它与NuFold的其他变体进行比较。
探究各因素对于模型性能的影响
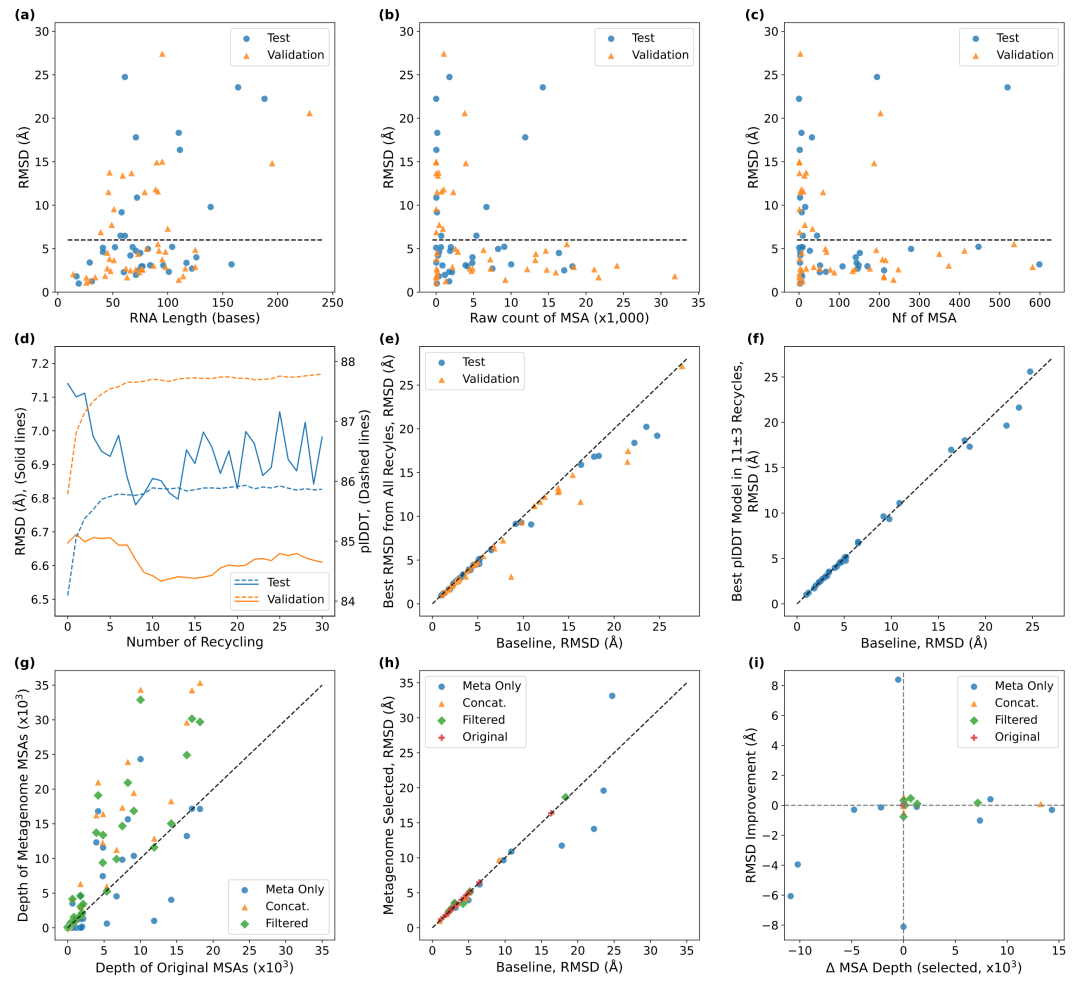
图 2
在图2中,作者研究了目标长度和输入MSA深度如何影响基准模型的建模结果。显示了测试集(蓝色圆圈)和验证集(橙色三角形)的结果。在图2a中,作者观察到目标长度与建模准确性之间存在适度的相关性。测试集中所有短于50个核苷酸(nt)的小RNA目标都在6 Å RMSD内折叠,而对于超过100 nt的目标,RMSD较大的目标比例增加。这一趋势在测试集和验证集中都有观察到。
图2b、c研究了输入MSA深度的影响,分别从MSA中序列的原始计数(图2b)和MSA的有效计数数量(Nf)(图2c)方面进行研究。MSA的有效计数是MSA中具有彼此低于截断值相似性的非冗余序列的数量。在这里,作者使用80%作为序列同一性的截断值来考虑两个序列是否是冗余的,这是rMSA流程中使用的标准截断值。作者观察到浅层MSA在建模结果中表现较差的弱趋势。在测试集中23个MSA有效计数少于100的目标中,39.3%的目标RMSD大于6Å。在验证集中,31个有效计数少于100的目标中,45.2%的目标RMSD大于6Å。
Recycle次数对于模型性能的影响
回到表1,在标记为"基准+"的中间区块,作者尝试通过增加宏基因组序列来增加MSA深度,并通过增加循环次数来提高NuFold基准模型的建模准确性。
据相关文献报道,在蛋白质情况下,增加循环次数通常会提高结构预测准确性。循环将前一次运行的原子坐标输出和内部表示向量与原始输入混合,并将它们用作网络的输入(图1中的绿色框)。在训练期间,为每个批次随机选择零到三次循环。在推理中,作者在基准模型中使用了三次循环。在这里,作者研究了预测结构的RMSD中最多30次循环的效果。
在图2d中,作者检查了平均RMSD和pLDDT如何在30次循环迭代中变化,pLDDT是网络对模型准确性的自我评估的度量。在测试(青色)和验证(橙色)案例中,随着应用更多循环,RMSD(实线)中的平均模型准确性得到改善,但仅限于大约10次(在测试集中,最低平均RMSD是在第八次循环中实现的,而在验证集中是在第十一次)。但在那之后,随着更多循环迭代,RMSD再次变差。另一方面,pLDDT急剧增加到大约五次循环迭代,并随着更多循环的累积而持续上升(图2e、f中的虚线)。因此,显然模型在循环的后期迭代中变得过度自信。
在几乎所有情况下,与执行三次循环的基准模型相比,在延长的循环迭代中生成了更好的模型(具有更小的RMSD)(图2e)。延长循环中最佳模型的平均RMSD为6.38Å,比基准模型(6.98Å)改进了0.6Å。然而,选择最佳结构并不容易,因为pLDDT几乎单调地随循环迭代增加,并不能精确指示最佳结构(图2d)。在图2f中,在测试集上,作者从第8到14次循环中选择了最高pLDDT结构,这是在第11次循环(在验证集中产生最低平均RMSD)周围±3次循环。这种策略使平均RMSD略有改善,达到6.87Å,比基准改善了0.11Å。
二级结构预测准确性对模型建模的影响
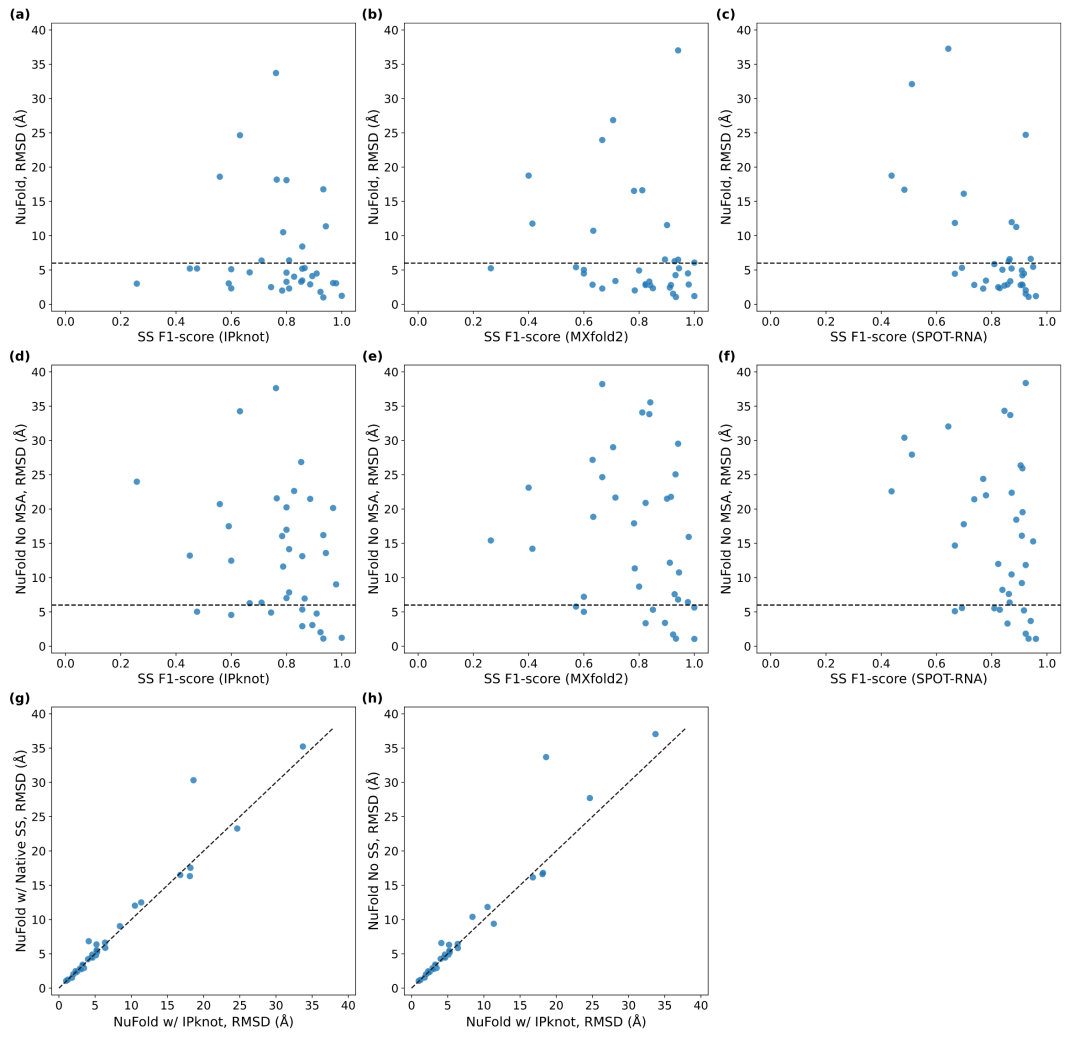
图 3
对于这个实验,作者使用了相同的网络模型,该模型使用IPknot预测进行了训练。作者没有针对每种方法的预测专门重新训练网络。图3a-c显示了测试目标的RMSD相对于二级结构预测的F1分数。在MSA输入的情况下,SPOT-RNA预测的二级结构F1分数与预测的三级结构的RMSD之间观察到中等程度的相关性,而其他两种方法仅观察到很弱的相关性。IPknot、MXfold2和SPOT-RNA的皮尔逊相关系数分别为-0.104、-0.174和-0.595。在图3d-f中,作者移除了MSA输入,试图看到二级结构预测准确性对建模性能的更清晰影响。然而,没有MSA,建模准确性大幅下降,只观察到弱相关性。IPknot、MXfold2和SPOT-RNA的皮尔逊相关系数分别为-0.245、-0.197和-0.322。
图3g、h比较了基准NuFold与具有两种极端二级结构信息的NuFold:从天然结构计算得到的完全准确信息(图3g)和没有二级结构信息(图3h)。结果表明,二级结构信息并没有使RMSD相对于基准NuFold有太大改善。在36个目标中,图3g中有24个是平局(即RMSD差异小于0.5Å),而图3h中有23个目标是平局。如附表2所示,与没有二级结构信息的Nufold相比,添加预测或真实的二级结构信息平均改善了RMSD。然而,在几乎所有情况下,这种改善在统计上并不显著,除了在没有MSA输入的情况下使用IPknot预测的预测。
编译|黄海涛
审稿|王梓旭
参考资料
Kagaya, Y., Zhang, Z., Ibtehaz, N., Wang, X., Nakamura, T., Punuru, P. D., & Kihara, D. (2025). NuFold: end-to-end approach for RNA tertiary structure prediction with flexible nucleobase center representation.
Nature communications, 16(1), 881.

